Documentos de Académico
Documentos de Profesional
Documentos de Cultura
Resumen Capitulo 3, 4 y 5
Cargado por
Cindy PazDerechos de autor
Formatos disponibles
Compartir este documento
Compartir o incrustar documentos
¿Le pareció útil este documento?
¿Este contenido es inapropiado?
Denunciar este documentoCopyright:
Formatos disponibles
Resumen Capitulo 3, 4 y 5
Cargado por
Cindy PazCopyright:
Formatos disponibles
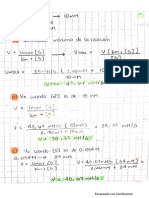
Resumen Capitulo 3
Punto 3.4: Estructura de las proteínas: estructura primaría.
Se definen normalmente cuatro niveles de estructura de las proteínas.:
1. La estructura primaria es una descripción de todos los enlaces covalentes (principalmente
enlaces peptídicos y puentes disulfuro) que unen los aminoácidos de una cadena
polipeptídica. El elemento más importante de la estructura primaria es la secuencia de los
residuos aminoácidos.
2. La estructura secundaria se refiere a disposiciones particularmente estables de los
aminoácidos que dan lugar a patrones estructurales repetitivos.
3. La estructura terciaria describe todos los aspectos del plegamiento tridimensional de un
polipéptido. Cuando una proteína posee dos o más subunidades polipeptídicas, su
disposición en el espacio se denomina estructura cuaternaria.
la estructura primaria de una proteína determina la forma en que se pliega en una estructura
tridimensional única y ésta, a su vez, determina la función de la proteína
La función de una proteína depende de su secuencia de aminoácidos.
Cada tipo de proteína también tiene una secuencia de aminoácidos única.
Una observación general sobre las proteínas y sobre sus variaciones en la secuencia de
aminoácidos proporciona una serie de pistas empíricas que ayudan a sustanciar la
importante relación entre secuencia de aminoácidos y función biológica.
Sabemos, por tanto, que si se altera la estructura primaria, la función de la proteína
también puede cambiar.
Se calcula que entre el 20 y el 30% de las proteínas humanas son polimórficas
Se ha determinado la secuencia de aminoácidos de millones de proteínas.
La identificación del residuo amino-terminal puede ser el primer paso de la secuenciación
de un polipéptido.
El procedimiento de la degradación de Edman permite obtener la secuencia completa de
un péptido.
Los polipéptidos cortos se secuencian utilizando procedimientos automáticos.
para marcar e identificar el residuo amino-terminal Sanger desarrolló el reactivo l-fluoro-
2,4-dini trobenceno (FDNB), otros reactivos utilizados son el cloruro de dabsilo y el cloruro
de dabsilo, que generan derivados más fácilmente detectables que los derivados
dinitrofenilados.
la etapa de hidrólisis destruye el polipéptido sin embargo, puede ayudar a determinar el
número de polipéptidos químicamente diferentes en una proteína, siempre que cada uno
tenga un residuo amino-terminal diferente.
la degradación de Edman, marca y elimina sólo el residuo amino-terminal de un péptido,
dejando el resto de enlaces peptídicos intactos. Se hace reaccionar el péptido con
fenilisotiocianato en condiciones alcalinas suaves, lo que convierte el aminoácido amino-
terminal en un aducto feniltiocarbamilo (PTC). El enlace peptídico contiguo al aducto de
PTC se rompe a continuación mediante reacción en ácido tricloroacético anhidro, que
conlleva la eliminación del aminoácido amino-terminal en forma de anilinotiazolinona. El
aminoácido modificado se extrae con disolventes orgánicos, se convierte en un derivado
más estable, feniltiohidantoína, mediante tratamiento con ácido en disolución acuosa y
finalmente se identifica.
La degradación de Edman se realiza en un instrumento, denominado secuenciador.
Las proteínas grandes deben secuenciarse a partir de fragmentos más pequeños.
La precisión global en la determinación de una secuencia de aminoácidos disminuye
generalmente a medida que aumenta la longitud del polipéptido.
En primer lugar se rompe la proteína en un conjunto de fragmentos específicos
mediante métodos químicos o enzimáticos. Si existen puentes disulfuro, es necesario
romperlos. A continuación se purifica cada fragmento y se secuencia mediante el
procedimiento de Edman. Finalmente se determina el orden en que los fragmentos
aparecen en la proteína original y se determina dónde están localizados, si es que los
hay, los enlaces disulfuro.
Rotura de los enlaces disulfuro: Los puentes disulfuro interfieren en el procedimiento
de secuenciación
Los puentes disulfuro también interfieren en la rotura enzimática o química del
polipéptido
Rotura de la cadena polipeptídica: Los enzimas denominados proteasas catalizan la
rotura hidrolítica de los enlaces peptídicos.
Rotura de puentes disulfuro en proteínas: La oxidación de un residuo de cistina con
ácido perfórmico produce dos residuos de ácido cisteico. La reducción con ditiotreitol
o Beta-mercaptoetanol que da lugar a residuos de Cys se ha de seguir con una
modificación adicional de los grupos —SH reactivos para impedir la reformación del
puente disulfuro. La acetilación con yodoacetato es útil en este último caso.
Secuenciación de los péptidos: Todos los fragmentos peptídicos resultantes de la
acción de la tripsina se secuencian separadamente por el procedimiento de Edman.
Ordenación de los fragmentos peptídicos: Ahora debe determinarse el orden de estos
“fragmentos trípticos” en la cadena polipeptídica original.
el bromuro de cianógeno sólo rompe aquellos enlaces peptídicos en los que el grupo
carbonilo es aportado por Met.
Localización de los puentes disulfuro: Si la estructura primaria incluye puentes
disulfuro, su Localización se determina en un paso adicional una vez completada la
secuenciación.
Las secuencias de aminorados se pueden deducir también mediante otros métodos.
Cada aminoácido está codificado por una secuencia específica de tres nucleótidos en el
DNA.
Para describir el complemento proteico entero codificado por el DNA de un organismo, los
investigadores han acuñado el término proteoma.
Las moléculas para analizar, a las que nos referimos como analitos.
como espectrometría de masas de ionización/desorción por láser asistida por matriz
(matrix-assisted láser desorption/ ionization) o MALDI MS, se ha utilizado con éxito para
medir la masa, de una gran variedad de macromoléculas.
El disolvente que rodea las macromoléculas se evapora rápidamente y los iones con
múltiples cargas resultantes se introducen de esta forma en la fase gas de manera no
destructiva. Esta técnica se denomina espectrometría de masas de ionización por
electrospray o ESI MS.
Es posible sintetizar químicamente péptidos y proteínas pequeñas.
Existen tres manera de obtener un péptido:
1. por purificación a partir del tejido, tarea a menudo dificultada por las concentraciones
extremadamente pequeñas de algunos péptidos.
2. mediante ingeniería genética.
3. por síntesis química directa.
La secuencia de aminoácidos proporciona información bioquímica importante.
Gran parte de la información funcional encapsulada en las secuencias proteicas se presenta en la
forma de secuencias consenso. Este término se aplica a secuencias de RNA, DNA o proteína.
la secuencia consenso es la que contiene la base o el aminoácido más frecuente en cada posición.
Las secuencias de proteínas permiten deducir ia historia de la vida en la Tierra.
Bioinformática.
Otro factor que complica la deducción de la historia evolutiva es la poco frecuente transferencia
de un gen o grupo de genes de un organismo a otro, según un proceso que se denomina
transferencia génica lateral.
Los miembros de las familias de proteínas son proteínas homólogas u homólogos.
Si dos proteínas de una misma familia (es decir, dos homólogos) se encuentran en la misma
especie, nos referimos a ellos como parálogos. Los homólogos de especies cüferentes se
denonünan ortólogo
También podría gustarte
- Introducción a la Biología: RESÚMENES UNIVERSITARIOSDe EverandIntroducción a la Biología: RESÚMENES UNIVERSITARIOSCalificación: 5 de 5 estrellas5/5 (1)
- Inducción A Los Procesos de Refinación Del SNRDocumento169 páginasInducción A Los Procesos de Refinación Del SNRErika MartínezAún no hay calificaciones
- Informe 7-Hidroxi-4metilcumarinaDocumento7 páginasInforme 7-Hidroxi-4metilcumarinaOscar Ramirez MartinezAún no hay calificaciones
- Acceso a Universidad para Mayores de 25 años. Biología 2013-2017.: Solucionario Pruebas 2013-2017De EverandAcceso a Universidad para Mayores de 25 años. Biología 2013-2017.: Solucionario Pruebas 2013-2017Aún no hay calificaciones
- Propiedades Químicas y Fisicoquímicas de Las ProteínasDocumento26 páginasPropiedades Químicas y Fisicoquímicas de Las Proteínas.:("*"BLacK BuLLeT"*"):.89% (45)
- Clase - 2 y 3 PDFDocumento79 páginasClase - 2 y 3 PDFMiguel Rodas Salazar100% (1)
- Acceso a Universidad para Mayores de 25 años. Biología.: Solucionario Pruebas 2006-2012De EverandAcceso a Universidad para Mayores de 25 años. Biología.: Solucionario Pruebas 2006-2012Aún no hay calificaciones
- Secuenciación de ProteínasDocumento6 páginasSecuenciación de ProteínasVALERIA ALZATE MERIZALDEAún no hay calificaciones
- Estructura de ProteinasDocumento9 páginasEstructura de ProteinasernestoruizxdAún no hay calificaciones
- Secuenciación e Identificación de Proteínas.Documento7 páginasSecuenciación e Identificación de Proteínas.Luis Gerardo GarciaAún no hay calificaciones
- Obtencion de EmulsinaDocumento10 páginasObtencion de Emulsina.:("*"BLacK BuLLeT"*"):.67% (3)
- Metabolismo de Proteinas y AminoacidosDocumento56 páginasMetabolismo de Proteinas y AminoacidosYaritza Romero100% (1)
- Reacciones de AminoácidosDocumento5 páginasReacciones de Aminoácidosnatalya oquendoAún no hay calificaciones
- ProteínaDocumento91 páginasProteínaRocky TinadoAún no hay calificaciones
- Guia Sobre Las Proteinas para Tercer Año - Iraima AqunoDocumento4 páginasGuia Sobre Las Proteinas para Tercer Año - Iraima AqunoRitaAún no hay calificaciones
- Bioquímica ResumenDocumento13 páginasBioquímica ResumenAlejandra LAún no hay calificaciones
- PROTEINASDocumento21 páginasPROTEINASCristian Daniel Angulo HerreraAún no hay calificaciones
- Síntesis de Proteínas en EucariotasDocumento5 páginasSíntesis de Proteínas en EucariotasMarco Antonio ContrerasAún no hay calificaciones
- Resumen Síntesis de Novo Purinas y PirimidinasDocumento14 páginasResumen Síntesis de Novo Purinas y Pirimidinasconi22Aún no hay calificaciones
- 15 Genes FuncionDocumento7 páginas15 Genes FuncionCarla Pabón FloresAún no hay calificaciones
- Transcripcion Del and (BQN)Documento20 páginasTranscripcion Del and (BQN)Erick A. ZepedaAún no hay calificaciones
- Proteinas 09Documento34 páginasProteinas 09Neila SanjuanAún no hay calificaciones
- Sintesis de ProteinaDocumento3 páginasSintesis de ProteinaJose AguilarAún no hay calificaciones
- Algoritmos en Bioinformática EstructuralDocumento98 páginasAlgoritmos en Bioinformática EstructuralLuis MacayaAún no hay calificaciones
- Unidad 5 Bio-2bDocumento10 páginasUnidad 5 Bio-2bRocío P AscAún no hay calificaciones
- Sintesis de ProteinasDocumento6 páginasSintesis de ProteinasZuriel Billyvan Hdz NolascoAún no hay calificaciones
- Tarea de Aminiácidos y ProteinassDocumento16 páginasTarea de Aminiácidos y ProteinassStyven Anthony Lopez TrujilloAún no hay calificaciones
- 4 Mod. PostranscripcionalesDocumento25 páginas4 Mod. PostranscripcionalesMelissa AlonsoAún no hay calificaciones
- 7 - Teoria Metabolismo de ProteinasDocumento35 páginas7 - Teoria Metabolismo de ProteinasCRISTHIAN MAYCOL SULLUCHUCO AGUIRREAún no hay calificaciones
- Bioquimica 2do ParcialDocumento18 páginasBioquimica 2do ParcialCamila Rios DiazAún no hay calificaciones
- Enzimas II Ped CienciasDocumento47 páginasEnzimas II Ped CienciasaadadAún no hay calificaciones
- 7 - Teoria Metabolismo de ProteinasDocumento36 páginas7 - Teoria Metabolismo de ProteinasYampier Quispe CallupeAún no hay calificaciones
- Generalidades de Las ProteinasDocumento33 páginasGeneralidades de Las ProteinasJuan RamirezAún no hay calificaciones
- Proteinas Bioquimica Equipo #3Documento24 páginasProteinas Bioquimica Equipo #3KARLA ROGELIA REYES GARCIAAún no hay calificaciones
- Proteinas DescripcionDocumento18 páginasProteinas DescripcionLEONARDOAún no hay calificaciones
- Tema 3 BioquimicaDocumento29 páginasTema 3 BioquimicaAlexandra María Marín BernalAún no hay calificaciones
- Ácidos NucleicosDocumento3 páginasÁcidos NucleicosNatalia CaballeroAún no hay calificaciones
- Ensayo Sobre La Síntesis de ProteínasDocumento8 páginasEnsayo Sobre La Síntesis de ProteínasAvenamar CB100% (1)
- PeroxisomasDocumento7 páginasPeroxisomasa381638Aún no hay calificaciones
- Funciones de Las ProteínasDocumento58 páginasFunciones de Las Proteínashampol gamesAún no hay calificaciones
- Biología Celular y Molecular Tarea 5 GrupalDocumento12 páginasBiología Celular y Molecular Tarea 5 GrupalANDREA YULIANA TRIANA CAMACHOAún no hay calificaciones
- Generalidades de Las ProteinasDocumento32 páginasGeneralidades de Las ProteinasJuan RamirezAún no hay calificaciones
- Sesion 2 - Problemas ResueltosDocumento16 páginasSesion 2 - Problemas ResueltosEli Conejo Dada100% (2)
- LipotropinaDocumento61 páginasLipotropinaJosé Enrique Jurado MenesesAún no hay calificaciones
- ProteínaDocumento13 páginasProteínayuta catboyAún no hay calificaciones
- Resumen de Bioquímica - Mecanismo de Acción de Los PéptidosDocumento16 páginasResumen de Bioquímica - Mecanismo de Acción de Los PéptidosIVAN MONTEZUMAAún no hay calificaciones
- T-37 Traducción Sintesis de ProteinasDocumento24 páginasT-37 Traducción Sintesis de ProteinasAnonymous iIaMAfVAún no hay calificaciones
- Sintesis de Acidos Nucleicos y ProteinasDocumento49 páginasSintesis de Acidos Nucleicos y ProteinasOmar FuentesAún no hay calificaciones
- Sintesis de ProteínasDocumento7 páginasSintesis de ProteínasKevin RojasAún no hay calificaciones
- Estructura y Funciones de Proteínas y EnzimasDocumento43 páginasEstructura y Funciones de Proteínas y EnzimasBet-oso PaezAún no hay calificaciones
- LipotropinaDocumento58 páginasLipotropinaJosé Enrique Jurado Meneses50% (2)
- Metabolismo BacterianoDocumento8 páginasMetabolismo BacterianoZue Ellen AlvarezAún no hay calificaciones
- Metabolismo de Las ProteínasDocumento5 páginasMetabolismo de Las ProteínasSarai BocelAún no hay calificaciones
- Clasificación de Las ProteinasDocumento13 páginasClasificación de Las ProteinasJoosé Angell M. RamirezAún no hay calificaciones
- Sintesis de ProteinasDocumento4 páginasSintesis de ProteinasJuniorRomeroAún no hay calificaciones
- Resumen Certamen III - BioquímicaDocumento17 páginasResumen Certamen III - BioquímicaFrancisco JavierAún no hay calificaciones
- Descifrar El Código Genético de Los Seres VivosDocumento9 páginasDescifrar El Código Genético de Los Seres VivosBarbara Ravelo Molina100% (1)
- Artículo 3Documento2 páginasArtículo 3JHADIRA ANGGIE RISALVE CUEVAAún no hay calificaciones
- Metabolismo de Las Proteína1Documento5 páginasMetabolismo de Las Proteína1Sarai BocelAún no hay calificaciones
- Biomoléculas 1Documento14 páginasBiomoléculas 1Esteban HiguerasAún no hay calificaciones
- Proteína - Wikipedia, La Enciclopedia Libre PDFDocumento79 páginasProteína - Wikipedia, La Enciclopedia Libre PDFCristhian Stiven Mozos PerdomoAún no hay calificaciones
- Protein AsDocumento4 páginasProtein AsAngel EsAvalAún no hay calificaciones
- Ejercicios Resueltos BioquímicaDocumento7 páginasEjercicios Resueltos BioquímicaCindy PazAún no hay calificaciones
- Apuntes Laboratorio BioquímicaDocumento20 páginasApuntes Laboratorio BioquímicaCindy PazAún no hay calificaciones
- Resumen Capitulo 3, 4 y 5Documento4 páginasResumen Capitulo 3, 4 y 5Cindy PazAún no hay calificaciones
- Apuntes BioquíDocumento36 páginasApuntes BioquíCindy PazAún no hay calificaciones
- Propiedades Generales de La ProteínasDocumento7 páginasPropiedades Generales de La ProteínasJulian VillanuevaAún no hay calificaciones
- Implementacion de Los Biocombustibles en La Industria AeronauticaDocumento21 páginasImplementacion de Los Biocombustibles en La Industria Aeronauticamijial canedoAún no hay calificaciones
- PRACTICA N°2 Preparación de Medios de CultivoDocumento9 páginasPRACTICA N°2 Preparación de Medios de CultivoOmar PonceAún no hay calificaciones
- Comparativo Del Arn y AdnDocumento2 páginasComparativo Del Arn y AdnJoa LizAún no hay calificaciones
- CeraVe Suero de Retinol para Marcas de Acné y Textura de La Piel Refinación de Poros, Rejuvenecimiento, Suero FaciDocumento1 páginaCeraVe Suero de Retinol para Marcas de Acné y Textura de La Piel Refinación de Poros, Rejuvenecimiento, Suero FaciAlejandra GálvezAún no hay calificaciones
- 1-El Petroleo y Los Hidrocarburos PDFDocumento22 páginas1-El Petroleo y Los Hidrocarburos PDFpaula quirogaAún no hay calificaciones
- Esquema de Proyecto-1Documento16 páginasEsquema de Proyecto-1Lis VeraAún no hay calificaciones
- 7 Fotosintesis - Digestion para EnviarDocumento17 páginas7 Fotosintesis - Digestion para Enviarhilary valdiviaAún no hay calificaciones
- La Arginina Regula El Recambio de Proteínas en Las Células Epiteliales Mamarias Porcinas para Mejorar La Síntesis de Proteínas de La LecheDocumento8 páginasLa Arginina Regula El Recambio de Proteínas en Las Células Epiteliales Mamarias Porcinas para Mejorar La Síntesis de Proteínas de La LecheMARIAAAAAAAAAAAAún no hay calificaciones
- Polimeros A Partir de CerealesDocumento3 páginasPolimeros A Partir de CerealesJuan Bedoya0% (1)
- Green Care 1-2021Documento4 páginasGreen Care 1-2021Lizeth MatuteAún no hay calificaciones
- Caracterización de Residuos de Acetato de Celulosa de La Industria DentalDocumento2 páginasCaracterización de Residuos de Acetato de Celulosa de La Industria DentalEduardo Elias CastellanosAún no hay calificaciones
- Tratamiento Biologico de Aguas ResidualesDocumento7 páginasTratamiento Biologico de Aguas ResidualesRoxana VelascoAún no hay calificaciones
- Composicion de La CanelaDocumento8 páginasComposicion de La CanelaDavid Zamora0% (1)
- Transfromación Termoquímica de Biomasa PDFDocumento67 páginasTransfromación Termoquímica de Biomasa PDFMARIA ALEJANDRA VILLAMIZAR PEÑAAún no hay calificaciones
- Lab #8 - ÁCIDOS CARBOXÍLICOSDocumento15 páginasLab #8 - ÁCIDOS CARBOXÍLICOSRaul Vidal0% (1)
- Aldehidos Y Cetonas: Informe #10Documento6 páginasAldehidos Y Cetonas: Informe #10Yairkhat LovegoodAún no hay calificaciones
- Portafolio Completo BajaDocumento20 páginasPortafolio Completo BajaJohan Esteban AlfonsoAún no hay calificaciones
- Práctica 6 PreinformeDocumento2 páginasPráctica 6 Preinformecamilatimaran43Aún no hay calificaciones
- Aguas MétodosDocumento8 páginasAguas MétodosKiraly QuinteroAún no hay calificaciones
- Diapositivas LipasasDocumento11 páginasDiapositivas LipasasMily Benavides CiezaAún no hay calificaciones
- Guía Química 11 - Periodo II - 2023Documento11 páginasGuía Química 11 - Periodo II - 2023SANDRA MILENA RIOS MALDONADOAún no hay calificaciones
- Lab. 3 Esteriosomeria Cis - TransDocumento1 páginaLab. 3 Esteriosomeria Cis - TransMARIA SUIRAAún no hay calificaciones
- BIODELTA. Presentacion Comercial Maiz V22021Documento32 páginasBIODELTA. Presentacion Comercial Maiz V22021Martin Alejandro Correa TrujilloAún no hay calificaciones
- 1508 Material Docente Compendio de Bioquimica 2a EdiciónDocumento83 páginas1508 Material Docente Compendio de Bioquimica 2a EdiciónJonathan GCAún no hay calificaciones
- Semana 12. Reacciones QuímicasDocumento69 páginasSemana 12. Reacciones QuímicasPablo VasconezAún no hay calificaciones
- Frotis Bacterianos y TincionesDocumento42 páginasFrotis Bacterianos y TincionesBritziAún no hay calificaciones