Documentos de Académico
Documentos de Profesional
Documentos de Cultura
Biosintesis en Adn y Reparación
Cargado por
Paul CapellanTítulo original
Derechos de autor
Formatos disponibles
Compartir este documento
Compartir o incrustar documentos
¿Le pareció útil este documento?
¿Este contenido es inapropiado?
Denunciar este documentoCopyright:
Formatos disponibles
Biosintesis en Adn y Reparación
Cargado por
Paul CapellanCopyright:
Formatos disponibles
BIOSINTESIS EN ADN Y REPARACIÓN
DOGMA CENTRAL DE LA BIOLOGÍA
Establece que la información fluye desde el ADN al ARN y de este a las proteínas.
Fases:
1. Replicación (Biosíntesis de ADN)
2. Transcripción (Biosíntesis de ARN)
3. Traducción (Biosíntesis de Proteínas)
4. Transcripción inversa (Biosíntesis de ADN con molde de ARN)
* 2 + 3 = Expresión de la Información Genética *
REPLICACIÓN DE ADN EN CÉLULAS PROCARIOTAS
La replicación es un proceso que permite conservar inalterada la información genética y
transmitirla integra a la siguiente generación. No debe afectar a la integridad de la
información genética, por lo que se realiza asegurando la fidelidad de la copia. Se realiza a
partir de un molde preexistente. Ocurre solo una vez en la vida de la célula, justo entes de
la división celular. Sus características son:
• SEMICONSERVADOR: Cada una de las hebras del ADN parental se asocia con
una de las hebras de nueva síntesis, dando dos moléculas de ADN idénticas
a la original cada una de ellas está compuesta de una hebra del ADN original
y de una complementaria nueva (cada cadena de la doble hélice sirve para la
síntesis de una nueva complementaria).
• Tiene un ORIGEN DE REPLICACIÓN (se inicia en un punto): las moléculas de
ADN inician su ciclo de replicación en una secuencia de bases única llamada
origen de replicación. Es único de organismos procariotas y casi todos los
virus. Por ejemplo, en el E. Coli hay una secuencia de 245pb (ori C) que
contiene varios sitios de unión para la proteína dnaA que inicia la replicación
y un agrupamiento en tándem de una secuencia rica en A y T en el que
comienza a abrirse la horquilla por separación de las dos cadenas de la
doble hélice.
• Es BIDIRECCIONAL y tiene lugar en horquillas de replicación. También podría
ser unidireccional (en ADN mitocondrial y algunos virus) en la que la
horquilla parte de un origen y avanzaría a lo largo del ADN en un solo
sentido. El ser bidireccional implica que de un mismo punto de origina se
forman dos horquillas que avanzan en sentidos opuestos.
• La síntesis es siempre en EL SENTIFO 5´a 3´. Ya que las enzimas sólo pueden
catalizar la adición de nuevos nucleótidos a un grupo hidroxilo libre situado
en el extremo 3´de la cadena.
• Hay una HEBRA GUIA que se replica como cadena continua y una HEBRA
REZAGADA que se sintetiza mediante fragmentos cortos de unos 1000 –
2000 nucleótidos que después se unen entre sí. (Fragmentos de Okazaki)
• Este proceso esta catalizado por las ADN polimerasas. Todas las enzimas
necesitan disponer de:
o Cadena molde de ADN.
o Extremo 3´hidroxilo libre (3´-OH) que actúa como cebador.
o Mg2+ y los desoxirribonucleótidos trifosfatos (dATP, dGTP, dTTP, dCTP).
o Dirección 5´- 3´.
o Tener actividad exonucleasa para corregir algún posible error en la
incorporación de nucleótidos. Esto lo hacen en sentido 3´- 5´.
ADN POLIMERASA I: Cataliza la polimerización de dNTP en ADN. Tiene que tener un
molde y un cebador (pequeños oligonucleótidos que es complementario al molde y que
tiene un grupo OH en la posición 3´al cual se le pueden añadir nuevos nucleótidos). No
pueden empezar la síntesis de una hebra a partir de 0, a diferencia de las ARN polimerasa.
Por esa razón los cebadores son siempre moléculas de ARN cortos.
REACCIÓN GLOBAL: (DNA)n + dNTP ↔ (DNA)n+1 + PPi
REACCIÓN DE POLIMERIZACIÓN: Ataque nucleofílico del grupo OH 3´de la hebra
creciente sobre el fosfato en posición 5´del dNTP que se va a incorporar. Esta reacción
libera pirofosfato. Esta reacción esta favorecida por los 19 kJ/mol liberados de la hidrolisis
del pirofosfato.
PPi + H2O → 2Pi + 2H+
Otras propiedades del ADN polimerasa I:
• Actividad exonucleasa: Función correctora y reparadora que les confiere la
capacidad de degradar el ADN partiendo del extremo. Corriendo los errores en
la copia del ADN después de un ciclo catalítico en el sentido 3´- 5´. Esta
propiedad también hace que puedan eliminar cebadores en el sentido 5´- 3´.
Asegura la fidelidad de la copia. Esta actividad está en su entorno activo. Hay
mecanismos adicionales de reparación que reducen las tasas de error.
En la bacteria E. Coli hay varias polimerasas:
• POLIMERASA I: Abundante. “Limpia” durante la replicación, reparación y
recombinación.
• POLIMERASA II: Responsable de la reparación del ADN.
• POLIMERASA III: Responsable de la
replicación del ADN.
Las subunidades δ´, δ, τ, Ƴ forman
parte del complejo de carga de la
abrazadera.
La abrazadera necesaria para atrapar
la hebra de ADN, está formado por un
dímero de la subunidad β.
Hay una serie de requerimiento para el proceso de requerimiento:
• MOLDE de ADN preexistente (cadena o fragmento de cadena simple).
• CEBADOR: Pequeño fragmento de ARN (alrededor de 5 nucleótidos) con el
grupo 3´-OH libre, unido al ADN molde (sintetizado por la primasa, que forma un
cebador de ARN y ADN).
• PRECURSORES activados (dNTP)
• ADN polimerasa: Catalizan la formación de enlaces fosfodiéster dirigida por un
molde de ADN (actividad polimerasa 5´→ 3´) y la corrección de errores
(exonucleasa 3´→ 5´). Necesitan Mg2+.
o Polimerasa I: Elimina el cebador por exonucleasas (5´- 3´) y rellena el
hueco.
o Polimerasa III: Sintetiza una nueva cadena de ADN.
• Intervienen otras enzimas, como:
o HELICASA: Es una proteína motora que separa las 2 hebras del ADN
dúplex. Requiere ADN.
o PRIMASA: ARN polimerasa que se sintetiza al cebador. Forma parte del
complejo primosoma (contiene helicasas, primasas y otros polipéptidos
que catalizan el proceso de la replicación).
o DNA GIRASA (topoisomerasas II): Genera superenrollamiento negativo,
es decir, lo desenreda para facilitar la replicación. Requiere ATP.
o DNA LIGASAS: une fragmentos recién sintetizados.
o SSBP (Proteínas estabilizadoras): Proteínas de unión a cadena sencilla,
estabilización de la apertura del ADN de cadena sencilla. Evitan el
autoapareamiento entre bases complementarias, protegiéndola de la
acción de las nucleasas y de asociación intracatenarias. Sus análogas en
eucariotas son RPA.
PROCESO DE REPLICACIÓN
INICIACIÓN
1. Se inicia en lugares específicos. La unión de la dnaA inicia el proceso. Se forma un ojo y
una horquilla de replicación.
2. Las helicasas, dnaA separan las dos hebras. Requiere ayuda de AnaC que es una
proteína que actúa como cargador de la helicasa.
3. Las cadenas simples se estabilizan por la SSBP.
4. La primasa sintetiza cebadores de ARN sobre las hebras simples de la horquilla.
ELONGACIÓN
5. La DNA polimerasa III sintetiza un nuevo ADN a
partir el cebador siempre en dirección 5´→ 3´:
• La hebra guía se sintetiza de manera
continua.
• La hebra retardada forma un bucle
donde la síntesis de ADN ocurre de
manera discontinua en fragmentos de
Okazaki.
6. La DNA Polimerasa I elimina los ARN cebadores y reemplaza sintetizando ADN en su
lugar.
• Tienen actividad exonucleasa 5´→ 3´ y polimerasa 5´→ 3´.
7. Los fragmentos de la hebra retardada son ligandos por la DNA ligasa.
8. Durante todo el proceso la helicasa va desenrollado el ADN y generando tensión
topológica que es contrarrestada por la DNA girasa.
TERMINACIÓN
9. La replicación finaliza en una región
cromosómica rica en secuencias Ter (20pb) con
la participación de la proteína Tus. Después, la
DNA topoisomerasa IV separa los dos
cromosomas encadenados.
REPLICACIÓN DE ADN EN CELULAS EUCARIOTAS
EUCARIOTAS VS PROCARIOTAS
Homologías:
• Semiconservativa
• Bidireccional
• Semidiscontinua
• Mecanismo general
Diferencias:
• Regulada con el ciclo celular
• Mas compleja (Histonas)
• Velocidad más lenta
• Múltiples orígenes de replicación
• DNA polimerasas eucariotas
• Telómeros = transcripción inversa
MÚLTIPLES ORIGENES DE REPLICACIÓN
En eucariotas, las secuencias de replicación se llaman secuencias de replicación
autónoma (SRA). En levaduras se han identificado unas 400 SRA, lo que quiere decir que
cada cromosoma (16 en levadura) contiene mas de una. También se han encontrado
proteínas que se unen específicamente a estas SRA.
DNA POLIMERASAS EUCARIÓTICAS
En eucariotas también hay varios tipos de DNA-polimerasas. La replicación de los
cromosomas nucleares la realizan la DNA.-polimerasa “α” y la DNA-polimerasa “δ”.
La primera presenta actividad primasa = síntesis de cebadores. La DNA-polimerasa “δ”
presenta actividad exonucleasa 3´→ 5´ (función correctora) y se piensa que es la principal
responsable de la copia.
Una tercera polimerasa, la DNA-polimerasa “ε” parece cumplir la función de relleno y
reparación del DNA equivalente a la DNA Pol I bacteriana.
PROBLEMA DE LOS EXTREMOS DEL CROMOSOMA
TELOMERASA (RIBONICLEOPROTEINAS)
La telomerasa usa su propio ARN como molde
para elongar el extremo 3´
Las DNA Polimerasas completan la tarea
ERRORES EN LA REPLICACIÓN: MUTACIONES
Lesiones espontaneas en el ADN modifican de modo transitorio la información genética
La replicación convierte las lesiones en el ADN en cambios permanentes = mutantes
La tautomería de bases también causa mutaciones
La radiación UV también provoca lesiones en el ADN que dañan la información genética
MECANISMOS DE REPARACIÓN DEL ADN
• Reparación de bases discordantes (errores de replicación)
o Mejora de fidelidad de la replicación un factor de 10 2-103: Se basa en la
discriminación de hebra y requiere la participación de una docena de
proteínas.
• Reparación por escisión de base (BER)
o Las bases dañadas se retiran y se reemplazan
• Reparación por escisión de nucleótidos (NER)
o Se elimina y se reemplaza un fragmento en torno a la lesión: La escinucleasa
uvrABC escinde 12 nucleótidos en la zona dañada. Regeneración a cargo de la
DNA Polimerasa y la DNA ligasa.
• Reparación directa
o La región dañada se corrige “in situ”: la DNA fotoliasa utiliza energía de la luz
para eliminar los dímeros de pirimidina formados por radiación UV.
Reparación de bases discordantes (errores de la replicación)
Reparación por escisión de base
Este proceso comienza con la escisión de la base
modificada por la hidrolisis del enlace N-Glucosídico.
Implica la acción de una DNA-N-Glucosidasa para
eliminar la base modificada y una endonucleasa AP par
eliminar la desoxi-ribosa sin base.
DNA Polimerasa I y DNA ligasa rellena el hueco y
restauran la continuidad de la hebra.
Reparación por escisión de nucleótidos (NER)
La NER es un proceso general que se usa para corregir distintos tipos de lesiones entre
los que se incluye la reparación de los dímeros timina inducidos por radiación ultravioleta.
En el proceso NER participan las proteínas uvrA, avrB, avrC, uvrD, ADN Polimerasa I y
ADN Ligasa.
El proceso comienza cuando un trímero uvrA2B
rastrea el ADN dúplex en busca de una lesión que
distorsiona la estructura helicoidal. Cuando encuentra
una zona dañada (como un dinero de timina), el
complejo se para y la proteína uvrB queda unida a la
lesión mientras que uvrA abandona el complejo.
Entonces, la escinucleasa uvrC se une a uvrB en la
zona lesionada y rompe un par de enlaces fosfodiéster
a ambos lados de la lesión dejando varios nucleótidos
de separación con el eslabón dañado.
En esta situación la helicasa uvrD se une en 5´al
fragmento dañado y desplazándose hacia 3´separa el
fragmento lesionado del resto de la molécula. Así se
genera una brecha, que es rellenada por el ADN
Polimerasa I, tomando las instrucciones originales de la
hebra parental, y la ADN Ligasa empalma los dos
fragmentos mediante un puente fosfodiéster que
restablece la continuidad de la hebra reparada
Reparación directa
Las radiaciones ionizantes y ultravioleta causan lesiones en el ADN provocando la
formación de dímeros de timina que distorsionan la doble hélice. La DNA fotoliasa, una
enzima reparadora activada por la luz, repara esta lesión eliminando los dímeros de timina.
La DNA fotoliasa se une al dímero de timina y tras captar un fotón transfiere un electrón
al dímero de timina, generando un radial libre que libera la ruptura de los enlaces entre las
dos timinas adyacentes. Después de que el dímero ha sido eliminado, la enzima recupera el
electrón que había cedido.
Distintos agentes químicos
provocan la alquilación o metilación
del átomo de oxigeno que ocupa la
posición 6 del anillo de guanina. Estos
cambios covalentes pueden dar lugar a
mutaciones. La enzima metilguanina-
DNA metil transferasa se une
directamente a las guaninas alquiladas
y elimina estos sustituyentes,
restituyendo la estructura nativa de la
base, evitando la aparición de
mutaciones.
También podría gustarte
- Tema 2 ReplicacionDocumento26 páginasTema 2 ReplicacionLucero Gerarda Gonzalez Pizarro100% (1)
- Introducción a la Biología: RESÚMENES UNIVERSITARIOSDe EverandIntroducción a la Biología: RESÚMENES UNIVERSITARIOSCalificación: 5 de 5 estrellas5/5 (1)
- Dogma Central de La Biologia MolecularDocumento61 páginasDogma Central de La Biologia MolecularNydia Córdova Pérez100% (2)
- Genética BacterianaDocumento111 páginasGenética BacterianaEylin EspinozaAún no hay calificaciones
- Replicación de ProcariotasDocumento36 páginasReplicación de ProcariotasCesar Rajapakse RiosAún no hay calificaciones
- Acceso a Universidad para Mayores de 25 años. Biología.: Solucionario Pruebas 2006-2012De EverandAcceso a Universidad para Mayores de 25 años. Biología.: Solucionario Pruebas 2006-2012Aún no hay calificaciones
- Tema 2 ReplicacionDocumento25 páginasTema 2 ReplicacionArsenio Flores VasquezAún no hay calificaciones
- Evaluacion de Sintesis de ProteinasDocumento2 páginasEvaluacion de Sintesis de ProteinasFamilia Medina HerreraAún no hay calificaciones
- Replicación Grupo 10Documento25 páginasReplicación Grupo 10Elsa MoranAún no hay calificaciones
- TEMA5Documento32 páginasTEMA5Claribel ValdezAún no hay calificaciones
- Etapas de La ReplicaciónDocumento34 páginasEtapas de La ReplicaciónChris Guerrero100% (1)
- Unidad 4. Replicaci-N. BM 2024ADocumento38 páginasUnidad 4. Replicaci-N. BM 2024Adecker1813Aún no hay calificaciones
- Acidos NucleicosDocumento74 páginasAcidos NucleicosMayerOrozcoAún no hay calificaciones
- BIOQUIMICADocumento93 páginasBIOQUIMICASanti VedoyaAún no hay calificaciones
- ReplicacionDocumento28 páginasReplicacionAurelia Gonzales CalleAún no hay calificaciones
- Metabolismo Del AdnDocumento59 páginasMetabolismo Del AdnM Orihuela100% (1)
- Adn Inf GeneticaDocumento7 páginasAdn Inf Geneticaaitana pucheAún no hay calificaciones
- Foro Molecular Replicación MolecularDocumento7 páginasForo Molecular Replicación MolecularLeonardo Quintero MárquezAún no hay calificaciones
- Módulo 12. Ácidos Nucleicos. Replicación y Transcripción Del ADNDocumento68 páginasMódulo 12. Ácidos Nucleicos. Replicación y Transcripción Del ADNLuis Enrique Vera CamposAún no hay calificaciones
- Replicación Del ADNDocumento13 páginasReplicación Del ADNRomina OrejuelaAún no hay calificaciones
- REPLICACION PresentacionDocumento32 páginasREPLICACION PresentacionMaría Nathalie Hidálgo González BAún no hay calificaciones
- Replicacion de La Informacion GeneticaDocumento77 páginasReplicacion de La Informacion Geneticaangeldb1992Aún no hay calificaciones
- Replicaciondeladn 180416143831Documento22 páginasReplicaciondeladn 180416143831EVELIN NAOMI PEREZ MATEOAún no hay calificaciones
- 23UIDocumento9 páginas23UIWiam Kenzaoui EssalamaAún no hay calificaciones
- 3 Replicación de ADN. Grupo2Documento27 páginas3 Replicación de ADN. Grupo2July Aguila100% (1)
- Replicación Del AdnDocumento28 páginasReplicación Del AdnisaacAún no hay calificaciones
- Adn OdontologiaDocumento46 páginasAdn OdontologiaLourdesAún no hay calificaciones
- Tema 15 BioDocumento6 páginasTema 15 BioLauraAún no hay calificaciones
- ReplicaciónDocumento29 páginasReplicaciónPaula OrtegaAún no hay calificaciones
- Tema 5 - Biología MolecularDocumento8 páginasTema 5 - Biología MolecularMari Carmen ReinéAún no hay calificaciones
- TP3 BiotecnolgíaDocumento7 páginasTP3 BiotecnolgíaMartinaAún no hay calificaciones
- La Replicación Del ADNDocumento2 páginasLa Replicación Del ADNDavid Nieves CamañoAún no hay calificaciones
- QUIM Tema 10. ReplicaciónDocumento5 páginasQUIM Tema 10. ReplicaciónCarmen LostalAún no hay calificaciones
- 09 - Sintesis Del DNA. Reparacià NDocumento30 páginas09 - Sintesis Del DNA. Reparacià NAbraham HuamanAún no hay calificaciones
- Replicación: Síntesis de ADN: Biología Molecular y GenéticaDocumento34 páginasReplicación: Síntesis de ADN: Biología Molecular y GenéticaJosé CisternasAún no hay calificaciones
- 13-02-5 BiDocumento96 páginas13-02-5 BicarolinaAún no hay calificaciones
- Taller de Replicación Trasncripcion TraducciónDocumento5 páginasTaller de Replicación Trasncripcion TraducciónLaura Isabel Tabares BuitragoAún no hay calificaciones
- Biología 2ºbach Apuntes Tema 10 - Genética MolecularDocumento7 páginasBiología 2ºbach Apuntes Tema 10 - Genética MolecularDainAún no hay calificaciones
- ReplicaciónDocumento34 páginasReplicaciónPAZ MARTINA ALLENDESAún no hay calificaciones
- Bmi TDocumento176 páginasBmi TJuan Arcos HuasasquicheAún no hay calificaciones
- Replica AdnDocumento8 páginasReplica Adnking of endAún no hay calificaciones
- Replicacion Transcri 361893 Downloadable 3511455Documento3 páginasReplicacion Transcri 361893 Downloadable 3511455Ana BaldeonAún no hay calificaciones
- Genetica Molecular y Microbiana 2016 IDocumento169 páginasGenetica Molecular y Microbiana 2016 IMavix Arias AguilaAún no hay calificaciones
- 12 - Genética Molecular IDocumento17 páginas12 - Genética Molecular IMiguel SanchezAún no hay calificaciones
- Dogma Central de La Biología MolecularDocumento3 páginasDogma Central de La Biología Molecularjosecerc27Aún no hay calificaciones
- Replicación Del ADN - DeR 1Documento33 páginasReplicación Del ADN - DeR 1thiagoscathcartAún no hay calificaciones
- Clase 12Documento40 páginasClase 12alfredoAún no hay calificaciones
- T11 Flujo de La Información GenéticaDocumento11 páginasT11 Flujo de La Información GenéticaMaria BurgosAún no hay calificaciones
- Replicación Del DnaDocumento3 páginasReplicación Del DnaLuz MarianaAún no hay calificaciones
- DNA RecombinanteDocumento70 páginasDNA RecombinanteRobert CaballeroAún no hay calificaciones
- Replicación Del DNADocumento60 páginasReplicación Del DNACarolinaPonceHenriquezAún no hay calificaciones
- REPLICACIONDocumento21 páginasREPLICACIONElias CortezAún no hay calificaciones
- RTT Del ADNDocumento9 páginasRTT Del ADNAnayantzi AllendeAún no hay calificaciones
- NúcleoDocumento6 páginasNúcleoMaría Paula MendezAún no hay calificaciones
- El Adn Mutaciones E Ingeniería GenéticaDocumento49 páginasEl Adn Mutaciones E Ingeniería GenéticaHjb HujjAún no hay calificaciones
- Clase 3 Replicación Del DNA en Organismos ProcariotasDocumento5 páginasClase 3 Replicación Del DNA en Organismos Procariotasfrankk ;;Aún no hay calificaciones
- DNA, RNA, Replicación, Transcripción y TraducciónDocumento141 páginasDNA, RNA, Replicación, Transcripción y TraducciónKarenziita MdzAún no hay calificaciones
- ReplicaciónDocumento24 páginasReplicaciónCristhian OlivarAún no hay calificaciones
- Replicacion Del AdnDocumento14 páginasReplicacion Del AdnCristian ViloriaAún no hay calificaciones
- Guía BiologíaDocumento30 páginasGuía BiologíaKatyAún no hay calificaciones
- UntitledDocumento17 páginasUntitledNatalia LawwlietAún no hay calificaciones
- Tracducción y Biosintesis de ProteinasDocumento6 páginasTracducción y Biosintesis de ProteinasPaul CapellanAún no hay calificaciones
- Transcripción y Biosintesis Del ArnDocumento12 páginasTranscripción y Biosintesis Del ArnPaul CapellanAún no hay calificaciones
- Introduccion Al MetabolismoDocumento19 páginasIntroduccion Al MetabolismoPaul CapellanAún no hay calificaciones
- Información GenéticaDocumento5 páginasInformación GenéticaPaul CapellanAún no hay calificaciones
- Adn ArmDocumento17 páginasAdn Armyojaida niñoAún no hay calificaciones
- Replicacion Alberts - En.esDocumento18 páginasReplicacion Alberts - En.esPaula NastarAún no hay calificaciones
- Biología MolecularDocumento14 páginasBiología MolecularMaria Gala Saavedra UribeAún no hay calificaciones
- Ejer Cici Os TraducciónDocumento2 páginasEjer Cici Os TraduccióngilbertoperezzaletaAún no hay calificaciones
- Ácidos Nucleicos Medicina 2017 150Documento149 páginasÁcidos Nucleicos Medicina 2017 150Alix pierina Chavez minoAún no hay calificaciones
- Ejercicio SIntesis de ProteinasDocumento2 páginasEjercicio SIntesis de ProteinasSandra Pilar Mancilla peña100% (3)
- 5 ExamenDocumento11 páginas5 ExamenNalleli Lava AlfonsoAún no hay calificaciones
- Plásmidos RecombinantesDocumento3 páginasPlásmidos RecombinantesLaura PeñaAún no hay calificaciones
- Sintesis Del AdnDocumento7 páginasSintesis Del AdnSantiago RochaAún no hay calificaciones
- Biologia Semana 14Documento4 páginasBiologia Semana 14carlos MEdinaAún no hay calificaciones
- Genética UCSUR - T2 - Bases Cromosomicas de La HerenciaDocumento63 páginasGenética UCSUR - T2 - Bases Cromosomicas de La HerenciaDiego Cruzado oroscoAún no hay calificaciones
- Proceso de Maduracion de Los Diferentes ArnDocumento4 páginasProceso de Maduracion de Los Diferentes ArnJosselynSánchezYRichardBarrezuetaAún no hay calificaciones
- Genetica Medica - 2022Documento164 páginasGenetica Medica - 2022Rut Ester González VásquezAún no hay calificaciones
- CARIOTIPODocumento3 páginasCARIOTIPOcad3792Aún no hay calificaciones
- Interpretacion de Tecnicas Moleculares.Documento19 páginasInterpretacion de Tecnicas Moleculares.CATALINA VASQUEZ LOPEZAún no hay calificaciones
- Mapa Grupo 1Documento2 páginasMapa Grupo 1domenica guamanAún no hay calificaciones
- DOGMA CENTRAL DE LA BIOLOGÍA MOLECULAR XimenaDocumento2 páginasDOGMA CENTRAL DE LA BIOLOGÍA MOLECULAR XimenaXImena FernandezAún no hay calificaciones
- Keratina de OvejaDocumento17 páginasKeratina de OvejaAaron SosaAún no hay calificaciones
- Taller Expresión Génica 1Documento3 páginasTaller Expresión Génica 1Edgar NavarroAún no hay calificaciones
- Tecnicas MolecularesDocumento7 páginasTecnicas MolecularesElvis Aviles MendozaAún no hay calificaciones
- Replicación Del DNADocumento25 páginasReplicación Del DNASayda Alisson Huacre TuctoAún no hay calificaciones
- Examen TEMA 7 (Posibles Preguntas)Documento5 páginasExamen TEMA 7 (Posibles Preguntas)Lucia MoralesAún no hay calificaciones
- Secuenciación de Sanger - VegaDocumento2 páginasSecuenciación de Sanger - VegaKarla VegaAún no hay calificaciones
- Una Duplicación Genética Mediada Por Retrotransposon Subyace A La Variación Morfológica de La Fruta de TomateDocumento8 páginasUna Duplicación Genética Mediada Por Retrotransposon Subyace A La Variación Morfológica de La Fruta de TomateDaniela PadillaAún no hay calificaciones
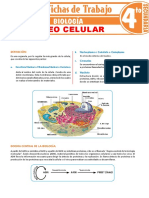
- Nucleo Celular para Cuarto Grado de SecundariaDocumento4 páginasNucleo Celular para Cuarto Grado de SecundariaGloria LorenzoAún no hay calificaciones
- Monografía, Primera ParteDocumento6 páginasMonografía, Primera ParteEmilio CordovaAún no hay calificaciones
- Generalidades y Transcripción Eucariotas y ProcariotasDocumento4 páginasGeneralidades y Transcripción Eucariotas y ProcariotasPaulitagbAún no hay calificaciones
- Proyecto de Biologia 2do de Bachillerato Parcial 2Documento18 páginasProyecto de Biologia 2do de Bachillerato Parcial 2Yury RamirezAún no hay calificaciones
- Gen Sry: Alteraciones Genéticas de La Determinación SexualDocumento1 páginaGen Sry: Alteraciones Genéticas de La Determinación Sexualcamila gomezAún no hay calificaciones