Documentos de Académico
Documentos de Profesional
Documentos de Cultura
DNA Mismatch Repair (MMR) Cristian Cuaspud
Cargado por
Criistiian Dariioo0 calificaciones0% encontró este documento útil (0 votos)
22 vistas5 páginasfisca
Derechos de autor
© © All Rights Reserved
Formatos disponibles
PDF, TXT o lea en línea desde Scribd
Compartir este documento
Compartir o incrustar documentos
¿Le pareció útil este documento?
¿Este contenido es inapropiado?
Denunciar este documentofisca
Copyright:
© All Rights Reserved
Formatos disponibles
Descargue como PDF, TXT o lea en línea desde Scribd
0 calificaciones0% encontró este documento útil (0 votos)
22 vistas5 páginasDNA Mismatch Repair (MMR) Cristian Cuaspud
Cargado por
Criistiian Dariioofisca
Copyright:
© All Rights Reserved
Formatos disponibles
Descargue como PDF, TXT o lea en línea desde Scribd
Está en la página 1de 5
Reparación
desajustes
del ADN
Est: Cristian Dario
Cuaspud Ruiz
Maestria Fisica Medica
Curso Radiobiologia
1 Universidad Nacional de Colombia- Bogotá DC.
Este mecanismo de reparación, es un sistema para identificar y corregir (la inserción,
eliminación e incorporación errónea ) de bases las nitrogenadas (A,T,G Y C) que
pueden surgir durante la replicación del ADN.
* La reparación de desajustes es específica de la hebra. Durante la síntesis de ADN, la
cadena (hija) recién sintetizada comúnmente incluirá errores. Para comenzar la
reparación, la maquinaria de reparación de desajustes distingue la hebra recién
sintetizada de la plantilla (parental).
** Los ejemplos de bases no coincidentes incluyen un emparejamiento G/T o A/C.
*** Los desajustes se deben comúnmente a la tautomerización de las bases durante la
replicación del ADN.
**** El daño se repara mediante el reconocimiento de la deformidad causada por el
desajuste, determinando la hebra plantilla y no plantilla, y extirpando la base mal
incorporada y reemplazandola con el nucleótido correcto. El proceso de eliminación
implica algo más que el propio nucleótido no coincidente. Se pueden eliminar unos
pocos o hasta miles de pares de bases de la cadena de ADN recién sintetizada.
2 Universidad Nacional de Colombia- Bogotá DC.
Tautomerización
Watson y Crick postularon la tautomería de las bases
Las formas tautoméricas son formas de las bases nitrogenadas que no poseen los
grupos funcionales correspondientes a la base a la que pertenecen pudiendo
ocasionar mutaciones. dichas formas existen debido a la migración de los electrones
por los dobles enlaces conjugados.
Tautomería imina-amina Tautomería ceto-enólica (lactama-lactima)
Interconvierte un amino (—NH2) Interconvierte un grupo ceto (=O) y enol
extracíclico en un imino (=NH) cerca de (—OH) extracíclicos cerca de un N cíclico.
un N cíclico. Se puede dar en G, A y C. Se puede producir en la G, C, T.
3 Universidad Nacional de Colombia- Bogotá DC.
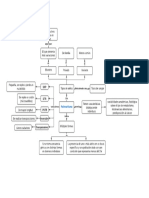
Representación
esquemática de
la reparación de
desajustes de
ADN.
4 Universidad Nacional de Colombia- Bogotá DC.
1. La proteína enzimática MutS identifica el
desajuste y se une al ADN.
2. La proteína MutL se une en la misma
posición donde MutS se une con el ADN
3. A esto le sigue la división del ADN
monocatenario con error por la acción de
la enzima endonucleasa (una proteína de
degradación del ADN)
4. Una proteína helicasa desenrolla el ADN
y lo estabiliza mediante proteínas ligantes
de ADN de cadena sencilla (SSB)
5. SSB se une a la proteína RecJ y elimina
la hebra de error
6. Finalmente, la enzima polimerasa III
resintetiza la hebra de ADN correcta, y la
brecha se llena con la enzima ligasa I
Radiation Biology for Medical Physicists. C.S. Sureka
5 and C. Armpilia, 2017
También podría gustarte
- Tema 1 Bioquímica IiDocumento16 páginasTema 1 Bioquímica IiSara CoiraAún no hay calificaciones
- Reparación Del ADN 2Documento6 páginasReparación Del ADN 2Alex Valle SotoAún no hay calificaciones
- Reparación Del AdnDocumento22 páginasReparación Del AdnRubí RecinosAún no hay calificaciones
- Cuestionario Daño Al ADNDocumento8 páginasCuestionario Daño Al ADNmaria paula cely alvaradoAún no hay calificaciones
- Reparación de Adn, ImpresionDocumento24 páginasReparación de Adn, Impresionmiguel angelAún no hay calificaciones
- Carpeta Investigativa Iii ParcialDocumento83 páginasCarpeta Investigativa Iii Parcialcesia rosalesAún no hay calificaciones
- Mecanismos de Reparación Del ADN CORREGIDODocumento54 páginasMecanismos de Reparación Del ADN CORREGIDOBiiancaRodriguez100% (1)
- Mecanismos de Reparación Del ADNDocumento7 páginasMecanismos de Reparación Del ADNVIOLETA NICOLE LINARES PORRASAún no hay calificaciones
- Pluginfile - Php1326288mod Resourcecontent1dannosyreparacion IIC 2023 PDFDocumento29 páginasPluginfile - Php1326288mod Resourcecontent1dannosyreparacion IIC 2023 PDFmgxt4bbv4jAún no hay calificaciones
- Recombinación GenéticaDocumento6 páginasRecombinación GenéticaAve FenixAún no hay calificaciones
- Semana 6 - Práctica 1Documento25 páginasSemana 6 - Práctica 1GERMAN LÔPEZZAún no hay calificaciones
- Cuestionario Mecanismos de Reparacion Del DNADocumento4 páginasCuestionario Mecanismos de Reparacion Del DNAMicaAún no hay calificaciones
- Reparación Del DNADocumento3 páginasReparación Del DNAAlexa CuevasAún no hay calificaciones
- Clase de ADN 2Documento23 páginasClase de ADN 2Laura BarberaAún no hay calificaciones
- Ejercicios de GenéticaDocumento32 páginasEjercicios de GenéticaSheila GarcíaAún no hay calificaciones
- Mecanismos de Reparación Del DnaDocumento10 páginasMecanismos de Reparación Del Dnalaura torresAún no hay calificaciones
- Biologia Selectividad Examen Resuelto Canarias 2 WWW - Siglo21x.blogspotDocumento7 páginasBiologia Selectividad Examen Resuelto Canarias 2 WWW - Siglo21x.blogspotmonografiasxAún no hay calificaciones
- Mecanismos de Reparación - ExposiciónDocumento9 páginasMecanismos de Reparación - Exposiciónalfredo camposAún no hay calificaciones
- Recombinaci�n gen�ticaDocumento4 páginasRecombinaci�n gen�ticaEl Tío ShadowAún no hay calificaciones
- Tema 2 Biologia Molecular Reparacion Del AdnDocumento15 páginasTema 2 Biologia Molecular Reparacion Del AdnAldo Ezequilla RamirezAún no hay calificaciones
- Exposicion 1Documento17 páginasExposicion 1Tefy VegaAún no hay calificaciones
- Examen biologíaDocumento6 páginasExamen biologíaRocio De Miguel GarciaAún no hay calificaciones
- Taller ReparacionDocumento6 páginasTaller ReparacionMeriele AndreaAún no hay calificaciones
- Taller de Genoma y Reproducción Celular Segunda ParteDocumento6 páginasTaller de Genoma y Reproducción Celular Segunda ParteZharick NuñezAún no hay calificaciones
- Resumen Reparacion AdnDocumento6 páginasResumen Reparacion AdnKike FernandezAún no hay calificaciones
- Cecil 1Documento24 páginasCecil 1Sofía L.Aún no hay calificaciones
- 05 Los GenesDocumento16 páginas05 Los Geneselizabeth marsigliaAún no hay calificaciones
- Replicación Del DNA 2022Documento101 páginasReplicación Del DNA 2022jeniferAún no hay calificaciones
- Quimica L 1Documento8 páginasQuimica L 1Diego Linero RamirezAún no hay calificaciones
- Tema 6 (II) CMC. Biotecnología.Documento31 páginasTema 6 (II) CMC. Biotecnología.pilukillaAún no hay calificaciones
- 08 Tema 7 GenéticaDocumento38 páginas08 Tema 7 GenéticaMÉNDEZ SARABIA ESTEFANI LETICIAAún no hay calificaciones
- Tarea 2 Unidad I Ingeniería Forestal y AmbientalDocumento5 páginasTarea 2 Unidad I Ingeniería Forestal y AmbientalCRISTIAN DE LA TORRE BENDEZUAún no hay calificaciones
- Enlaces QuimicosDocumento6 páginasEnlaces QuimicosStefania LAún no hay calificaciones
- Tema 3. Mutación y Reparación.Documento11 páginasTema 3. Mutación y Reparación.Llucia Ramis LladoAún no hay calificaciones
- Actividad Biologia - 11-2Documento6 páginasActividad Biologia - 11-2nataliaAún no hay calificaciones
- Junio 2016 Opción ADocumento4 páginasJunio 2016 Opción AJorge Azuara ChulviAún no hay calificaciones
- GenéticaDocumento6 páginasGenéticalore05Aún no hay calificaciones
- Apuntes 5.2 - 4 EsoDocumento7 páginasApuntes 5.2 - 4 Esoara232628Aún no hay calificaciones
- Práctica #12Documento14 páginasPráctica #12GUSTAVO DANIEL CALVA CAMPOVERDEAún no hay calificaciones
- Capitulo 13. Aplicacion GeneticaDocumento62 páginasCapitulo 13. Aplicacion GeneticaMayra RiosAún no hay calificaciones
- Síntesis ADNDocumento7 páginasSíntesis ADNSantiago RochaAún no hay calificaciones
- Resumen Genetica BCM 2021Documento59 páginasResumen Genetica BCM 2021Florencia SierraAún no hay calificaciones
- Tema16 MutacionesDocumento15 páginasTema16 MutacionesMaria RosseAún no hay calificaciones
- Biología: Examen de 2° Parcial sobre Tema 1Documento8 páginasBiología: Examen de 2° Parcial sobre Tema 1Saail GoxAún no hay calificaciones
- Biologia Selectividad Examen Resuelto Canarias 4 WWW - Siglo21x.blogspotDocumento8 páginasBiologia Selectividad Examen Resuelto Canarias 4 WWW - Siglo21x.blogspotmonografiasxAún no hay calificaciones
- Genética y FarmacogenómicaDocumento104 páginasGenética y FarmacogenómicaPablo Munarriz MillanAún no hay calificaciones
- Documento BiolDocumento1665 páginasDocumento BiolAlexandra MirandaAún no hay calificaciones
- genesDocumento16 páginasgenesFlavia MorenoAún no hay calificaciones
- GUÍA 1 ELEC Modelo y Replicación de ADNDocumento8 páginasGUÍA 1 ELEC Modelo y Replicación de ADNCarla Javiera Abarca0% (1)
- Duplicación Del ADNDocumento15 páginasDuplicación Del ADNPaula Alexandra Maza SerratoAún no hay calificaciones
- Taller Bases Moleculares y Celulares de La HerenciaDocumento2 páginasTaller Bases Moleculares y Celulares de La HerenciaKareth MuñozAún no hay calificaciones
- Replicación DNADocumento4 páginasReplicación DNAz5rrz9xgfmAún no hay calificaciones
- Extraccion de DNADocumento10 páginasExtraccion de DNACoello trinidad bartolo0% (2)
- Reparación de DNADocumento4 páginasReparación de DNAFrancisco SebastianAún no hay calificaciones
- Informe 10 - Replicación Del Adn. Reparación Del Adn.Documento11 páginasInforme 10 - Replicación Del Adn. Reparación Del Adn.Zea Guerrero MarialejandraAún no hay calificaciones
- Mecanismos de Reparación de Daños en La Molécula de AdnDocumento3 páginasMecanismos de Reparación de Daños en La Molécula de AdnGabriel Omar Hernández RamírezAún no hay calificaciones
- Mecanismo de Reparación de Quiebres en Doble Cadena DSBDocumento5 páginasMecanismo de Reparación de Quiebres en Doble Cadena DSBMiguel CarrizoAún no hay calificaciones
- Bases Quimicas de La HerenciaDocumento38 páginasBases Quimicas de La HerenciaRony Huañec MedinaAún no hay calificaciones
- Introducción a la Biología: RESÚMENES UNIVERSITARIOSDe EverandIntroducción a la Biología: RESÚMENES UNIVERSITARIOSCalificación: 5 de 5 estrellas5/5 (1)
- Taller 3 - Valeria Gutierrez Ruiz - 05-03-2021Documento6 páginasTaller 3 - Valeria Gutierrez Ruiz - 05-03-2021Criistiian DariiooAún no hay calificaciones
- Compuertas UniversalesDocumento10 páginasCompuertas UniversalesFernandoAún no hay calificaciones
- Taller 2 - Valeria Gutierrez Ruiz - 01-03-2021Documento3 páginasTaller 2 - Valeria Gutierrez Ruiz - 01-03-2021Criistiian DariiooAún no hay calificaciones
- Taller 4 - Valeria Gutierrez Ruiz - 17-03-2021Documento7 páginasTaller 4 - Valeria Gutierrez Ruiz - 17-03-2021Criistiian DariiooAún no hay calificaciones
- Taller 1 - Valeria Gutierrez Ruiz - 26-02-2021Documento3 páginasTaller 1 - Valeria Gutierrez Ruiz - 26-02-2021Criistiian DariiooAún no hay calificaciones
- Recombinacion HomologaDocumento5 páginasRecombinacion HomologaCriistiian DariiooAún no hay calificaciones
- Taller 5 - Valeria Gutierrez Ruiz - 12-04-2021Documento6 páginasTaller 5 - Valeria Gutierrez Ruiz - 12-04-2021Criistiian DariiooAún no hay calificaciones
- Intr Geofis 2021 I Parcial 2Documento2 páginasIntr Geofis 2021 I Parcial 2Criistiian DariiooAún no hay calificaciones
- Apoptosis RadiobiologiaDocumento5 páginasApoptosis RadiobiologiaCriistiian DariiooAún no hay calificaciones
- SSB Repair Santiago MoyaDocumento18 páginasSSB Repair Santiago MoyaCriistiian DariiooAún no hay calificaciones
- Reparación Daño Bases Adrian David OrdoñezDocumento6 páginasReparación Daño Bases Adrian David OrdoñezCriistiian DariiooAún no hay calificaciones
- El Accidente Radioactivo de Goiania: Una Experiencia en La Enseñanza de Cts Utilizando El Aprendizaje Centrado en EventosDocumento10 páginasEl Accidente Radioactivo de Goiania: Una Experiencia en La Enseñanza de Cts Utilizando El Aprendizaje Centrado en EventosCriistiian DariiooAún no hay calificaciones
- Manual Nomex v2Documento108 páginasManual Nomex v2Criistiian DariiooAún no hay calificaciones
- FINAL Posibles PerguntasDocumento12 páginasFINAL Posibles PerguntasFelipe DantasAún no hay calificaciones
- Actividad 2 BiologiaDocumento6 páginasActividad 2 BiologiaRICARDO FLORES ROMEROAún no hay calificaciones
- Genética UCSUR - T1 - Bases Moleculares de La HerenciaDocumento57 páginasGenética UCSUR - T1 - Bases Moleculares de La HerenciaDiego Cruzado oroscoAún no hay calificaciones
- Unidad 16 - Transcripcion y TraducciónDocumento19 páginasUnidad 16 - Transcripcion y TraducciónLaura MoyaAún no hay calificaciones
- Informe de La Maqueta de BiologiaDocumento6 páginasInforme de La Maqueta de BiologiaDanilo RomeroAún no hay calificaciones
- Taller de BiologiaDocumento4 páginasTaller de BiologiaEdwYona herfigue robriAún no hay calificaciones
- Gentica CuantitativaDocumento22 páginasGentica CuantitativaDANIELAún no hay calificaciones
- Fases de La MeiosisDocumento3 páginasFases de La Meiosissofia garciaAún no hay calificaciones
- Examen Recup CCientificaDocumento3 páginasExamen Recup CCientificaVioleta GonzaAún no hay calificaciones
- Informe de Resultados Her2 RevisadoDocumento2 páginasInforme de Resultados Her2 RevisadoAguilar PattyAún no hay calificaciones
- Work Papers Genetica VDocumento6 páginasWork Papers Genetica Vfacha fachosoAún no hay calificaciones
- Operon TRPDocumento13 páginasOperon TRPNicole YepesAún no hay calificaciones
- Estructura y funciones del núcleo celularDocumento29 páginasEstructura y funciones del núcleo celularRubí RecinosAún no hay calificaciones
- Historia Ecuador GenesDocumento20 páginasHistoria Ecuador GenesDavid ChicaizaAún no hay calificaciones
- RetoDocumento2 páginasRetoAna VillamarinAún no hay calificaciones
- Mutaciones GenéticasDocumento11 páginasMutaciones GenéticasAllemby Alejandro Hau MaglahAún no hay calificaciones
- Tema 7. Genética Molecular.Documento11 páginasTema 7. Genética Molecular.Natalia Milla LopezAún no hay calificaciones
- Rúbrica Del Prueba DiagnósticaDocumento5 páginasRúbrica Del Prueba DiagnósticaHeidy Prisscila MontealegreAún no hay calificaciones
- Rasgos genéticos humanos y herencia mendelianaDocumento9 páginasRasgos genéticos humanos y herencia mendelianaDiana TarazonaAún no hay calificaciones
- Epigenetic2 Modifications and Human Diseases - Af.esDocumento12 páginasEpigenetic2 Modifications and Human Diseases - Af.esJUAN DIEGO CABEZAS PORRASAún no hay calificaciones
- Tarea Semana 4Documento7 páginasTarea Semana 4Yuneydis GuzmanAún no hay calificaciones
- Vectores-Nociones Bibliotecas Parte 1 y 2-2021 PDFDocumento35 páginasVectores-Nociones Bibliotecas Parte 1 y 2-2021 PDFtito tatoAún no hay calificaciones
- Práctica 13Documento6 páginasPráctica 13ABIGAIL PATRICIA JUCHANI RODRIGUEZAún no hay calificaciones
- Análisis de polimorfismos en una población de cabras criollasDocumento27 páginasAnálisis de polimorfismos en una población de cabras criollasJoaquin Faillace CastañoAún no hay calificaciones
- Problemas 2a Práctica EvoluciónDocumento4 páginasProblemas 2a Práctica EvoluciónKaren MonccaAún no hay calificaciones
- Saber Qué Es y Cómo Afecta La Herencia GenéticaDocumento15 páginasSaber Qué Es y Cómo Afecta La Herencia Genética125 hkAún no hay calificaciones
- Adn, Arn y Ciclo CelularDocumento21 páginasAdn, Arn y Ciclo CelularCesia PérezAún no hay calificaciones
- PolimorfismosDocumento1 páginaPolimorfismosValentina Pimentel PatiñoAún no hay calificaciones
- Conceptos Basicos de Genetica Clinica para La PracDocumento7 páginasConceptos Basicos de Genetica Clinica para La PracNatalia TorresAún no hay calificaciones
- EXAMEN MENSUAL DE BIOLOGÍA EN I.E.P. DON BOSCODocumento2 páginasEXAMEN MENSUAL DE BIOLOGÍA EN I.E.P. DON BOSCOraul condoriAún no hay calificaciones