Documentos de Académico
Documentos de Profesional
Documentos de Cultura
Biol 240
Cargado por
Leonidas LarrondoTítulo original
Derechos de autor
Formatos disponibles
Compartir este documento
Compartir o incrustar documentos
¿Le pareció útil este documento?
¿Este contenido es inapropiado?
Denunciar este documentoCopyright:
Formatos disponibles
Biol 240
Cargado por
Leonidas LarrondoCopyright:
Formatos disponibles
lOMoARcPSD|27237116
Solemnes biologia moleculas
Biología Molecular (Universidad Nacional Andrés Bello)
Studocu no está patrocinado ni avalado por ningún colegio o universidad.
Descargado por Leonidas Larrondo Cazanga (leonyomg17@gmail.com)
lOMoARcPSD|27237116
SOLEMNE 1
Pregunta 1
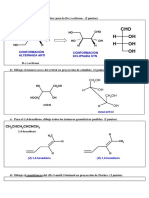
En referencia al siguiente nucleótido responda lo siguiente
a) ¿de qué nucleótido se trata?
b) Asigne la figura geométrica con la estructura o función que se indica
- Parte de la molécula que es agregada a un ácido nucleótido en crecimiento
- Átomos que forman puentes de hidrógenos con la base complementaria
- Carbono 5´
- Grupo químico que diferencia un nucleótido de un desoxinucleotido
- Enlace N-glucosídico
- Enlace fosfodiéster
- Grupo químico requerido para permitir la elongación la cadena de acido nucleico en
crecimiento
A) Se trata de adenosina trifosfato
B) Flecha: Grupo químico requerido para permitir la elongación de la cadena de ácido nucleico en
crecimiento.
Circulo: Grupo químico que diferencia un nucleótido de un desoxinucleotido
Cabeza de flecha: Enlace N-glicosídico
Asteriscos: Átomos que forman puentes de hidrogeno con la base complementaria.
Rectángulo: Parte de la molécula que es agregada a un ácido nucleico en crecimiento
Descargado por Leonidas Larrondo Cazanga (leonyomg17@gmail.com)
lOMoARcPSD|27237116
Pregunta 2
A) ¿Cuáles serian los efectos de los siguientes tratamientos sobre la curva de ¨melting¨ de una
solución de DNA de doble hebra? A) disminución de la fuerza iónica de la solución; B)
aumento de la fuerza iónica de la solución; C) agregar ion magnesio a la solución.}
B) En 1963, Jerome Vinogard extrajo el DNA circular del virus del papiloma. La composición se
ajusta a las reglas de Chargaf, pero observo dos bandas en lugar de una única al centrifugar
el DNA en un gradiente de cloruro de cesio. ¿A que se deben estas dos bandas?
2-a: Al disminuir la fuera iónica de la solución, implica disminuir los contra iones que neutralizan la alta
carga negativa de los fosfatos del DNA de la doble hebra, razón por la cual la unión entre las hebras se hace
más inestable y, en consecuencia, se requiere una menor temperatura para fundir el DNA.
2-b) En este caso la unión del DNA se vuelve más estable pues se logra neutralizar las cargas negativas del
esqueleto fosfodiester y, se requerirá una temperatura mayor para fundir la doble hélice.
2-c) Idem a opción b. Resp:
2B) Al observar el grafico de la distribución del DNA del virus, post centrifugaciones en gradiente se
observan dos peak distribuidos, uno cerca del final del tubo (indica mayor mayor tamaño molecular) y el
otro peak cerca del inicio del tubo (indicando menor tamaño molecular). Al tratarse de un DNA de doble
hebra circular, lo que este resultado indica, es que estamos observando la co-existencia de dos formas del
mismo DNA: El peak ubicado a 6.782 corresponde al DNA circular relajado y que presenta mayor tamaño
molecular y, el ubicado a 6.65 corresponde al mismo DNA del virus pero que adopta una estructura de
sobrenrrollamiento haciéndose más compacto y por eso migra en una posición equivalente a ser un DNA
de menor tamaño
Descargado por Leonidas Larrondo Cazanga (leonyomg17@gmail.com)
lOMoARcPSD|27237116
Pregunta 3
A) Durante la síntesis de DNA, ¿Por qué las cadenas de DNA en crecimiento se sintetizan a
partir de nucleótidos trifosfato y no a partir de nucleótidos monofosfato?
B) Explique cómo solucionan las células eucariotas el problema del acortamiento de los
telómeros que se producen en cada ciclo de división celular. ¿Por qué no se da este
problema en las células procariotas?
3-A) Porque para que se forme el enlace éster entre el último nucleótido de la cadena en crecimiento y el
nucleótido que se incorpora a ella es necesaria energía, y ésta se obtiene de la escisión de los dos grupos
fosfato terminales del nucleótido trifosfato entrante.
3-B) Lo solucionan por la acción de la telomerasa, un enzima que, utilizando como molde una cadena de
RNA que lleva incorporada como grupo prostético, añade algunas decenas de nucleótidos sin información
a la cadena que termina en el extremo 3' de manera que el equipo enzimático de la replicación puede
alargar la cadena complementaria compensando así el acortamiento. Este problema no se da en
procariotas, pues su DNA es circular cerrado
Descargado por Leonidas Larrondo Cazanga (leonyomg17@gmail.com)
lOMoARcPSD|27237116
Pregunta 4
Las siguientes proteínas participan en la replicación del DNA procariota, DnaA, DnaB, DnaC, DnaG,
SSB, DNA pol III, complejo γ/т (tau), β clamp (sliding clamp), entre otros. Al respecto, para cada
proteína de bacteria nombre la correspondiente enzima equivalente en eucariota y describa
brevemente la función que realiza cada una en la replicación en eucariotas.
DnaA: Equivale a ORC que reconoce el origen de replicación, lo que permite atraer el resto de los
componentes necesarios para formar el replisoma.
DnaB: Es la helicasa replicativa en bacterias, luego equivale al complejo de helicasa replicativa eucariota
CMG (CDC45:MCM2-7:GINS)
DnaC. Equivale a CDT1, la proteína que carga a MCM2-7 en eucariotas, que es la helicasa que inicia la
apertura de la doble hélice al formar el DH.
DnaG: Equivale a la primasa, la cual en eucariotas forma parte de la DNA pol alfa. Actividad que permite
sintetizar el partidor de RNA.
SSB: equivale a las proteínas RPA de eucariotas, que unen DNA de simple hebra durante la replicación, para
evitar que se reasocie y asi, favorecer la síntesis de DNA.
DNA pol III: Es la polimerasa replicativa en bacterias, su equivalente en eucariotas son las DNA pol
delta/épsilon.
Complejo γ/τ (tau): corresponde a la actividad que carga la abrazadera deslizante en bacterias, lo que tiene
su contrapartida en eucariotas con la proteína RFC (replication factor C)
Beta clamp: corresponde a la proteína que aumenta la procesividad de la DNA polimerasa, que tiene por
nombre en eucariotas PCNA, antígeno nuclear de proliferación celular)
Descargado por Leonidas Larrondo Cazanga (leonyomg17@gmail.com)
lOMoARcPSD|27237116
Pregunta 5
Sobre la replicación en procariota. En la siguiente figura se destacan 8 componentes y estructuras
del replisoma bacteriano
A) Identifique y nombre los 8 elementos destacados
B) ¿Qué ocurriría con el proceso de replicación si una bacteria adquiera una mutación que
cause un defecto en la función de los elementos N°1, N°4 y N°6? Fundamente su respuesta
1 Sliding clamp (DnaN, subunidad beta)
2 DNA polimerasa III
3 Hebra lider
4 Sliding clamp loader (o complejo gamma)
5 tau
6 Helicasa
7 Hebra discontinua
8 Fragmento de Okazaki
B) Si falta Nº1. Sin la proteína Dnan (sliding clamp) la DNA polimerasa III no tendría la procesividad necesaria
para poder amplificar todo el ADN. La DNA polimerasa se <soltaría= del ADN con mucha más facilidad. Si
falta Nº4. Sin el complejo gamma (sliding clamp loader) no se podrá cargar al ADN templado la proteína
DnaN (sliding clamp) lo que también resultaría en una pérdida de la procesividad de la DNA polimerasa III.
Si falta Nº6. Sin la proteína helicasa no se podrá abrir la doble hebra de ADN de manera de exponer las
hebrás templado. De esta forma no podrá ocurrir la replicación del ADN
Descargado por Leonidas Larrondo Cazanga (leonyomg17@gmail.com)
lOMoARcPSD|27237116
Pregunta 6
Sobre el proceso del inicio de la replicación bacteriana. En la siguiente figura se muestra un
esquema de oriC bacteriano.
A) Nombre y explique la función que tiene las diferentes secuencias de repetidos de 13pb y
de 9pb dentro de oriC
B) Explique el proceso de inicio de la replicación desde oriC hasta la formación de la burbuja
de replicación. Nombre las proteínas involucradas y sus orígenes de unión, fundamentando
el mecanismo por el cual permiten el inicio de la replicación.
A) La región de repetidos de 13pb es una región rica en AT y tienen la función de permitir que se abra
el ADN para generar la burbuja de replicación. La región de los repetidos de 9pb es el lugar donde
se une la proteína DnaA. La unión de DnaA y su polimeración permite torcer la región del oriC para
abrir la región de repetidos de 13pb.
B) El proceso comienza con la unión de DnaA a las regiones de repetidos de 9pb, estas proteínas
comienza a interactuar entre ellas generando una torsión del ADN que lleva a que las regiones de
13pb se abran, debido a que es una región rica en AT. Una vez que el ADN de la región de repetidos
de 13pb se expone, llegan las proteínas SSB y el complejo DnaB/DnaC. Lo que permite finalmente
que la DnaB (helicasa) se cargue en las dos hebras templados.
Descargado por Leonidas Larrondo Cazanga (leonyomg17@gmail.com)
lOMoARcPSD|27237116
Pregunta 7
El cambio de un solo nucleótido en el gen apoE puede ser indicativo de riesgo de padecer
alzheimer. Usted obtuvo muestra de DNA genomico de 4 pacientes y realizo una secuenciación por
Sanger de una porción del gen apoE. Usando una persona sana como control. En la figura muestra
la secuenciación de Sanger.
a) Mencione los fundamentos de la técnica de secuenciación por Sanger
b) ¿Qué paciente(s) tienen la mutación? Marque con un circulo
c) Escriba la secuencia de DNA del control y de cada paciente en la dirección 5´a 3´
A) El método de secuenciación ideado por Sanger se basa en el empleo de dideoxinucleótidos que carecen
del grupo hidroxilo del carbono 3', de manera que cuando uno de estos nucleótidos se incorpora a una
cadena de ADN en crecimiento, esta cadena no puede continuar elongándose. Esto sucede porque la ADN
polimerasa necesita un grupo terminal 3’ OH para añadir el siguiente nucleótido y el dideoxinucleótido
incorporado carece de este grupo hidroxilo. De esta forma se preparan 4 reacciones de amplificación donde
en cada una además se agrega una pequeña cantidad de cada dideoxinucleótido. De esta forma en el tubo
donde se agrega un dideoxinucleotido de Adenina se generarán fragmentos de distinto tamaño
corresponidentes a la terminación de la cadena en cada posición correspondiente a una adenina. La
comparación del patrón de los fragmentos generados por los 4 dideoxinucleotidos en un gel de agarosa o
poliacrilamida permite leer la secuencia de ADN desde abajo hacia arriba (5´ a 3´)
B) Los pacientes 2 y 3 tienen la mutación (T a G en la posición 4).
C) Control. 5´ ATATTGTCGTAG 3`
Paciente 1 5´ ATATTGTCGTAG 3`
Paciente 2 5´ ATAGTGTCGTAG 3`
Paciente 3 5´ ATAGTGTCGTAG 3`
Paciente 4 5´ ATATTGTCGTAG 3`
Descargado por Leonidas Larrondo Cazanga (leonyomg17@gmail.com)
lOMoARcPSD|27237116
Pregunta 8
Su laboratorio necesita amplificar las regiones de 1000pb desde el genoma de E. coli que se
muestra en la figura. Para poner a prueba a sus 4 estudiantes le pide a cada una y uno de ellos que
diseñen partidores desde un cultivo de E. coli que usted preparo y luego realicen la reacción de
PCR.
En el gel de agarosa que se muestra a continuación se puede ver el resultado de las 4 reacciones
de PCR.
A) ¿Cuál(es) de las 4 amplificación(es) considera usted exitosa? ¿Por qué? Fundamente su
respuesta.
B) Explique que pudo haber ocurrido en todas aquellas condiciones donde no se obtuvo el
resultado deseado. Fundamente su respuesta
A) De las 4 amplificaciones la numero 1 es la exitosa. Esto debido a que se aprecia una sola banda nítida
que corresponde al tamaño esperado de 1000pb.
B) En los otros 3 carriles tenemos problemas de amplificación.
Carril 2: Se observa una banda del tamaño esperado pero también se observan dimeros de partidores
(primers) al final de la corrida. Los dimeros de partidores pueden formarse por interacciones inter o intra
moleculares. Siendo la causa un mal diseño de estos partidores.
Carril 3: Se observa una banda del tamaño esperado pero también se observa una amplificación inespecífica
de 500pb. La causa puede ser un mal diseño de partidores en caso que alguno de los partidores (primer 1 o
primer 2) pueda unirse a una región interna de la región a amplificar.
Carril 4: En este carril no se observa ninguna banda por ende no hubo amplificación. Esto puede haber
ocurrido por numerosas razones incluyendo: 1) un DNA templado degradado o contaminado que haya
inhibido la reacción de PCR, 2) la omisión de algún componente de la reacción de PCR que haya impedido
la amplificación de la región de 1000pb. (Nota: Cualquier otra razón que se entregue que se ajuste a la
ausencia de amplificación será dada como buena).
Descargado por Leonidas Larrondo Cazanga (leonyomg17@gmail.com)
lOMoARcPSD|27237116
Pregunta 9
Para determinar la secuencia de una región de ADN de interés decide realizar una reacción de
secuenciación utilizando el Método de Sanger. El gel que obtuvo se muestra en la figura
A) Explique el fundamento de la técnica de secuenciación de Sanger.
B) Escriba la secuencia de ADN secuenciada (5’ a 3’)
C) Escriba la secuencia de ADN de la hebra templado que generó este resultado.
A) El método de secuenciación de Sanger se basa en el empleo de dideoxinucleótidos que carecen
del grupo hidroxilo del carbono 3’, de manera que cuando uno de estos nucleótidos se incorpora a
una cadena de ADN en crecimiento, está cadena no puede continuar alongándose. Esto sucede
porque el ADN polimerasa necesita un grupo terminal 3’ OH para añadir el siguiente nucleótido y
el dideoxinucleotido incorporado caracense de este grupo hidroxilo
B) 5’ ATATTGTCGTAG 3’
C) 3’ TATAACAGCATC 5’
Descargado por Leonidas Larrondo Cazanga (leonyomg17@gmail.com)
lOMoARcPSD|27237116
Pregunta 10
Usted busca determinar la expresión de un gen en su línea celular favorita. Usted lisa las células y
purifica el ADN utilizando el método de extracción por fenol-cloroformo, y realiza un qPCR
utilizando la tecnología de Sybr Green. La curva de amplificación que obtiene se ve en la siguen
figura.
A. ¿Por qué razón su curva experimental del <Experimento 1= no se comportó como usted
esperaba?
B. En el experimento 2, usted determina la expresión de su gen bajo distintas condiciones
experimentales (A-E) siendo su condición basal la curva de amplificación de color verde.
¿En cuál(es) condición(es) experimental usted tiene mayor cantidad del ADN blanco? ¿Y
por qué?
A.- una curva experimental de amplificación puede alejarse de la curva teórica o esperada por numerosas
razones incluyendo: presencia de inhibidores y contaminantes del método de extracción o naturaleza de
la muestra o un mal diseño de partidores.
B.- hay una mayor cantidad de ADN blanco en la muestra de las condiciones A y B. Esto se debe a que
alcanzaron la fase exponencial de amplificación antes que la condición basal, lo que se traduce en Cts
menores (ciclo en los cuales la fluorescencia generada por la amplificación de ADN es mayor a la
fluorescencia basal)
Descargado por Leonidas Larrondo Cazanga (leonyomg17@gmail.com)
lOMoARcPSD|27237116
Pregunta 11
A) ¿Cuáles son los tres componentes imprescindibles que debe tener una secuencia de DNA
lineal para comportarse como un cromosoma estable y cuál es la función de cada uno?
B) Si decimos que el porcentaje guanina del DNA de un organismo es del 17% ¿Cuál es el
porcentaje de cada una de las bases en el DNA? Indique su razonamiento.
A) Telómero: secuencia de DNA repetida hasta 250 veces, encontrada en los extremos de los
cromosomas y, que tiene como función evitar el acortamiento de los mismos evitando en
consecuencia, la inestabilidad genómica y perdida de secuencias subtelomericas
Origen de replicación: corresponde a una zona del cromosoma desde donde se inicia la
replicación del DNA de manera bidireccional
Centrómero: región del cromosoma que se caracteriza por una alta compactación y que durante
la mitosis sirve de andamio para la formación del cinetocoro, estructura donde se ancla la fibra
del huso mitótico, facilitando la segregación de los cromosomas a las células hijas.
B) 17%+17%=34%
100% - 34% =66%
66% corresponde a bases de adenina y timina, como son complementarias correspondería 33%A y 33%T.
Entonces, 17%G y 17%C
Descargado por Leonidas Larrondo Cazanga (leonyomg17@gmail.com)
lOMoARcPSD|27237116
Pregunta 12
El diagrama representa la burbuja de replicación que se ha formado en un organismo de
replicación bacteriano. La línea punteada muestra donde la replicación comenzó
A) Indique en cuales cuadrantes (A,B,C o D) se formaran las hebras lidere y las hebras rezagada
durante la replicación del cromosoma
B) ¿Cuáles son las señales y proteínas involucradas en la replicación del cromosoma desde
oriC?
A B
C D
A) Hebra líder: cuadrante A y D
Hebra rezagada: cuadrante B y C
B) La iniciación de la replicación del cromosoma bacteriano comienza en una región de DNA
conocida como el oriC. Origen que posee secuencias de dos tipos: 1) repetidas de 13pb en
una región rica en AT y 2) repetidas de 9pb que median la unión a la proteína DnaA
La replicación comienza mediante la unión de la proteína DnaA a los sitios de 9pb y la formación
de un filamento helicoidal que favorece el desenrollamiento de la región de 13pb rica en AT. Esto
además recluta el complejo DnaB/DnaC donde DnaC es la proteína que carga al DNA la proteína
helicasa DnaB en parte gracias a su afinidad por la proteína DnaA. Esto finalmente posiciona la
helicasa DnaB en la naciente burbuja de replicación
Descargado por Leonidas Larrondo Cazanga (leonyomg17@gmail.com)
lOMoARcPSD|27237116
Pregunta 14
Usted se encuentra estudiando 2 nuevas bacterias que encontró en un salar en el norte de Chile.
Para estudiar el mecanismo de replicación del DNA decide repetir el clásico experimento realizado
por Meselson y Stahl usando isotipos de Nitrógenos. La siguiente figura muestra el resultado de su
experimento luego de dos generaciones de haber crecidos en presencia de isotipos de nitrógenos
14
¿Cuál es el mecanismo de replicación del DNA utilizado por cada bacteria? Justifique su respuesta
Replicación semiconservativa bacteria 1
Replicación conservativa bacteria 2
Descargado por Leonidas Larrondo Cazanga (leonyomg17@gmail.com)
lOMoARcPSD|27237116
Solemne 2
Pregunta 1
Para estudiar la regulación del operón lac usted construye una sonda para estudiar el mecanismo
de regulación del operón mediante, ADN Footprinting y EMSA, en 3 condiciones que reflejan
distintos niveles de glucosa y lactosa. La sonda (ver figura) corresponde a todo el operón lac
incluyendo su promotor, todos los elementos regulatorios y los 3 genes estructurales. De acuerdo
con la figura:
A) ADN Footprinting: El experimento se realizó incubando la sonda (ver figura) con todos las
proteínas regulatorias y la maquinaria de transcripción procarionte. Describa los resultados
del footprinting destacando todas las proteínas que se encuentran unidas al ADN en las 3
condiciones.
B) EMSA: El experimento se realizó incubando la sonda (ver figura) solamente con la proteína
LacI. Describa y explique los resultados del EMSA destacando todas las proteínas que se
encuentran unidas al ADN en las 3 condiciones
C) Tomando en cuenta los resultados de footprinting y EMSA, ¿cuáles son las concentraciones
esperadas de glucosa, lactosa y cAMP en las condiciones 1, 2 y 3 (alta, baja o basal) y como
seria la expresión del operón en cada una de ellas? Fundamente su respuesta mencionada
los resultados del footprinting y EMSA.
A) El experimento de footprinting permite observar la ubicación de la unión de las proteínas al ADN.
Condición 1: Está unida CAP, la RNA polimerasa (junto a Sigma)
Condición 2: Solamente unida LacI
Condición 3: Está unida CAP y LacI
B) El experimento de EMSA muestra que la proteína LacI se une al promotor del operón lac solamente
en las condiciones 2 y 3.
C) Condición 1: Baja glucosa porque está unida CAP, alta lactosa porque LacI no está unidad y está
unida la RNA polimerasa (junto a Sigma) Resultado del EMSA 1 2 3 Sólo sonda Sonda + LacI Sonda
para el Footprinting y EMSA -60 -35 -10 +1 Operador Sitio Unión CAP -10 -35 +1 -60 Control 1 2 3 -
60 -35 -10 +1 Sitio Unión CAP Resultado del Footprinting
Condición 2: Alta glucosa porque no está unida CAP y baja lactosa porque solamente unida LacI
Condición 3: Baja glucosa porque está unida CAP y baja lactosa porque LacI está unida al operador.
Descargado por Leonidas Larrondo Cazanga (leonyomg17@gmail.com)
lOMoARcPSD|27237116
Pregunta 2
Sobre la regulación del operon triptófano. Usted analiza el RNA mensajero del operon en una
bacteria mutante y se da cuenta que tanto en presencia como ausencia de trp, se expresa un único
RNA mensajero, de gran tamaño y correspondiente a todos los genes del operon. ¿Qué tipo
mutación(es) explicaría el comportamiento de la bacteria con el operon mutado en presencia y
ausencia de trp? Fundamente su respuesta.
La detección del RNA mensajero del operon completo tanto en presencia como en ausencia de triptófano
da cuenta de que la regulación por atenuación no está ocurriendo. En condiciones de alto triptófano, la
regulación por atenuación produce un RNA pequeño correspondiente a la región líder (región 5´no
traducida) debido a la formación de una horquilla de termino intrínseco de la transcripción entre las
regiones 3 y 4 de la región lider. El hecho de que no ocurra este proceso puede explicarse por una mutación
en la región 3 o 4 de la región líder ya que en su ausencia no podrá formarse la horquilla de terminación
Descargado por Leonidas Larrondo Cazanga (leonyomg17@gmail.com)
lOMoARcPSD|27237116
Pregunta 3
Responda las siguientes preguntas sobre la transcripción génica en procariontes. Fundamente su
respuesta.
A) ¿Qué función cumplen las proteínas Sigma? ¿Con que regiones de la secuencia de un
promotor bacteriano interaccionan?
B) ¿En qué consiste el proceso de isomerización? ¿Qué moléculas participan de este proceso?
C) En un gen que posee un de término de la transcripción dependiente de factores
Brevemente explique ¿Qué ocurriría si usted elimina la secuencia rut en el ADN?
A) Las proteínas Sigma permiten posicionar a la RNA polimerasa en un promotor (reconocimiento del
promotor), son las que hacen el primer contacto con las cajas -10 y -35 del promotor bacteriano y
luego posicionan a la RNA polimerasa para el comienzo de la transcripción. La región sigma 2
interactua con la caja -10 y la región sigma 4 con la caja -35
B) El proceso de isomerización corresponde a la apertura del ADN de doble hebra en la región -10 (rica
en AT) por la región sigma 2 del factor Sigma.
C) Si se elimina la secuencia rut entonces la proteína Rho no podrá unirse al RNA mensajero,
posicionarse y liberar el transcrito en el termino de la transcripción. Por lo que la transcripción no
podrá terminar
Descargado por Leonidas Larrondo Cazanga (leonyomg17@gmail.com)
lOMoARcPSD|27237116
Pregunta 4
En el siguiente panel se observa una molécula de DNA que contiene el operón Lac. Molécula que
además tiene una marca radioactiva en uno de sus extremos para poder visualizar un experimento
do footprinting y EMSA
Dibuje los genes de footprinting y EMSA de las 4 condiciones (carriles) mencionadas más abajo y
para condición mencione las proteínas que se unirán a la sonda
Carril 1: Bacteria crecida en ausencia de glucosa y presencia de lactosa
Carril 2: Bacteria crecida en presencia de glucosa y ausencia de lactosa
Carril 3: Bacteria del carril 1 pero mutante en la región operadora (no posee operador)
Carril 4: Bactreia del carril 1 pero mutante en CAP (no produce CAP)
Carril 1: en ausencia de glucosa los niveles de cAMP estarían altos por lo tanto CAP estarían unidos
al promotor. En presencia de lactosa (inductor) este inactivaría a Lacl. Por lo tanto, Lacl no estaría
unido al promotor. La RNA poli también estaría unida
Carril 2: en presencia de glucosa los niveles de cAMP serian bajos por lo tanto CAP no estaría unido.
En ausencia de lactosa Lacl estaría unido al operador
Carril 3: en ausencia de glucosa los niveles de cAMP estarían altos por lo tanto CAP estaría unido
al promotor. En presencia de lactosa (inductor) este inactivaría a Lacl. Por lo tanto, Lacl no estaría
unido al promotor. La RNA poli también estaría unida. Como no hay región operadora Lacl no
podría unirse, pero en esta condición tampoco lo haría. El patrón es idéntico al carril 1.
Carril 4: en ausencia de glucosa, los niveles de cAMP estarían altos por lo tanto CAP podría unirse
al promotor, sin embargo, la mutante no posee CAP, por lo que no estaría unido al promotor. En
presencia de lactosa este inactivaría a Lacl. Por lo tanto, no estaría unido al promotor. La RNA poli
de todas formas podría unirse.
Descargado por Leonidas Larrondo Cazanga (leonyomg17@gmail.com)
lOMoARcPSD|27237116
Pregunta 5
Usted se encuentra estudiando los procesos de transcripción y regulación del operón triptófano
(trp) en una cepa de E. coli que adquirió mutaciones que usted quisiera estudiar.
A) Usted ha identificado que en la bacteria mutante, la RNA polimerasa tiene problemas para
identificar la región -35 de una gran cantidad de regiones promotoras ¿Qué tipo de
mutación explicaría dicho comportamiento? Fundamente su respuesta
B) Al estudiar el término de la transcripción en dicha bacteria mutante, usted se da cuenta
que solo en aquellos operones que poseen una secuencia rut, la RNA polimerasa no finaliza
la transcripción donde debería hacerlo, extendiendo el RNA mensajeo más allá de que
debería ¿Qué tipo de mutación explicaría dicho comportamiento? Fundamente su
respuesta.
C) Finalmente, en el operón triptófano usted detecta una deleción de la región 1 que forma
parte del UTR del mRNA del operón ¿Qué esperaría que ocurriera con la regulación de la
expresión de los genes del operón (en condiciones de presencia y ausencia de trp) en esta
mutante? Fundamente su respuesta
A) Una mutación en el dominio sigma 4 del factor sigma explicaría este comportamiento. El
factor sigma es el encargado de digerir a la RNA poli a la región promotora de los genes, en
este contexto es el factor sigma el que hace el primer contacto con el DNA de la región
promotora y el dominio sigma 4 es que hace contacto con la región -35.
B) Una mutación que inactive la proteína Rho podría explicar dicho comportamiento. En
condiciones normales la proteína rho se uniria a la secuencia rut en el mRNA y esto le
permitiría viajar por el mRNA hasta encontrar a la RNA poli para luego terminar la
transcripción gracias a su actividad DNA:RNA helicasa, lo que libera el mRNA de RNA poli
C) La eliminación de la región 1 provocaría la perdida de los codones de trp que están
codificados en esta región. En condiciones de ausencia de trp, el ribosoma no hará una
pausa en la región líder y se favorecerá la formación de horquillas de terminación de la
transcripción cuando en situaciones normales haría una pausa y se formaría la horquilla de
antiterminacion. De todas formas, la regulación mediada por la unión de TrpR al operador
se mantiene inalterada
Descargado por Leonidas Larrondo Cazanga (leonyomg17@gmail.com)
lOMoARcPSD|27237116
Pregunta 6
Usted se encuentra estudiando el fenómeno de splicing en levaduras. Y para ello utiliza dos cepas,
una normal (WT) y otra mutante Δslt22-1, la cual tiene una deleción en el gen de una proteína que
parece tener rol en el splicing en levaduras. Genera de cada cepa, un extracto proteico y con U-
RNAs, competente en generar splicing in vitro. Incuba cada extracto con su sustrato sintético
marcado radiactivamente en todos sus nucleótidos y, después de 10 min, analiza el resultado en
un gel de poliacrilamida. En el carril WT, se observa el mRNA maduro generado, como los
intermediarios del splicing, (rectángulo negro: exón 1; rectángulo blanco: exón 2; línea negra:
intrón). En el carril Δslt22-1, observa el resultado del splicing obtenido a partir del extracto de la
cepa mutante. De su conocimiento del proceso de splicing:
A) En la mutante, ¿Cuál sería el paso principal del proceso de splicing que se ve afectado en la
cepa mutante? Explique
B) ¿basado en este resultado, que rol asignaría usted, en el splicinga la proteína Stl22-1?
Explique
A) En el ensayo de slicing de la cepa WT, se observa la
generación del mRNA maduro, la generación del
intermediario, esto es el producto generado cuando la
Adenina del punto de ramificación realiza el ataque
nucleofílico al límite exón 1/ intrón; además se observa el
lazo, lo que indica que el intermediario, sirve de sustrato
para continuar y finalizar con el splicing. En la cepa
mutante para Stl22-1 se observa que no existe producto
final de splicing. No se observa generaciones del
intermediario, lo que indica que el paso previo, es decir,
el ataque nucleofílico de la A del punto de ramificación es
deficiente o ausente. Por lo cual, el paso afectado en la
mutación, pueden ser dos: el reconocimiento de la A del
sitio de ramificación o el posterior ataque nucleofílico de
la A al límite exon1/intrón
C) Stl22-1 tendría rol similar a los factores que realizan el reconocimiento inicial de la Adenina
del punto de ramificación o ser un factor importante en el rol de U2 al inicio del splicing,
cuando este ultimo interacciona con U6/U5 para el paso final del ataque nucleofílico del
límite exon1/intrón. Por lo cual, al estar mutado, este paso se ve severamente afectado y
no produce el intermediario
Descargado por Leonidas Larrondo Cazanga (leonyomg17@gmail.com)
lOMoARcPSD|27237116
Pregunta 7
Las siguientes sentencias, ¿son verdaderas o falsas? Al elegir una opción debe explicar el porque
de la misma
A) En el proceso de maduración del mRNA, en ausencia de la adenina del punto de
ramificación, el intermediario ¨lariat¨es formado, pero no liberado del transcrito inmaduro.
B) En el procesamiento del extremo 3´del mRNA, la secuencia AAUAAA, la última A (en
negrita), corresponde a la adenina que sirve como punto de partida para agregar la cola de
poli A.
C) TFIIA es un factor de transcripción requerido para ¨llevar¨ la RNA polimerasa II hacia el
promotor.
D) Los enhancers son proteínas regulatorias que controlan a distancia la expresión de genes
transcritos por a RNA poli II
A) FALSO: al no haber A en el punto de ramificación, no habrá conocimiento por U2, no se
formará el complejo terciario U2-U5-U6 y no se realizará el ataque nucleofílico al límite
exón 5´- intrón.
B) FALSO: la secuencia indicada es denominada Secuencia de poliadenilación, la cual es
reconocida por un complejo proteico, que permite iniciar entre 10 a 30 nucleótidos, rio
abajo de la última A, un corte endonucleolitico y posterior adición de la cola poli A, por la
PAP
C) FALSO: TFIIA se une a continuación de TFIID y su rol es estabilizar a TBP en su unión a la
caja TATA estabilizando así, el reconocimiento de este segmento del promotor
D) FALSO: los enhancer son secuencia de DNA que controlan a distancia la expresión de genes,
pues son reconocidos por factores proteicos que se unen a estas secuencias y, activan la
transcripción, pues curvan el DNA y se conectan al PIC del gen que controlan a través del
mediador
Descargado por Leonidas Larrondo Cazanga (leonyomg17@gmail.com)
lOMoARcPSD|27237116
Pregunta 8
El siguiente gel muestra el resultado de un ensayo con DNasa I usando como sustrato una
secuencia de DNA que contiene la región promotora y parte de la secuencia transcrita del gen
SCP de levadura. Se identifican regiones claves a analizar: caja TATA, iniciador- Inr: MTE y DPE.
El ensayo se llevo a cabo uniendo solo TFIIA (IIA) al DNA, solo TFIID (IID) o ambos. El primer y
ultimo carril corresponde al DNA control son proteínas y digeridas con DNasa I, como control.
Del análisis e interpretación del resultado experimental:
A) ¿Cuál de los dos factores, TFIID o TFIIA, se une primero al DNA para permitir la unión del
otro factor? Explique
B) ¿De este resultado, cual factor(es) reconoce la caja TATA? Explique
C) Indique que región clave analizada, reconoce exclusivamente TFIIA
A) Se observa que al utilizar TFIIA por si solo, no hay protección del
DNA: contrario al caso de TFIID por si solo, donde si hay
protección en Inr, MTE y DPE. Y al ver el patrón de protección
utilizando ambas proteínas se ve el mismo patrón de protección
de TFIID, cuando esta solo. Luego, el que se une al DNA es
primero TFIID
B) El factor que reconoce la caja TATA es la recombinación de
TFIID/A. pues ninguno de los dos por si solo protege esta región
C) TFIIA por si solo, no reconoce ni se une a ninguna de las regiones
estudiadas
Descargado por Leonidas Larrondo Cazanga (leonyomg17@gmail.com)
lOMoARcPSD|27237116
Pregunta 9
El siguiente gel muestra un ensayo in vitro del procesamiento de un pri-miRNA (probe), usando
como proteína el complejo Drocha-DGCR8 (Flag-DGCR8) a dos diferentes concentraciones. De la
interpretación del gel:
A) ¿hay procesamiento del pri-miRNA por parte del complejo micro-procesador?
B) ¿el producto observado corresponde al microRNA maduro? Explique
A) Al ver los resultados del procesamiento in vitro, se observa
generaciones de producto de 63nt, lo cual no se observa en
el control (probe). Por lo cual la respuesta es SI.
B) No, el producto no corresponde a miRNA maduro; por el
tamaño es el pre.miRNA.
miRNA maduro tienen tamaño de 21nt en promedio
Descargado por Leonidas Larrondo Cazanga (leonyomg17@gmail.com)
lOMoARcPSD|27237116
Pregunta 10
En el siguiente esquema del ensamblaje jerárquico de los factores de transcripción de la RNA
poli II para generar el complejo de preiniciacion (PIC) en el promotor.
Asigne a cada numero a que factor de transcripción corresponde. Siendo 1, el primer factor
que se une al promotor y 6 el ultimo
1.- TFIID
2.- TFIIA
3.- TFIIB
4.- TFIIF – Poli II
5.- TFIIE
6.- TFIIH
Descargado por Leonidas Larrondo Cazanga (leonyomg17@gmail.com)
lOMoARcPSD|27237116
Pregunta 11
En el protocolo de CHiP, inmunoprecipitación de cromatina, para determinar las
modificaciones de las histonas y relacionarlas con la actividad transcripcional de un gen
determinado
A) ¿Cuál es el propósito del paso en que las células vivas se tratan con agentes fijadores?
B) ¿Cómo se detectan, por ejemplo, una modificación como la trimetilacion de lisina 9 de la
histona H3?
A) El propósito es entrecruzar (fijar) in vivo las proteínas que están unidas al DNA en el
momento del ensayo y, así poder posteriormente aislar complejo DNA/proteínas para si
posterior estudio
B) Para este propósito, una vez tratadas las células con el agente fijador, estas se lisan, se
fragmenta el DNA y se realiza la inmunoprecipitación utilizando un anticuerpo especifico
que reconoce la trimetilacion indicada. Se realizan lavados para eliminar lo inespecífico,
quedando de esa manera lo reconocido por el anticuerpo de interés.
Descargado por Leonidas Larrondo Cazanga (leonyomg17@gmail.com)
lOMoARcPSD|27237116
Pregunta 12
Usted se encuentra estudiando la vía de síntesis de los miRNAs miR240 y miR241, en dos tipos
de células de mamíferos (células A y células B). Mediante ingeniería genética realiza un knock-
out del gen de Drosha y procede a analizar el efecto de la ausencia de Drosha en las vías
canónicas de síntesis de los miRNAs indicados. En la célula A, encuentra que no se produce en
absoluto los miRNAs indicados. En cambio, en las células B, aun cuando los niveles de los miRNAs
240 y 241 son 50% menos que los producidos en las células control, estos si se sintetizan.
A.- ¿Cuál es el rol que cumple Drosha en la vía canónica de síntesis de los miRNAs?
B.- ¿Por qué considera usted que a pesar que Drosha esta ausente en las células B, igual se
producen ambos miRNAs? Y no sucede lo mismo en las células A.
A.- Drosha es una proteína que pertenece al complejo de microprocesadores, compuestos
además por las proteínas DGRB8. El rol de Drosha es generar el pre-miRNA al cortar la base del
tallo del pri-miRNA.
B.- Al eliminar Drosha, la vía canónica de síntesis del miRNA debiese ser completamente abolida.
Sin embargo, a pesar de esto el hecho de generarse los miRNAs maduros, implica
necesariamente que debe haber una vía alternativa de biosíntesis de miRNAs
Descargado por Leonidas Larrondo Cazanga (leonyomg17@gmail.com)
lOMoARcPSD|27237116
Pregunta 13
Responda las siguientes preguntas sobre transcripción y regulación de la expresión génica en
procarionte. Fundamente su respuesta.
A) En un promotor bacteriano ¿Qué propiedades fisicoquímicas pose la región -10 que
permite la iniciación de la transcripción? Nombre la proteína y su dominio que se une a
esta región-
B) En un promotor bacteriano ¿Qué función cumple la región extendida -10 del promotor?
Nombre la proteína y su dominio que se une a esta región
C) Brevemente explique el papel que cumple la región rut en la terminación dependiente
de factores
A.- La región -10 (caja TATA o caja pribnow) es una región rica en AT, lo que se traduce en que
resulta más fácil para el demonio 2 del factor sigma abrir la doble hélice, ya que una región rica
en AT tiene menos puentes de hidrógenos.
B.- La región -10 extendida (TGN) ocurre en algunos promotores como mecanismo de regulación
adicional a la región -10 tradicional. A diferencia de la región -10 donde se une el dominio 2 del
factor sigma, en la región -10 extendida se une el dominio 3 del factor sigma.
C.- La región rut es una región que se encuentra el mRNA y que permite la unión de la proteína
Rho. La proteína Rho es una ATPasa dependiente de RNA y RNA-DNA helicasa que una vez que
se una a la secuencia rut recorre el mRNA hasta llegar al complejo de transcripción,
desestabilizándolo el hibrido de RNA-DNA llevando a cabo la transcripción.
Descargado por Leonidas Larrondo Cazanga (leonyomg17@gmail.com)
lOMoARcPSD|27237116
Pregunta 14
Las siguientes sentencias, ¿son verdaderas o falsas? Al elegir una opción debe explicar el porqué
de la misma.
A) En el proceso de maduración del mRNA, en ausencia de U2, el intercambio <lariet= es
formado, pero no liberado del transcripto inmaduro.
B) La adenina (A) del punto de ramificación, es reconocida por U6, lo que provoca ataque
nucleofílico a la unión exón/intrón 5´.
C) En el procesamiento del extremo 3´del mRNA, la secuencia AAUAAA, la última A (en
negrita), corresponde a la adenina que sirve como punto de partida para agregar la cola
de poli A.
D) Los eventos de maduración del pre-mRNA: caping, eliminación de intrones y adición de
la cola de poli A, son co-transcripcionales y no depende del CTD de la RNA pol II.
E) Para realizar el clereance del promotor, durante el inicio de la transcripción en
eucariotas, es necesario TFIIH.
A.- FALSO: U2 es necesario para reconocer la A del punto de ramificación. Posterior a esta unión
se produce el ataque nucleofílico de la A, al limite 5´exon/intrón, generando el intermediario
lariat.
B.- FALSO: La adenina del punto de ramificación es reconocida por U2, el cual permite generar
el primer ataque nucleofílico al limite 5´exon/intrón, seguido de la liberación del intrón como el
intermediario lariat.
C.- FALSO: La secuencia indicada corresponde a la secuencia consenso denominada señal de
poliadenilación. Esta secuencia es reconocida por un complejo proteica, que posterior a la unión
genera corte del mRNA entre 10 a 30 nucleótido rio debajo de esta secuencia.
D.- FALSO: Si bien es cierto, los eventos indicados, son co-transcripcionales, depende
absolutamente del CTD de la RNA polimerasa II. El CTD sufre fosforilaciones que dependiendo
de ellas, permite atraer los diferentes factores responsables de la maduración del mRNA.
E.- VERDADERO: TFIIH es responsable de fosforilar el CTD de la RNA polimerasa. Lo que provoca
la baja de la afinidad del mediador por la enzima y, además de activar la función helicasa de este
factor, lo que facilita el proceso del clearence del promotor.
Descargado por Leonidas Larrondo Cazanga (leonyomg17@gmail.com)
lOMoARcPSD|27237116
Pregunta 15
Un grupo de investigadores quiere determinar los cabios de expresión génica global (a genoma
completo) en un hongo tiene aproximadamente 10.000 genes. Este hongo percibe luz, y su genoma
presenta 5 genes que codifican para foto-receptores (que son proteínas perciben luz y gatillan
cambios de expresión génica). Para analizar los cambios de expresión génica, los investigadores
realizan un experimento de micro array, utilizando dos cepas del hongo: una silvestre (que se
muestra como WT en la figura) y una cepa mutante (denominada MUT en la figura). Proceden a
analizar la expresión solo para uno de los genes que codifican para foto-receptores. Amabas cepas
son sometidas a dos condiciones experimentales: crecimiento en oscuridad (indicado como DD en
la figura) durante 3 días, y crecimiento en oscuridad durante 3 días seguidos de un pulso de luz de
60 min (indicado como LP en la figura; Light Pulse). Para representar los niveles de expresión de
todos los genes (representados como líneas horizontales en la figura), los investigadores realizaron
un análisis de cluster, y los resultados se presentan como <heat map=; el que se muestra a
continuación. Los niveles de expresión se muestran en la escala de colores indicados a la derecha:
desde niveles <-2=, hasta niveles <+2=.
A) ¿De acuerdo con el heat map, considera (si
o no) que la cepa mutante generada en el laboratorio es <ciego= a
la luz? Explique su respuesta.
B) Considerando solo la cepa WT, (DD v/s LP), indique si el
numero de genes que muestran una fuerte inducción por luz es
mayor o menor al numero de genes que muestra una fuerte
represión. Explique su respuesta. (puede usar como referencia
distintas zonas de la figura para ayudar en su justificación)
A.- No se puede considerar ciega. Si fuera ciega, no vería luz. Y el cluster (o representación
gráfica) muestra claramente que la mutante percibe luz pues se induce genes y se reprime genes
en repuestas a la luz (compara grupo MUT DD v/s MUT LP). Se aprecia una evidente respuesta a
la luz.
B.- Por otro lado, el ensayo considero el análisis de un solo gen y en total son 5, luego quedan
otros foto-receptores. En consecuencia, la mutante tiene todavía 4 foto-receptores (lo que se
explica en el enunciado) y daría cuenta además del cambio de expresión génica ante la
exposición a la luz. Fuerte inducción seria de verde a rojo. Una fuerte represión seria lo contrario.
Al mirar la figura. Se puede observar que los bloques (o clusters) son verde a rojo son
mayoritarios a los cluster que muestran represión por luz.
Descargado por Leonidas Larrondo Cazanga (leonyomg17@gmail.com)
lOMoARcPSD|27237116
Pregunta 16
En el siguiente gel se muestra el resultado de un ensayo de splicing in vitro en levadura. WT,
muestra la cinética de splicing del pre-mRNA entre los 5 y 20 min en el modelo normal. El exón
1 es representado por el rectángulo negro, el intrón por la línea negra y el exón 2, por el
rectángulo blanco. El lazo y el lazo-exón 2 también son indicados. El mRNA maduro, se indica en
el fondo del gel. Al repetir el experimento, con el mismo pre-mRNA, pero esta vez en una cepa
de levadura, con una mutación en la proteína stl22-1, bajo las mismas condiciones que para WT,
se observa un efecto importante, pero no total sobre el proceso de splicing.
A) Según este resultado y su conocimiento del proceso secuencial de splicing ¿Qué paso(s)
del splicing y que snRNAs estaría afectando la ausencia de esta proteína?
B) ¿A qué cree usted que se debe que los intermediarios, de lazo-exón 2 y lazo, migren más
arriba que el pre-mRNA? Explique
A.- Al comparar el tiempo 5 min en el modelo WT v/s slt22-1, se observa que hay disminución
en los intermediarios tipo lazo y lazo-exón 2 en la mutante. Esto sugiere que el mutante tendría
mutaciones en U2, snRNA que da inicio al proceso de splicing reconociendo la A del punto de
ramificación y permite el posterior ataque nucleofílico al límite exón 1 intrón. Y al encontrarse
disminuido el intermediario lazo-exón 2, sugiere que también hay deficiencia en U4/U6 que son
los responsables de favorecer el corte del lazo-exón 2 provocando la liberación del exón
Descargado por Leonidas Larrondo Cazanga (leonyomg17@gmail.com)
lOMoARcPSD|27237116
Pregunta 17
En la figura de la izquierda, se encuentra el resultado de un ensayo de poliadenilación in vitro,
utilizando un RNA sintético y donde la adición de la cola de poliA, se indica por (A)n.
Los RNAs usados contienen el extremo 3’ procesado (carril 1 y 2) o, corresponde al precursor no
procesado (carril 3 y 4). Los respectivos RNAs fueron incubados por 35 min sin ATP (carril 1 y 3)
o con 1nM ATP (carril 2 y 4) en presencia de la enzima poli-A-polimerasa (PAP). Terminado el
ensayo, se aislaron los respectivos productos de RNAs y se analizaron en geles denaturantes. Se
muestra una representación esquemática de los sustratos usados en el ensayo.
A) Para el primer experimento, explique por qué no hay poliadenilación en 2 y si en 4
En el gel de la derecha, se muestra el resultado de la poliadenilación in vitro de diferentes
versiones del mismo RNA, a los cuales se le ha adicionado al extremo 3´varios nucleótidos, desde
2 citidinas hasta 6 citidinas. La parte superior indica los diferentes tiempos de incubación en
presencia de la PAP. Como control, se usó como sustrato el mismo RNA del primer experimento
(gel izquierdo, carril 1 y 2)
B) A la luz de estos resultados, explique porque estos RNAs con las colas crecientes de
citidinas si son más eficientemente poliadenilados.
A.- Al observar la estructura del sustrato de RNA, en 2 se observa que el extremo 3´está en un
asa, en cambio en 4 el extremo 3´esta lejos del asa. Luego, este hecho sugiere que la
accesibilidad al extremo 3’ por parte de la enzima es requisito para una eficiente poliadenilación.
B.- Al alejar el extremo 3´de la base del asa, observamos que mientras más lejos este (por la
adición de las distintas Cs), observamos que la eficiencia de poliadenilación aumenta (comparar
2 Cs v/s 6 Cs). Luego es requisito para la PAP tener acceso a un extremo 3´OH que no forme parte
de una estructura secundaria para poder realizar su función
Descargado por Leonidas Larrondo Cazanga (leonyomg17@gmail.com)
lOMoARcPSD|27237116
Pregunta 18
La figura muestra un ensayo de protección por DNAsa de la región promotora y parte del inicio
del gen de Hsp70 humano, realizado en presencia del factor de transcripción TFIID. Se usaron
concentraciones crecientes de TFIID, desde 63ng (carril 2), 130ng (carril 3), 250ng (carril 4) y
500ng (carril 5) de la proteína. Carril 1, corresponde al ensayo de DNAsa en ausencia de TFIID.
G/A, estándar de DNA digerido. Se muestra la protección en ambas hebras del DNA, indicadas
en la parte superior del gel. Con este resultado.
A) Indique, ¿cuál(les) son las regiones del promotor protegido por TFIID?
B) Las regiones del gen de Hsp70 que son transcritas, ¿son también protegidas por este
factor? Explique
C) Finalmente, al comparar el patrón de protección de ambas hebras, se observan
diferencias evidentes. ¿Por qué, si es una única región de DNA de doble hebra la que es
protegida por TFIID, el patrón de protección es ligeramente diferentes en ambas hebras?
A.- Las regiones promotoras protegidas por este factor corresponde a las zonas comprendidas
entre – 6 a aprox. -45 en la hebra no transcrita y -2 a -10 aprox. más -15 a -45 en la hebra que se
transcribe.
B.- Si, las regiones que se transcriben del gen van del nucleótido +1 al +35 en el gel, y se puede
ver claramente que hay zomas protegidas en la hebra que se transcribe, sobre todo en la región
cercana al +30 de esa hebra de DNA.
C.- La diferencia se debe a que el DNA es una molécula tridimensional de doble hélice, por lo
cual, al unírsele una proteína, se unirá aparte de la hélice y sin poder interaccionar con la zona
de la doble hélice que está en el lado opuesto de la misma.
Descargado por Leonidas Larrondo Cazanga (leonyomg17@gmail.com)
lOMoARcPSD|27237116
Pregunta 19
En referencia a las técnicas usadas en transcriptómica, como microarreglos y RNAseq, explique
A) Tres características que deben tener los oligonucleótidos usados en microarreglos.
B) ¿Cuál es el propósito de agregar adaptadores al RNA en los extremos 5´y 3´en el
protocolo de RNAseq?
C) ¿Cuál es el propósito de fragmentar el RNA total en el protocolo de RNAseq, previo a la
síntesis de cDNA?
A.- De al menos 20-25 base de longitud, óptimo de 45-6º bases de largo
Sin sobrelapamiento entre ellos
Composición de bases similares, esto asegura Tm similares
10 a 20 oligos diferentes por cada gen
Únicos en el genoma
B.- EL uso de adaptadores en ambos extremos del RNA proporciona una situación de
homogenización de la población de RNAs, en la cual todos ellos, independiente del largo y de la
cantidad poseen los mismos extremos. Esto asegura que, en el siguiente paso de amplificación,
la totalidad de los cDNAs presentes sean sintetizados de la misma manera
C.- El propósito de la fragmentación del RNA es obtener una población de moléculas de RNA de
tamaño similar (aprox. 200b) que será usado de molde para la síntesis del cDNA. Al ser una
población homogénea en tamaño, se favorece que la síntesis tenga la misma eficiencia para
cualquier secuencia presente en la población
Descargado por Leonidas Larrondo Cazanga (leonyomg17@gmail.com)
lOMoARcPSD|27237116
Pregunta 20
Mediante técnicas de biología moleculares variadas usted esta estudiando el proceso de
transcripción y maduración del mRNA en varias cepas de levadura
A) En una de ellas encuentra que los mensajeros tienen sus intrones eliminados y presentan
cola poliA, sin embargo, carecen de CAP.
B) En otra cepa, encuentra que el proceso de inicio de la transcripción es inespecífico, pues
observa transcripto que no se inician en el nucleotido +1 del gen sino mas bien de manera
promotor independiente
C) En otra cepa, encuentra que el complejo de pre.iniciacion (PIC), se encuentra fijo en el
promotor sin poder dar inicio al proceso de transcripción
Ante estas observaciones y para cada una de ellas, explique cual seria la proteína deficiente que
daría cuenta de los hechos observados. Sea especifico en explicar la función que lleva a cabo la
proteína deficiente para dar cuenta de lo observado experimentalmente.
A.- En este primer caso existe dos posibilidades, la primera implica que el patrón de fosforilación
del CTD de la RNA polimerasa 2 no es acorde con el proceso de adición del CAP. Pues se debe
recordar que dependiendo del paso del proceso de transcripción, se tiene un patrón de
fosforilación del CDT particular. La otra posibilidad es que exista una deficiencia en la actividad
enzimática de la enzima que posibilita la adición del CAP-0. Enzima que corresponde a la que
elimina el grupo fosfato del extremo 5´del mRNA, esto es la RNAsa trifosfatasa. Pues el producto
de esta reacción sirve de sustrato para la adición de guanina
B.- En el caso de la observación de que una de las cepas inicia la transcripción de manera
inespecífica y no desde el promotor, el problema principal se debe a que existiría una mutación
que afecta la función del factor de inicio TFIIF, pues este facto es responsable de unir a la RNA
pol II y reclutar específicamente hacia al promotor, cumpliendo una función análoga al factor
sigma de la RNA polimerasa bacteriana, esto es disminuir su afinidad por cualquier secuencia de
DNA.
C.- En el caso de la observación que el PIC permanece inmóvil sin hacer el clereance del
promotor, implica que el CTD no ha sido fosforilado por TFIIH, implicando una mutación en este
factor de transcripción, pues cuando esto ocurre (fosforilación del CTD), la afinidad del mediador
se pierde y se da inicio a la transcripción
Descargado por Leonidas Larrondo Cazanga (leonyomg17@gmail.com)
lOMoARcPSD|27237116
Pregunta 21
Durante el proceso de la transcripción, un gen puede estar codificado en cualquier de las dos
hebras del DNA. Al respecto, cuando la RNA polimerasa transcribe un gen
A) ¿la secuencia correspondiente al promotor del gen, es transcrita? Explique brevemente
su respuesta.
B) ¿Cuándo la RNA polimerasa transcribe la hebra molde, en que sentido se mueve sobre
esta y que otro nombre recibe esta hebra del DNA?
C) La secuencia del mRNA recién sintetizado es idéntica a ¿Cuál hebra del DNA y qué sentido
tiene el transcrito recién sintetizado?
A.- No, la secuencia de los promotores corresponde a regiones del DNA que son reconocidas por
factores de transcripción y que permite que la RNA pol II pueda iniciar la transcripción desde el
nucleosoma +1
B.- Sobre la hebra molde, la RNA polimerasa se mueve en sentido 3´a 5´, lo que le permite
sintetizar el mRNA en dirección 5´a 3´. El otor nombre con que se le conoce a esta hebra es:
hebra no codificante (hebra templada)
C.- La secuencia del mRNA recién sintetizado es idéntica en secuencia a la hebra codificante del
DNA, excepto que en vez de timinina posee uracilo. El sentido del mRNA recién sintetizado es
5´a 3´
Descargado por Leonidas Larrondo Cazanga (leonyomg17@gmail.com)
lOMoARcPSD|27237116
Pregunta 22
Usted se encuentra estudiando la expresión del operón trp en dos bacterias (1 y 2). Una de ellas
posee un open trp normal y la otra adquirió mutaciones que usted quisiera estudiar. Para esto
creció ambas bacterias en presencia y ausencia de trp, luego extrajo mRNA realizo un
experimento de Northern Blot, utilizando una sonda radioactiva que reconoce el comienzo de la
región líder. De acuerdo al resultado de su experimento responda y fundamente:
A) De la bacteria 1 y 2. ¿cuál bacteria es la que posee un operón normal y cual uno mutado?
B) De las condiciones A y B. ¿Cuál es la condición de alta concentración de trp y cual es la
condición de bajo trp?
C) ¿Qué tipo de mutación(es) explicaría el comportamiento de la bacteria con el operón
mutado en presencia y ausencia de trp?
P: promotor
O: operador
trpL: región lider *
Sonda de DNA marcada
radioactivamente
Bacteria 1 Bacteria 2
A B A B
A.- Bacteria normal: 1 y bacteria con operón mutado:2
B.- Condición de alto trp: A y condición de bajo trp: B
C.- Mutación en los codones de trp en la región 1. Si la mutación cambia estos codones por
cualquier otro entonces el ribosoma no se detendrá, produciéndose la atenuación.
Mutación de la región líder que elimine la región 2. Entonces no se podría formar horquilla 2-3
por lo que se generaría la horquilla de terminación 3-4. Produciéndose la atenuación
Descargado por Leonidas Larrondo Cazanga (leonyomg17@gmail.com)
lOMoARcPSD|27237116
Pregunta 23
Indique y explique al menos tres razones de porque el CTD de la RNA polimerasa II es vital para el
proceso de la transcripción
1) Al inicio de la transcripción el CTD no se encuentra fosforilado, esto permite que el
mediado se una con alta afinidad al CTD, favoreciendo la unión de la RNA pol II al
promotor
2) Es importante para el clereance del promotor. Pues CTD, al ser fosforilado por TFIIH,
genera perdida de afinidad por el mediador y en consecuencia comienza la transcripción.
3) Promueve reclutamiento de factores de splicing y factores de poliadenilación
Pregunta 24
¿Cuál es el rol de la secuencia AAUAAA en el proceso de maduración del mRNA eucariota?
Explique
Esta secuencia sirve como señal para ser reconocida por factores proteicos, principalmente el
complejo CPSF (factor de especificidad de corte y poliadenilación) que posterior a reconocer la
señal de poliadenilación, inducen un corte del mRNA entre 10 a 30 nucleótidos rio debajo de la
misma. Y reclutan posteriormente al complejo enzimático de la poli-A-polimerasa (PAP),
responsable de agregar la cola de poli A al extremo 3’ del mRNA
Descargado por Leonidas Larrondo Cazanga (leonyomg17@gmail.com)
lOMoARcPSD|27237116
Pregunta 25
En referencia a los roles que cumplen las siguientes moléculas en el proceso de la transcripción
y maduración del mRNA eucariota, explique que paso o proceso se vería afectado en caso de
que las siguientes moléculas no fueran funcionales:
TFIID: Reconoce y une la caja TATA del promotor, a través de la subunidad TBP. Además de
unirse al iniciador y los elementos rio abajo del promotor. Por lo tanto, se vería afectado el inicio
de la transcripción
TFIIH: Este factor es requerido para el clearence del promotor, pues posee actividad helicasa y
también es responsable de fosforilar la cola de CTD de la RNA polimerasa. Sin este factor, no se
podrá dar inicio a la transcripción.
TFIIF: Este factor reduce la unión especifica de la RNA pol al DNA. En consecuencia, aumenta la
especificidad de unión de la enzima por el promotor.
Guanilil transferasa: Es una enzima responsable de transferir una guanosina trifosfato al
extremo 5´del mRNA inmaduro-. Generando una unión 5´-5´. Característica del CAP 0
U2 snRNP: Esta ribonucleoproteina reconoce la A del punto de ramificación, presente en los
intrones y favorece el posterior reconocimiento del sitio de splicing 5´
Poli-A-polimerasa: Enzima responsable de adicionar la cola de poli A al extremo 3´de los mRNAs, finalizando
así el proceso de maduración de los mismo.
Descargado por Leonidas Larrondo Cazanga (leonyomg17@gmail.com)
lOMoARcPSD|27237116
Pregunta 26
El inicio de la replicacion en eucariotas es un proceso de multiples pasos donde intrvienen decenas
de proteinas que se aseguran el corecto desempeño de este proceso. Al respecto, indique que
evento en especifico se veria afectado en el caso de que las siguientes proteinas fuesen
defectuasoas en su funcionamiento.
A) Cdt1
B) Geminina
C) MCM 2-7
D) DNA polimerasa alfa
E) GINS
A) Cdt1: proteina responsable de reclutar al primer hexamero de MCM2-7 al origen de
replicacion. En ausencia de esta actividad, no se formara el doble hexamero y en
consecuencia no habra actividad helicasa, responsable de abrir la doble helice del DNA
B) Geminina: tiene como funcion secuenciar a la proteina Cdt1, evitando asi que esta pueda
reclutar a los dos hexameros MCM2-7 y ¨prender¨ el origen de replicacion. En caso de ser
deficiente, el inico de la replicacion a partir de un mismo origen no se veria impedido,
provocando que se vuelva a replicar el DNA a partir del mismo gen
C) MCM2-7: proteina con actividad helicasa, que son cargadas en el origen de replicacion por
Cdt1 en forma de un doble hexamero (DH). Su funcion primaria es dar inicio a la ruptura de
la doblehelice del DNA para iniciar la replicacion. En su ausencia, no se podra iniciar la
replicacion
D) DNA pli alfa: enzima polimerizante que forma un complejo con la primasa y en
consecuencia, es responsable de la sintesis del partidor y de iniciar la replicacion del DNA.
Siendo reemplazada posteriormente por la DNA poli delta o epsilon. En ausencia de esta
actividad, no habra sisntesis del DNA a partir de ninguna de las hebras
E) GINS: GINS junto a Cdc45 se unen al DH formando un complejo con una furerte actividad
helicasa el cual va por delante de la horquilla de replicacion por lo caul es importante para
la progresividad de la sintesis de DNA. Sin esta actividad, la progresion de la replicacion se
veria seriamnete afectada
Descargado por Leonidas Larrondo Cazanga (leonyomg17@gmail.com)
lOMoARcPSD|27237116
Solemne 3
Pregunta 1
Usted a generado distintas mutantes bacterianas que posee distintos problemas en el proceso
del RNA mensajero a proteína. A continuación, se mencionan los fenotipos de algunas de estas
mutantes. Para cada caso mencione que componentes del proceso de traducción en procarionte
podría estar mutado (ausente). Explique y fundamente su respuesta.
A) Mutante 1. Usted se da cuenta de que en esta mutante el proceso de traducción no ha
podido comenzar, pero no encuentra problema ni con los factores de iniciación ni con
las subunidades de ribosomas ni con el mRNA.
B) Mutante 2. Usted se da cuenta de que todos los ribosomas se encuentran detenidos con
un tRNA unidos a un polipéptido en el sitio P. al estudiar los contenidos celulares, de que
todas las proteínas EF-Tu se encuentran unidas solamente a GTP.
C) Mutante 3. Al analizar los ribosomas se da cuenta de que se encuentran detenidos. Hay
un tRNA vacío en el sitio P un tRNA con un polipéptido asociado en el sitio A.
D) Mutante 1. Usted analiza los ribosomas de esta bacteria y se da cuenta de que el peptido
recién formado se encuentra pegado al tRNA en el sitio P del ribosoma, y hay un aminoacil-
tRNA en el sitio A del ribosoma.
E) Mutante 2. Usted examina los ribosomas de esta bacteria y se da cuenta de que no hay
aminoacil-tRNA en el sitio A del ribosoma.
F) Mutante 3. Usted examina los ribosomas de esta bacteria y se da cuenta de que todo el
RNA mensajero está en el citoplasma pero NO unido a la subunidad 30s del ribosoma.
A) Mutante 1: La mutante tiene problemas para generar la molécula 1-Met-tRNAf
B) B.- Mutante 2: La mutante no esta produciendo el factor EF-TS que permite generar
(reciclar) EF-TU-GTP.
C) C.- Mutante 3: Mutaciones en el factor EFG tendría este fenotipo, ya que el ribosoma no
podría llevar a cabo el proceso de transcripción.
D) Si el tRNA del sitio P está cargado con un aminoácido al igual que el tRNA del sitio A y
se encuentra detenido el ribosoma, esto quiere decir que no está ocurriendo la reacción
detranspeptidación (formación del enlace peptídico entre los dos aminoácidos. Esta
reacción ocurre gracias a la subunidad mayor del ribosoma por lo que la mutación
puede estar a este nivel.
E) El hecho que todos los ribosomas tengan un tRNA unido a EF-tu en el sitio A significa
que EF-tu no ha podido liberarse del ribosoma, proceso que solamente ocurre cuando
se hidroliza GTP. Por lo tanto, cualquier mutación que afecte la capacidad de EF-Tu
dehidrolizar el GTP explicaría dicho fenotipo
F) Si la proteína RF1 se encuentra en el sitio A, significa que el factor RRF no ha llegado a
liberar el péptido del último tRNA ni a liberar la proteína RF1. Por lo tanto, una mutación
en RRF explicaría dicho fenotipo.
Descargado por Leonidas Larrondo Cazanga (leonyomg17@gmail.com)
lOMoARcPSD|27237116
Pregunta 2
Sobre las librerías genómicas y de expresión: (12 puntos)
A) Defina y discuta las similitudes y diferencias que existen entre ambas librerías.
B) ¿En qué consiste el análisis de librerías por hibridación? Describa como podría esta técnica
permitirle analizar ambos tipos de genotecas.
A) Una librería genómica incluye el ADN de todo el genoma de una célula u organismo,
mientras que una librería de expresión contiene el ADN que proviene de aquellos
genes que se estaban expresando en un determinado tiempo.
Las librerías genómicas son más complejas y grandes que las librerías de expresión.
Para la construcción de ambas se utilizan vectores tales como plásmidos, cosmidos, fagos,
BAC y YAC. Pero para una librería genómica se necesitan vectores que permitan
clonamiento de regiones grandes mientras que una librería de expresión puede
construirse con vectores de menor tamaño (no BACs ni YACs).
B) El método de análisis basado en hibridación permite identificar el gen o la región de
ADN que uno desea dentro de los cientos de clones de una librería genómica o de
expresión. Para esto se necesita conocer una parte de la secuencia que se busca ya que
se necesita sintetizar in vitro una sonda (producto de PCR u oligonucleótido) el cual es
marcado radioactivamente. Una vez que se ha sintetizado la sonda, se utiliza un papel
filtro para generar una réplica de las colonias bacterianas y se utiliza dicho filtro con las
colonias bacterianas embebidas en él para llevar a cabo una serie de procesos de
desnaturalización y fijación para luego realizar la incubación con la sonda marcada. Esta
sonda sólo se unirá mediante complementariedad de bases a aquellas colonias que
posean el ADN buscado y el papel filtro es finalmente expuesto a un film
autoradiografico para la identificación de aquella colonia en cuyo ADN se unió esta .
Descargado por Leonidas Larrondo Cazanga (leonyomg17@gmail.com)
lOMoARcPSD|27237116
Pregunta 3
Usted desea clonar un fragmento de ADN en el sitio de multiple clonamiento (MCS) del
vector pBIOL240 (ver figura). En la imagen se destacan los sitios de corte únicos como
también los sitios de reconocimiento y corte para cada enzima. De acuerdo con esta
información responda lo siguiente:
A) Nombre 3 estrategias de clonamiento (combinaciones) que permitirían clonar dicho
fragmento de ADN en el vector pBIOL240. Ejemplo: <Cortaría el vector con la enzima
X y el fragmento de ADN con las enzimas Y y Z=.
B) Usted realiza el clonamiento descrito en A. ¿De qué color serían las colonias de un
clonamiento exitoso si usted plaquea la reacción de transformación en una placa
suplementada con Ampicilina y Xgal (sustrato cromogenico de la proteína codificada
por el gen lacZ)? Fundamente su respuesta.
A) Cortar tanto vector como fragmento con KpnI
Cortar con EcoRI y XhoI el fragmento y ligarlo al plasmidio cortado con EcoRI y SalI
Cortar con Ecori y KpnI tanto vector como fragmento.
B) Las colonias serán blancas, esto debido a que el clonamiento en el MCS
interrumpe el marco de lectura abierto del gen lacZ. En un clonamiento no
exitoso, la proteína LacZ (betagalactosidada) se producirá y romperá el sustrato
cromogénico Xgal resultando en una colonia de color azul
Descargado por Leonidas Larrondo Cazanga (leonyomg17@gmail.com)
lOMoARcPSD|27237116
Pregunta 4
Usted recibe un plásmido de 6.3 Kb y se le pide construir un mapa de restricción usando
distintas combinaciones de enzimas de restricción. Usted corre un gel de agarosa al 1%
con cada una de estas reacciones y obtiene un perfil de digestión como el de la figura.
Tomando en cuenta esta información dibuje el mapa de restricción del plásmido.
Descargado por Leonidas Larrondo Cazanga (leonyomg17@gmail.com)
lOMoARcPSD|27237116
Pregunta 5
El siguiente esquema representa una vía de reparación del DNA a propósito de un daño
específico en la doble hebra. Basado en el esquema mostrado, indique cual es la vía de
reparación esquematizada (1p). Y para cada número, indique el nombre de la enzima
involucrada (puede indicar también la actividad enzimática).
1.- DNA glicosidasa
2.- Sitio AP
3.- Endonucleasas
4.- Exonucleasa / Helicasa
5.- DNA polimerasa
6.- DNA ligasa
Descargado por Leonidas Larrondo Cazanga (leonyomg17@gmail.com)
lOMoARcPSD|27237116
Pegunta 6
En el inicio de la traducción en eucariotas, ¿Cuál considera usted que es el paso limitante
del proceso? Es decir, aquel que condiciona todos los pasos que facilitan el
reconocimiento del codón de inicio. Explique, desde un punto de vista molecular el
porqué de su respuesta.
En el inicio de la traducción, el paso limitante del proceso, el cual condiciona todos los
otros pasos posteriores, es el reconocimiento del CAP por eIF4E, el cual al unirse al CAP
permite que eIF4A e IF4G puedan, el primero con su actividad helicasa, desarmar
cualquier estructura secundaría del 5’ UTR y permitir la posterior unión de la subunidad
menor del ribosoma conteniendo el tRNAmet y así iniciar posteriormente el scaning del
mRNA en busca del codón de inicio; mientras eIF4G cumplirá su rol de andamio
molecular que conectará el 5’ del mRNA con la cola de poli A al 3’ mediante su unión a
la proteína PABP, asegurando de esa manera el control de calidad de la integridad del
mRNA y asegurando una eficiente traducción. Todos estos pasos se encuentran
condicionados sí o sí al reconocimiento del CAP por parte del eIF4E.
Pregunta 7
Si en una célula, una mutación tipo mistmatch no es reparada, ¿Cuál será el porcentaje
de las células hijas que contengan la mutación después de la tercera generación de
división celular? Explique su respuesta. (Puede usar esquemas)
Descargado por Leonidas Larrondo Cazanga (leonyomg17@gmail.com)
lOMoARcPSD|27237116
Pregunta 8
Nombrar los componentes
1 2 3
4 8
5 7
6
9
1) Sitio E: exit o salida del tRNA
2) Sitio P: sitio peptidil-RNA
3) Sitio A: sitio aminoacil-tRNA (sitio aceptor
4) Subunidad grande ribosoma 50S
5) Subunidad pequeña ribosoma 30S
6) Sitio de unión del mRNA
7) Loop T
8) Loop D
9) Loop anticodón
Descargado por Leonidas Larrondo Cazanga (leonyomg17@gmail.com)
lOMoARcPSD|27237116
Pregunta 9
De acuerdo con la secuencia de ADN de la hebra codificante de la imagen,
escriba la secuencia de: Tips:
• El ARN mensajero transcrito desde esta secuencia
• Los codones del RNA mensajero +1: inicio de la transcripción
• Los anticodones del tRNA correspondiente ATG: Codon de inicio
• La secuencia completa de aminoácidos del
polipeptido traducido RBS: Sitio unión al ribosoma (Shine Dalgarno)
+1
ACTCTACTAATTAGGGAGGTAAGTTCTATCTATGTACTTTATTGGATAATGATGCCTCCGGTAGGAGGCTTTTTTTTGACTC
RBS Repetidos invertidos
ricos en GC
A) La secuencia va desde el +1 hasta la extensión de poly-U (había que
reconocer el termino intrínseco de la transcripción. Además, la secuencia
es idéntica a la hebra codificante con la diferencia de que en las
posiciones que haya T se cambia por U
ACUAAUUAGGGAGGUAAGUUCUAUCUAUGUACUUUAUUGGAUAAUGAUGCCUCC
GGUAGGAGGCUUUUUUUU.
B) AUG UAC UUU AUU GGA UAA (este último es el codón de stop)
C) UAC AUG AAA UAA CCU
D) Met-Tyr-Phe-Ile-Gly
Descargado por Leonidas Larrondo Cazanga (leonyomg17@gmail.com)
lOMoARcPSD|27237116
Pregunta 10
Defina los siguientes tipos de mutaciones y explique cual es el efecto que puede generar en
la expresión de una proteína.
A) Mutación sinónima
B) Mutación sin sentido
C) Cambio de sentido
A) Mutación sinónima: es aquella donde ocurre un cambio de una base
en un codón, pero el codón alterado sigue codificando el mismo
aminoácido por lo tanto no genera ninguna alteración en la proteína.
B) Mutación sin sentido: es aquella mutación donde ocurre un cambio
de base en el codón que genera la aparición de un codón de termino.
Lo que induce el termino prematuro de la proteína
C) Cambio de sentido: es aquella mutación donde el cambio de base en
el codón puede generar dos situaciones.
- Genera una mutación conservadora, esto es el codón alterado
determina un aminoácido químicamente similar (base por
base)
- Genera una mutación no conservada, esto es el codón alterado
determina un aa químicamente distinto (polar por polar)
Descargado por Leonidas Larrondo Cazanga (leonyomg17@gmail.com)
lOMoARcPSD|27237116
Pregunta 11
En el mecanismo de reparación del DNA denominado Reparación por Escisión de Nucleótido
(NER) en eucariotas ¿Cuál es el rol de las siguientes proteínas?
A) TFIIF
B) XPC/Rad23
a) TFIIF: reconoce el daño en el DNA, el rol de TFIIH es provocar la apertura
de la doble hélice en la zona donde está localizado el daño, para
favorecer la localización de la base dañada
b) XPC/Rad23: esta proteína son las responsables de reconocer la distorsión
generada por el daño al DNA inducido por los dímeros de pirimida y,
permitiendo el posterior reclutamiento de las proteínas encargadas de
repara el DNA
Descargado por Leonidas Larrondo Cazanga (leonyomg17@gmail.com)
lOMoARcPSD|27237116
Pregunta 12
Usted desea clonar el gen yfg desde el fragmento de PCR que se muestra en la figura
(fragmento de color azul), el vector pBIOL240. Tomando en cuenta la información
entregada en la figura responda
A) ¿Qué enzima(s) utilizaría para clonar el gen yfj en el plásmido? Fundamente su
decisión
B) Describa brevemente paso a paso, el proceso de utilizaría para realizar dicho
clonamiento desde la reacción de digestión hasta la transformación, selección y
análisis de clones para confirmar el clonamiento exitoso
A) EcoRI por un lado y por el otro EcoRV o Spel. Aunque el sitio EcoRV sería el preferido al
permitir el uso del gen lacZ para discriminar el correcto clonamiento
B) En primer lugar, habría que digerir tanto el plásmido como el producto de PCR con más
enzimas de restricción correspondientes. Digestiones que podrían se simples o dobles y
que deberían tomar en cuenta la temperatura adecuada para la reacción de digestión.
Posteriormente habría que purificar el DNA digerido (o inactivar las enzimas de
restricción presentes) para posteriormente realizar la reacción de ligación. Finalmente
se realizaría la transformación en una cepa de E. coli de clonamiento ya sea por el
método de electroporación o transformación química, seleccionando la presencia del
plásmido mediante la expresión del gen de resistencia a ampicilina. Si se realiza el
clonamiento en el sitio de múltiples clonamientos (MCS) se pueden escoger la colonia
blanca para el análisis posterior., ya que un clonamiento en dicha región interrumpirá
el gen lac y no se podrá hidrolizar el sustrato cromogenico. La verificación del correcto
clonamiento se puede realizar posteriormente mediante una reacción de PCR utilizando
partidores específicos que flanqueen el MCS, partidores específicos para la región
clonada o una combinación de un partidor del plásmido y una del fragmento clonado.
Visualizando el resultado de esta reacción de PCR en geles de agarosa, teñido con
bromuro de estadio
Descargado por Leonidas Larrondo Cazanga (leonyomg17@gmail.com)
lOMoARcPSD|27237116
Pregunta 13
En una placa de Petri, que contiene un medio que no tiene histamina, siembran 1000
bacterias S. typhimurium auxotrofas de histamina (células mutantes que requieren de
histamina para vivir). Al cabo de dos días usted cuenta con total de 10 colonias.
A) ¿Cómo es posible que haya aparecido colonias en la ausencia de histaminas?
Explique
Repite el experimento, ahora en presencia de 0,4ug de 2-aminoantraceno. Al cabo de dos
días de cultivo, usted cuenta sobre 10000 colonias en placa.
B) ¿Por qué en presencia de este compuesto obtiene un crecimiento de bacterias 1000
veces mayor al del caso anterior? Explique el por que
A) Debido a la alta de duplicación bacteriana, lo mas probable es que haya aparecido
revertantes naturales, que revierten la mutación original que les impedía crecer en
ausencia de histaminas. Y esto explica la aparición de una baja proporción de
colonias en ausencia de aminoácidos
B) El alto numero de colonias aparecidas en presencia del compuesta indicado, sugiere
que este en un mutágeno muy eficiente y, es por eso que la taza de reversión se ve
fuertemente aumentada
Descargado por Leonidas Larrondo Cazanga (leonyomg17@gmail.com)
lOMoARcPSD|27237116
Pregunta 14
Sobre el proceso de traducción
A) Explique que significa que código genético sea: i) redundante (degenerado), ii)
universal y no ambiguo y iii) conservado
B) Utilice la tabla del código genético para dar 1 ejemplo de cada una de las 3
características.
A) Redundancia: algunos aa tienen un solo codón (metionina) otro hasta 6 codones
(leucina)
Universal y no ambiguo: todos los codones (combinaciones) siempre significan algo
Conservado: en general, mutaciones en un nucleótido son toleradas
B) Redundancia: leucina donde CUU, CUC, CUA, CUG codifican para el mismo aa
No ambiguo: todos los aa codifican algo, por ejemplo, UAA, codón de termino
Conservado: se toleran por lo general algunas mutaciones, por ejemplo AAU que
mute a AAC no cambia el aa
Descargado por Leonidas Larrondo Cazanga (leonyomg17@gmail.com)
lOMoARcPSD|27237116
Pregunta 15
Sobre la traducción en procariontes
Usted ha generado distintas mutantes bacterianas que poseen distintos problemas en el
proceso de traducción del RNA mensajero a proteínas. A continuación, se mencionan los
fenotipos de algunas de estas mutantes. Para cada caso mencione que componente del
proceso de traducción en procariontes podría estar mutando (ausente).
Explique y fundamente su respuesta.
A) Mutante 1. Al analizar los ribosomas se da cuenta de que se encuentran detenidos en
los codones de término de sus respectivos mRNA. El sitio P se encuentra ocupado por
el último tRNA de un codon codificante y el sitio A con el codón de termino se encuentra
vacio (4p
Este fenotipo se explicaría por una mutación que impida que los factores RF1-3 o RRF
interactúen con el sitio A para terminar la traducción.
B) Mutante 2. Usted examina los ribosomas de esta bacteria y se da cuenta de que todo
el RNA mensajero está en el citoplasma, pero NO unido a la subunidad 30s del
ribosoma. (3p)
Este fenotipo se explicaría por una mutación en la secuencia anti-shine dalgarno (anti SD)
del 16s ribosomal de la subunidad menor del ribosoma.
C) Mutante 3. Al analizar todas las proteínas EF-Tu se da cuenta que solo están unidas a
GDP (3p).
Este fenotipo se explicaría por una mutación en el factor EF-TS que es el encargado de
reciclar EF-Tu- GDP a su forma con GTP para que pueda volver a ejercer su función
Descargado por Leonidas Larrondo Cazanga (leonyomg17@gmail.com)
lOMoARcPSD|27237116
Pregunta 16
Usted desea clonar el gen biol240 desde el fragmento de ADN doble hebra que se muestra
en la figura, en el vector pBIOL240. Tomando en cuenta la información entregada en la
figura responda: (7 puntos totales)
A) ¿Qué enzimas podría ocupar para clonar el fragmento de ADN en el sitio de múltiple
clonamiento (MCS) del vector? 4p
Las únicas enzimas que sirven para clonar en el MCS son ApaI y EcoRI.
B) Clasifique las 6 enzimas de restricción según el tipo de corte y extremos libres que
generan una vez que cortan sus secuencias de reconocimiento. (0.5 ptos cada uno)
ApaI: corte extremos cohesivo 3´, SmaI, corte romo; NheI, corte cohesivo 5´, SpeI, corte
coesivo 5´; PvuII, corte romo; EcoRI, corte cohesivo 5´.
Descargado por Leonidas Larrondo Cazanga (leonyomg17@gmail.com)
lOMoARcPSD|27237116
Pregunta 17
Estructura función traducción eucarionte
En lo referente a traducción en eucariotas, explique qué paso específico se vería afectado
en el hipotético caso de que los siguientes factores estuviesen ausentes. Explique su
respuesta (2 p c/u)
1) Pabp1; 2) eEF1A; 3) eIF4E; 4) eIF4G; 5) peptidil transferasa
a) Pabp1: Proteína que se une a la cola de poly A y mediante su interacción con eIF4G se
conecta con el CAP, generando el asa cerrada. Luego, en su ausencia, no se podría formar
el asa que conecta el 3’ del mRNA con el CAP.
b) eEF1A: Responsable de llevar el aatRNA al sitio A durante el proceso de la elongación,
en el denominado complejo ternario (TC). Por lo tanto, no habría elongación
c) eIF4E: Une directamente al CAP, sin él no se podría iniciar la búsqueda del codón de inicio
por parte de la subunidad menor del ribosoma según el modelo Kozak, además de no poder
formarse el modelo de asa cerrada, pues eIF4G no podría interaccionar con este factor.
d) eIF4G: Factor de inicio que sirve de andamio, entre otras funciones y, que a través de él
se conecta el extremo 3’ (PABP) y 5’ eIF4E) del mRNA, formando el asa cerrada. Por lo tanto,
no habría traducción
e) peptidil transferasa: Actividad enzimática llevada a cabo por el rRNA 28S, presente en la
subunidad mayor del ribosoma y, que es responsable de generar el enlace peptídico en la
proteína en formación.
Descargado por Leonidas Larrondo Cazanga (leonyomg17@gmail.com)
lOMoARcPSD|27237116
Pregunta 18
Inicio traducción eucariotas
Explique cuál es el rol del CAP en el proceso de traducción en eucariotas. Incluya en su
explicación, por cual factor en específico es reconocido (1p), la formación de qué estructura
de <control de calidad= de la integridad del mRNA se forma (1p) y, que proteínas participan
directamente en esta estructura, indicando el rol de cada una de ellas (5p)
a) El CAP presente al extremo 5’ del mRNA, es reconocida por la sub unidad eIF4E del factor
de iniciación eIF4F, siendo este evento el paso limitante del inicio de la traducción, pues
una vez reconocido el CAP, la subunidad eIF4G, permitirá que la sub unidad menor del
ribosoma, a través de eIF3 se una al CAP. Además, mediada por la misma subunidad, eIF4G,
Pab1p, que reconoce la cola de poli A, permitirá circularizar el mRNA, para asegurar la
calidad del mismo y poder iniciar la traducción.
b) La estructura que se forma es la denominada modelo de asa cerrada, que conecta el
extremo 3’, conteniendo cola de poli A, con el CAP del 5’. Lo que da cuenta de que el mRNA
está maduro.
c) Las proteínas mínimas que participan son:
eIF4E: Reconoce directamente el CAP del mRNA
PABP: Reconoce la cola de poli A en el extremo 3’
eIF4G: Sirve de intermediario entre eIF4E y PABP, para conectar los extremos 5’ y 3’ del
mRNA.
eIF4A: Con su actividad helicasa permite desarmar posibles estructuras secundarias propias
del 5’UTR al inicio de la traducción.
eIF4B: Factor que favorece la actividad de eIF4A y, por lo tanto necesario para potenciar la
actividad helicasa de este último.
Descargado por Leonidas Larrondo Cazanga (leonyomg17@gmail.com)
lOMoARcPSD|27237116
Pregunta 19
Etapas proceso traducción eucariotas
A) La puromicina al ser agregada a una célula que este en pleno proceso de traducción,
provoca que las proteínas que se estén sintetizando sean más cortas con respecto a
aquellas de las células control. Y desde el punto de vista molecular, al efectuar un análisis
fino, encuentra que el péptido naciente se encuentra unido a este antibiótico ¿Por qué cree
usted que este antibiótico genera el termino prematuro de la traducción? (4p) Explique
B) Indique con precisión en que etapas de la traducción (inicio, elongación y termino) son
requeridas las moléculas de ATP y/o GTP. Debe indicar el paso o proceso de cada etapa en
donde son requeridos estas moléculas (NO DEBE EXPLICAR) (6pt)
A) Debido a que el péptido que se está sintetizando es transferido al antibiótico puromicina,
esto implica que la puromicina está imitando a un aatRNA y, por lo tanto ingresa al sitio A,
desplazando a los aatRNAs reales, y en consecuencia, la peptidil transferasa transfiere a la
puromicina la proteína que se está sintetizando.
B) INICIO: Formación complejo ternario (TC):Una molécula GTP
Unión del PIC al mRNA: Una molécula de ATP
Unión sub-U mayor a la sub-U menor:Una molécula GTP
Escaneo para encontrar el codón de inicio: Un ATP¨
Reconocimiento del codón de inicio: Una molécula de ATP
ELONGACION: Unión del aatRNA al sitio A del ribosoma por eEF1A: Un GTP
Reciclaje de eEF1A por eEF1B: Un GTP.
Translocación del ribosoma por eEF2: Un GTP
TERMINO: Unión de eRF3 al sitio A: Un GTP
Descargado por Leonidas Larrondo Cazanga (leonyomg17@gmail.com)
También podría gustarte
- Solemnes Biologia MoleculasDocumento56 páginasSolemnes Biologia MoleculasSabrina ApablazaAún no hay calificaciones
- Pautas Solemne 1 y 2 Biol240Documento10 páginasPautas Solemne 1 y 2 Biol240Leonidas LarrondoAún no hay calificaciones
- Autoevaluación ReplicaciónDocumento3 páginasAutoevaluación ReplicaciónDragontarlAún no hay calificaciones
- TP 9Documento3 páginasTP 9matiasAún no hay calificaciones
- Examen Bioquimica Parcial3 URJC MedicinaDocumento7 páginasExamen Bioquimica Parcial3 URJC MedicinaLara Fernández PedreiraAún no hay calificaciones
- Solemnes Biología MolecularDocumento12 páginasSolemnes Biología MolecularLeonidas LarrondoAún no hay calificaciones
- Inhibidor de DNA girasa y marcaje con timidina y uridina tritiadasDocumento6 páginasInhibidor de DNA girasa y marcaje con timidina y uridina tritiadasTifanny Cuellar RamosAún no hay calificaciones
- Bmi TDocumento176 páginasBmi TJuan Arcos HuasasquicheAún no hay calificaciones
- Dogma Central de La Biologia Celular y MolecularDocumento16 páginasDogma Central de La Biologia Celular y MolecularBlanca OrtizAún no hay calificaciones
- Replicación, Transcripción y Traducción.Documento4 páginasReplicación, Transcripción y Traducción.Valentina VergaraAún no hay calificaciones
- PROBLEMA5Documento3 páginasPROBLEMA5ivone fernandez0% (1)
- 888Documento10 páginas888api-627823169Aún no hay calificaciones
- 1er Examen Parcial (AP - Basesmol)Documento5 páginas1er Examen Parcial (AP - Basesmol)Karla AguilarAún no hay calificaciones
- 1er Examen Parcial (Ap - Basesmol)Documento5 páginas1er Examen Parcial (Ap - Basesmol)Karla AguilarAún no hay calificaciones
- Replicación Del DnaDocumento3 páginasReplicación Del DnaLuz MarianaAún no hay calificaciones
- Cap.06 Repli Repa Recom DNADocumento55 páginasCap.06 Repli Repa Recom DNAJuan Carlos Ledo GarciaAún no hay calificaciones
- Semana 3.1-Dogma de La BiologiaaDocumento9 páginasSemana 3.1-Dogma de La Biologiaaaimipatogallina1234Aún no hay calificaciones
- Taller 6 Flujo de La Información GenéticaDocumento7 páginasTaller 6 Flujo de La Información GenéticaAbigail BourdettaAún no hay calificaciones
- RESUMEN - Traducción, Transcripción y TraducciónDocumento6 páginasRESUMEN - Traducción, Transcripción y Traducciónanmenc04Aún no hay calificaciones
- Las DNADocumento8 páginasLas DNAClara SerranoAún no hay calificaciones
- TALLER 3 - ReplicaciónIyIIDocumento5 páginasTALLER 3 - ReplicaciónIyIIShin Hyun YooAún no hay calificaciones
- Banco de Preguntas y RespuestaDocumento7 páginasBanco de Preguntas y RespuestaRuth Carina Hilario CondorAún no hay calificaciones
- Taller # 2 BioquimicaDocumento4 páginasTaller # 2 BioquimicaFelipe MartinezAún no hay calificaciones
- Cuestionario de Repaso Acidos NucelicosDocumento4 páginasCuestionario de Repaso Acidos NucelicosYorlen VillaAún no hay calificaciones
- Replicacion Del ADN 26.03.2023Documento12 páginasReplicacion Del ADN 26.03.2023GabrielaAún no hay calificaciones
- TEMA 18 ADN Como Portador de InfoDocumento3 páginasTEMA 18 ADN Como Portador de InfoAkimitaAún no hay calificaciones
- Mecanismos Moleculares de La Replicación DelDocumento8 páginasMecanismos Moleculares de La Replicación DelItzel Rendon EugenioAún no hay calificaciones
- Preguntas de Repaso, Arumir Ortiz Adriana MayteDocumento7 páginasPreguntas de Repaso, Arumir Ortiz Adriana MayteArumir OrtizAún no hay calificaciones
- Sanchez - 5633 - Cuesationario de Acidos NucleicosDocumento9 páginasSanchez - 5633 - Cuesationario de Acidos NucleicosGabriela SanchezAún no hay calificaciones
- 7155-Texto Del Artã Culo-27926-1-10-20160908Documento8 páginas7155-Texto Del Artã Culo-27926-1-10-20160908jennifer MoralesAún no hay calificaciones
- Taller Sobre Ácidos Nucleicos y Energía TERMINADODocumento5 páginasTaller Sobre Ácidos Nucleicos y Energía TERMINADOJuanandres DuquerodrigezAún no hay calificaciones
- BIO PCE T3 SolucionesDocumento8 páginasBIO PCE T3 SolucionesAndrea ManziniAún no hay calificaciones
- SEGUNDO EXAMEN Bio Mol 2023ADocumento2 páginasSEGUNDO EXAMEN Bio Mol 2023ACRISTOPHER JOSUE MEDINA HUAMANAún no hay calificaciones
- Pauta Solemne 1 BIOL240 2nd SemestreDocumento9 páginasPauta Solemne 1 BIOL240 2nd SemestreLeonidas LarrondoAún no hay calificaciones
- 14 ACN3 Actividad de Complementación y Nivelación 2023-10Documento16 páginas14 ACN3 Actividad de Complementación y Nivelación 2023-10evalerap1Aún no hay calificaciones
- VI DNA Estructura y FunciónDocumento72 páginasVI DNA Estructura y FunciónXsiSkullAún no hay calificaciones
- U 5Documento8 páginasU 5Anabell Sanchez PuertinezAún no hay calificaciones
- ACTIVIDAD 7 Ciclo IVB Dogma de La Teoría CelularDocumento3 páginasACTIVIDAD 7 Ciclo IVB Dogma de La Teoría Celularandres santiago gonzalez muñozAún no hay calificaciones
- Foro Molecular Replicación MolecularDocumento7 páginasForo Molecular Replicación MolecularLeonardo Quintero MárquezAún no hay calificaciones
- Preguntas múltiple elección sobre replicación y transcripción del ADNDocumento63 páginasPreguntas múltiple elección sobre replicación y transcripción del ADNCale Poblette MacayaAún no hay calificaciones
- Biologia Capitulo4Documento21 páginasBiologia Capitulo4Domenica DelgadoAún no hay calificaciones
- Taller de Replicación - 2020-2Documento6 páginasTaller de Replicación - 2020-2Omar Guerrero0% (1)
- TallerDocumento22 páginasTallersebastian torresAún no hay calificaciones
- Prácticas de Introducción A La Biotecnología 2023Documento30 páginasPrácticas de Introducción A La Biotecnología 2023luis enrique rayon barronAún no hay calificaciones
- Actividad Celula II ABRIL 2020 UNIDAD IDocumento14 páginasActividad Celula II ABRIL 2020 UNIDAD IKATIA VERONICA GUILLEN SANCHEZAún no hay calificaciones
- Apuntes Biología Celular - Replicación y ReparaciónDocumento12 páginasApuntes Biología Celular - Replicación y Reparaciónaguirremicaela0Aún no hay calificaciones
- Semana 6 - Práctica 1Documento25 páginasSemana 6 - Práctica 1GERMAN LÔPEZZAún no hay calificaciones
- Seminario Bioquímica IIIDocumento6 páginasSeminario Bioquímica IIIMireia PenaAún no hay calificaciones
- Laboratorio PCRDocumento9 páginasLaboratorio PCRWendy Quiroz BerdugoAún no hay calificaciones
- Guía Colegió Prescott - Ácidos NucléicosDocumento8 páginasGuía Colegió Prescott - Ácidos NucléicosIsabella Cornejo LinaresAún no hay calificaciones
- Biotecnología, Tarea 4Documento7 páginasBiotecnología, Tarea 4monomonkisidaAún no hay calificaciones
- Recopilacion Genetica Examenes Listo 1Documento17 páginasRecopilacion Genetica Examenes Listo 1Alejandra FloresAún no hay calificaciones
- TALLER 3 - Replicación - ReparaciónDocumento3 páginasTALLER 3 - Replicación - ReparaciónIrene BecerraAún no hay calificaciones
- Estructura de Acidos NucleicosDocumento26 páginasEstructura de Acidos NucleicosCeleste PonceAún no hay calificaciones
- Examen 2Documento1 páginaExamen 2Dante AvilaAún no hay calificaciones
- ADN polimerasa: la enzima clave de la replicación del ADNDocumento11 páginasADN polimerasa: la enzima clave de la replicación del ADNTANIAAún no hay calificaciones
- Replicación Del DNA 2022Documento101 páginasReplicación Del DNA 2022jeniferAún no hay calificaciones
- Acceso a Universidad para Mayores de 25 años. Biología.: Solucionario Pruebas 2006-2012De EverandAcceso a Universidad para Mayores de 25 años. Biología.: Solucionario Pruebas 2006-2012Aún no hay calificaciones
- Introducción a la Biología: RESÚMENES UNIVERSITARIOSDe EverandIntroducción a la Biología: RESÚMENES UNIVERSITARIOSCalificación: 5 de 5 estrellas5/5 (1)
- Solemne 3 BIOL240 2021Documento7 páginasSolemne 3 BIOL240 2021Leonidas LarrondoAún no hay calificaciones
- Solemne 2 BIOL240 2021Documento3 páginasSolemne 2 BIOL240 2021Leonidas LarrondoAún no hay calificaciones
- Solemnes Biología MolecularDocumento12 páginasSolemnes Biología MolecularLeonidas LarrondoAún no hay calificaciones
- Solemnes 2 AnterioresDocumento37 páginasSolemnes 2 AnterioresArian Fernado Aravena ArianAún no hay calificaciones
- Síntesis Conceptuales de Biología Celula REALREALREALDocumento137 páginasSíntesis Conceptuales de Biología Celula REALREALREALAcis SamuelAún no hay calificaciones
- Clase 8 y 9 - Replicación, Transcripción y TraducciónDocumento24 páginasClase 8 y 9 - Replicación, Transcripción y TraducciónAndrés Sepúlveda NavarroAún no hay calificaciones
- Variantes de Pcr175Documento5 páginasVariantes de Pcr175Jaqueline PedrozaAún no hay calificaciones
- Transcripcion de AdnDocumento6 páginasTranscripcion de AdnSantiago LópezAún no hay calificaciones
- Sintesis de ProteinasDocumento7 páginasSintesis de ProteinasGiovanny Alexander Zamudio CastellanosAún no hay calificaciones
- Genes y manipulación genética: transcripción y traducciónDocumento5 páginasGenes y manipulación genética: transcripción y traducciónalex mAún no hay calificaciones
- 1.5 Nucleósidos y NucleótidosDocumento40 páginas1.5 Nucleósidos y NucleótidosKenneth CruzAún no hay calificaciones
- Proceso de Replicación +transcripción+traducciónDocumento3 páginasProceso de Replicación +transcripción+traducciónjm rcAún no hay calificaciones
- Mecanismos Genéticos BásicosDocumento22 páginasMecanismos Genéticos BásicosCristobal GeneralAún no hay calificaciones
- Geneticamolecular1parteadnreplicacintranscripcinytraduccin 120330063336 Phpapp02Documento30 páginasGeneticamolecular1parteadnreplicacintranscripcinytraduccin 120330063336 Phpapp02claudiaAún no hay calificaciones
- Aplicación de Técnicas de PCR y Electroforesis-Presentación PDFDocumento15 páginasAplicación de Técnicas de PCR y Electroforesis-Presentación PDFOmar MohamedAún no hay calificaciones
- EpigenéticaDocumento54 páginasEpigenéticaValeria CuervoAún no hay calificaciones
- Cuestiones de Genética Molecular (Resuelto)Documento2 páginasCuestiones de Genética Molecular (Resuelto)genusxyzAún no hay calificaciones
- PARCIAL 3 UNIFICADO BiologiaDocumento27 páginasPARCIAL 3 UNIFICADO BiologiaCORAPONEIVAAún no hay calificaciones
- Adn ExpoDocumento16 páginasAdn Expoarellys sonccoAún no hay calificaciones
- Estructura de Los Ácidos NucleicosDocumento5 páginasEstructura de Los Ácidos NucleicosYesid HernandezAún no hay calificaciones
- Cien 9 b2 p3 Est Web PDFDocumento10 páginasCien 9 b2 p3 Est Web PDFshineAún no hay calificaciones
- Las tres generaciones de secuenciaciónDocumento3 páginasLas tres generaciones de secuenciaciónIphyi BurgosAún no hay calificaciones
- Guia Didactica 3 y 4Documento5 páginasGuia Didactica 3 y 4Josue QuinteroAún no hay calificaciones
- Estudio dirigido de ácidos nucleicos: sus componentes y estructuraDocumento16 páginasEstudio dirigido de ácidos nucleicos: sus componentes y estructuraElizabeth De la ossaAún no hay calificaciones
- Control Genético de La Síntesis ProteicaDocumento21 páginasControl Genético de La Síntesis ProteicaAlma MirandaAún no hay calificaciones
- Plenaria Envoltura Nuclear y Replicación - Septiembre 26Documento24 páginasPlenaria Envoltura Nuclear y Replicación - Septiembre 26Estefanía HerreraAún no hay calificaciones
- TALLER REPLICACIONdocxDocumento10 páginasTALLER REPLICACIONdocxJohana PedrozoAún no hay calificaciones
- 2021 Guía 3 Estructura Del AdnDocumento2 páginas2021 Guía 3 Estructura Del AdnDuvan FelipeAún no hay calificaciones
- Genética Molecular - Dra. Gisselle Pérez QuezadaDocumento2 páginasGenética Molecular - Dra. Gisselle Pérez QuezadaMarlin TavarezAún no hay calificaciones
- Quien CodificoDocumento21 páginasQuien Codificosergio Art palmerosAún no hay calificaciones
- Código GenéticoDocumento40 páginasCódigo GenéticoEduardaBautistaBarredaAún no hay calificaciones
- Actividad 2. Procesos Del AdnDocumento2 páginasActividad 2. Procesos Del AdnAlejandro NgAún no hay calificaciones
- Guía de Práctica 7Documento4 páginasGuía de Práctica 7EVELYN MANUELA GONZALES CHAVEZAún no hay calificaciones
- Biología 4°A I. Bofi 27 04 20Documento2 páginasBiología 4°A I. Bofi 27 04 20DANY HERNANDEZ HERNANDEZAún no hay calificaciones