Documentos de Académico
Documentos de Profesional
Documentos de Cultura
2 Fundamentos PCR
Cargado por
Diego MeléndezDerechos de autor
Formatos disponibles
Compartir este documento
Compartir o incrustar documentos
¿Le pareció útil este documento?
¿Este contenido es inapropiado?
Denunciar este documentoCopyright:
Formatos disponibles
2 Fundamentos PCR
Cargado por
Diego MeléndezCopyright:
Formatos disponibles
Biología Molecular ENCB, IPN
7QM3
e
kaneBit
La Reacción en Cadena de la Polimerasa (PCR)
in vitro (solo enzimas)
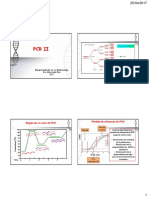
Reacción en cadena de la polimerasa: es una técnica que permite obtener rápidamente una
gran cantidad de copias de un fragmento específico de DNA. Consta de varios ciclos (25–45), y
cada ciclo tiene 3 etapas:
Separa DNAencadenas sencillas
• Desnaturalización (95ºC)
iniciadores
• Alineamiento de los iniciadores 1550) Pegan
Ciclo 3
• Polimerización, alargamiento o extensión (72ºC) Tag pol Temp. Optima
- se empiezan a obtener los fragmentos de interés, limitados por los iniciadores
Condiciones de reacción para una PCR convencional:
↳tandario
neutraliza carga
Si se estandarios y seprebar
dif. 2My] sin que aparezca
banda inespecifica
Cofactor Tay pul
Alta fidelidad
Arquea Otras arqueas
Termusetious Grum(
DNA polimerssadependiente DNA.I -> Pyrococcusturiosus
Pyrucocius i
woesi
I
Vida media qumin a 95%constante Thermus termophilos
Velocidad Thereus flars
3 1000
nucleitidos/ 72°
PCR depunto final
polimerizacich m in
inermococus litoralis
a
Termoestable
Direccick 5" - 3"
Programación del termociclador para una PCR convencional: Molde DWA
Iniciadores
y exonuclusa 5" - 3"
·
NO act.editura de
35
graescamesiere
E
0 95°
Dif. Temperatura entre base
y tapa
hace que no se evapor
Secute ahora poner aceite mineral a cada tubo
Clase del jueves 23 de marzo del 2023 1
5"Cy(94AAT AG4TGCT
4(
Biología Molecular LOS ENCB, IPN
E
7QM3 Alinear iniciadores
Tm= 402((+C) 2°(A T)
+
+
3A
Ta Tm
=
-
50) 5T
Confirmar experimentalmente
im
40(((4) 2°(6)
=
+
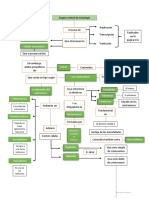
Principales factores que afectan a la PCR: Tm 68° =
Ta 68° 50) 63°
=
=
-
Temp. fusich
Factor Efecto sobre la PCR T50% DNA
Alta. Penrgicinética esta enduble
Baja de Sepega pero en
otrus lados y
Las temperaturas de desnaturalización (95ºC) y de polimerización cudence y 50%
encadena sencilla.
(72ºC) son fijas.
bandas inespecíficas
genera
Temperaturas Mi inicida
·
sopegre
a La temperatura de alineamiento de los iniciadores se calcula como
Ta = Tm-5ºC, donde Tm =*
por
4ºC[G+C] + 2ºC[A+T]. Regla de Wallace
secuencia blanco
lemp. Sptimall
-
enlaces de puente cit
Iniciador Forward: se une sobre la cadena molde del DNA. 3- 5
codificante Tamano
11 111134
,TM fragment o
013 s Reverse ampliad 8
Iniciador Reverse: se une cobre la cadena codificadora del DNA. sitI
un
sitoria,
115 200 -
500bp
Molche
Iniciadores El tamaño de los iniciadores es de 20 a 25 nucleótidos.
Ambos iniciadores deben tener las mismas Tm, y unirse
solamente a la secuencia de interés en el genoma.
Longitudde producto:(Ultima posicion iniciador Reverse)- (Primera posicion del iniciador Forward) #A
Es un cofactor de la DNA polimerasa.
Concentración de Mg++ Una mayor concentración de Mg++ aumenta la eficiencia de la
PCR, pero puede favorecer la amplificación de fragmentos
1.8 mi
inespecíficos.
ubien
a Los cationes (K+) neutralizan la carga negativa de los grupos
Fuerza iónica
Sino, la cadena mulde; amás se va a poder fosfato del DNA, para permitir el alineamiento de los iniciadores.
acerchr
Aten"miento Disminuye la amplificación de productos inespecíficos y los
Hot start Tay pol 72°C
artefactos derivados de dímeros de iniciadores.
I
Dependen de la cantidad de DNA presente en la muestra original.
Número de ciclos de la
No se recomiendan más de 45 ciclos de reacción, debido a la
reacción
inactivación de la Taq polimerasa.
Tha. pol
↓ de 25 cidos limite int.
Hasta DrAse duplica
10ry actividad.
atividad
cido 30 el
si
I
si tienes Drt efectivamente
a 250( lo puedus visualizar en
gel Después de 30 es una meseta
de ↳> sedesnaturaliza
agarosa.
Scleurace un
Taq polimerasa
antiverpo que
bloquea su sitio -> 45ciclos máximo de Act. residual
activo y hasta
el ver ciclo empieza
I de 45cicks, paras reaccion y
a
trabajar
agregas + pol.
Tay
Trabajar siempre en
bielo para no
general
productos
chespecifices
Más información:
El
primer
dimerg" Premier Biosoft PCR Primer Design Guidelines
(http://www.premierbiosoft.com/tech_notes/PCR_Primer_Design.html)
Clase del jueves 23 de marzo del 2023 2
-
lamaño iniciadores
Iniciador 8 nuclertidos
Sitiosmiincada 48 65,536bp =
voltearSuenciacione
*
Youbp. genam
ne n
a las companics
=>
49,000 sitrusen los que se puede pegar
Garanticar quesolo se
pegue
i vez.
⑧
↳ 17nucóticlos
4* 17,179,869,184bp.
=
Cmayor tamuno bp humano).
- ↳↳ I sitio mis en el
genoma
humano
Iniciadores.
Tamaro tipico -> 20 a 25 nuclertidos
· Famaño.20-25
nuclertidos
Concentracio final de cada
Contenido (C 40-60%
(Usualmentee
·
Iniciador
0.1-1.0pM
(hammeros che"s
·
No formar estructura 7o
entre si
· No complementarios
Desuntain
· Tienen misma Tm
(lungitud similar, composicion bases similar). GerirI tlarges,
· Sepegrensolo asemencia desende a
↑ paso alieamiento
estruct. 7 DNA.
Alinena una
regich del DrA que no farme en el
Puede
·
·
formar estructuras
SewadaNac
*
-Fiempo reall
Sistema Man Apired Systems
tay
Ivane en garradosue
-
SEB
3.
apagador
Emitflucescencia
en ↓ I
↳ wie captada pare
Aluorescente
*. ->
Ahoraen
qute
nuchutudes
Semeten todos los DNA de ↑ cavo por suespecificidad
duble cadena
ciclo
Fluorescensic Umbrat
Elcido umbral
inversamente
Du presente
con
en
(CT) conduciona
la cantidad
la mezcla de
de
~ ciclo
#
Log. (cuncentracich)
reasiol
↑ [PMAT - ciclo X
↓ [DNA] - ciclo
También podría gustarte
- 2010 - 2011 Febrero 1Documento6 páginas2010 - 2011 Febrero 1rafa0254Aún no hay calificaciones
- Cuadro Comparativos Tipos de Pcr.Documento4 páginasCuadro Comparativos Tipos de Pcr.Juan Sebastian Santofimio Villamizar0% (1)
- Mapa de Los Componentes para Realizar Un PCRDocumento2 páginasMapa de Los Componentes para Realizar Un PCREudaldo Cime FigueroaAún no hay calificaciones
- Clase 4 Técnicas Biología Molecular PCRDocumento38 páginasClase 4 Técnicas Biología Molecular PCRalexiajavierajlAún no hay calificaciones
- Biología CelularDocumento78 páginasBiología CelularConni MatteAún no hay calificaciones
- Tema 5 GMDocumento55 páginasTema 5 GMCristinu TorresAún no hay calificaciones
- Cuadro MicrobiológicoDocumento4 páginasCuadro MicrobiológicoMaira PerezAún no hay calificaciones
- Proteínas: estructura y funciónDocumento27 páginasProteínas: estructura y funciónSofía A. SánchezAún no hay calificaciones
- C8 Transcripción Del ADN 2020 20Documento17 páginasC8 Transcripción Del ADN 2020 20Lia LigardoAún no hay calificaciones
- Tecnicas de PCRDocumento64 páginasTecnicas de PCRjackridenAún no hay calificaciones
- Keny Davi Alvarado QuirozDocumento53 páginasKeny Davi Alvarado QuirozLidiaAlejandraRiveraPonceAún no hay calificaciones
- Célula NucleoDocumento42 páginasCélula NucleoItzel HinojosAún no hay calificaciones
- Diagrama de FlujoDocumento1 páginaDiagrama de FlujoDuncan Silva100% (2)
- And (Traduccion y Trascripcion)Documento1 páginaAnd (Traduccion y Trascripcion)Ale RamirezAún no hay calificaciones
- 4-Estudio de Las Mutaciones Relacionadas Con El Cáncer para Establecer La Terapia Adecuada-2Documento1 página4-Estudio de Las Mutaciones Relacionadas Con El Cáncer para Establecer La Terapia Adecuada-2Marieta GarcíaAún no hay calificaciones
- Que Es La PCRDocumento9 páginasQue Es La PCRYasmine Guerrero RojasAún no hay calificaciones
- Biologia Molecular - PCR PDFDocumento78 páginasBiologia Molecular - PCR PDFmanuel1788Aún no hay calificaciones
- Factores que afectan la eficiencia de PCRDocumento12 páginasFactores que afectan la eficiencia de PCRJhoimar Ubaque BernalAún no hay calificaciones
- Biología Parcial 1 PDFDocumento52 páginasBiología Parcial 1 PDFSol VargasAún no hay calificaciones
- Biología MolecularDocumento15 páginasBiología MolecularCarolina De la peña fernandezAún no hay calificaciones
- Tabla de RadiofármacosDocumento3 páginasTabla de RadiofármacosGodslayer 33Aún no hay calificaciones
- Copia de Célula ProcarionteDocumento1 páginaCopia de Célula ProcarionteJorge Nitales PulidosAún no hay calificaciones
- Comparativo transcripción procariotas-eucariotasDocumento1 páginaComparativo transcripción procariotas-eucariotasAngel Vinicio GuachizacaAún no hay calificaciones
- Mapa Conceptual Semana 03 - Luna Victoria AcevedoDocumento1 páginaMapa Conceptual Semana 03 - Luna Victoria AcevedoDone MLAún no hay calificaciones
- EsquemaDocumento4 páginasEsquemanuriaob267Aún no hay calificaciones
- Presentación PCR VirtualDocumento32 páginasPresentación PCR VirtualPaula AbadAún no hay calificaciones
- La Teìcnica de PCR 2110Documento48 páginasLa Teìcnica de PCR 2110Miguel candelaAún no hay calificaciones
- Síntesis de ProteínasDocumento3 páginasSíntesis de ProteínasSofi JungAún no hay calificaciones
- Act BioDocumento3 páginasAct BioSofi JungAún no hay calificaciones
- POLIMEROSDocumento2 páginasPOLIMEROSLeonaedoAún no hay calificaciones
- Item 11Documento2 páginasItem 11CREACIONES Y SUMINISTROS SASAún no hay calificaciones
- Metodos Basados en Studio de ADN 29-04-09 S.arduinoDocumento20 páginasMetodos Basados en Studio de ADN 29-04-09 S.arduinoLednys Palomino ZambranoAún no hay calificaciones
- Clase 3 - Técnicas de Amplificación - 2022 HCDocumento21 páginasClase 3 - Técnicas de Amplificación - 2022 HCLuis David Gómez GálvezAún no hay calificaciones
- Mapa Mental BioquímicaDocumento5 páginasMapa Mental BioquímicaFernanda CuevasAún no hay calificaciones
- Reacción en Cadena de La Polimerasa (PCR), Transcripción Inversa RT-PCRDocumento39 páginasReacción en Cadena de La Polimerasa (PCR), Transcripción Inversa RT-PCRGraciela CalderonAún no hay calificaciones
- Dirección Estratégica Del TH Grupo ADocumento12 páginasDirección Estratégica Del TH Grupo ADaniel SalgadoAún no hay calificaciones
- MICROSCOPIODocumento3 páginasMICROSCOPIOAngieAún no hay calificaciones
- Técnicas de estudio en genética molecularDocumento40 páginasTécnicas de estudio en genética molecularFio LopezAún no hay calificaciones
- Membranas y TransportEDocumento7 páginasMembranas y TransportEValeria Vallecillo MolanoAún no hay calificaciones
- Cuadro ComparativoDocumento2 páginasCuadro ComparativoCristhian Rodriguez PeñaAún no hay calificaciones
- Clase 7 PCRDocumento59 páginasClase 7 PCRIbeth OnceAún no hay calificaciones
- Deteccion Virus Tabaco y TomateDocumento7 páginasDeteccion Virus Tabaco y TomateChary E. QuincheAún no hay calificaciones
- Dogma central biología procesos replicación transcripción traducciónDocumento3 páginasDogma central biología procesos replicación transcripción traducciónDiego Rojas ChacónAún no hay calificaciones
- Tecnicas de Diagnostico Molecular Aplicadas Al Lab 06 de NoviembreDocumento40 páginasTecnicas de Diagnostico Molecular Aplicadas Al Lab 06 de NoviembregemaAún no hay calificaciones
- PCRDocumento32 páginasPCRLuis Gerardo GarciaAún no hay calificaciones
- 11 Técnicas de Diagnóstico MolecularDocumento122 páginas11 Técnicas de Diagnóstico MolecularJuanjo Rosero Benavides100% (1)
- Núcleo y ADNDocumento16 páginasNúcleo y ADNcxnwndm45qAún no hay calificaciones
- HRMDocumento18 páginasHRMPaola RiosAún no hay calificaciones
- Unidad 7: Técnicas de PCRDocumento9 páginasUnidad 7: Técnicas de PCRMary Cruz Collahuazo FloresAún no hay calificaciones
- QuizDocumento6 páginasQuizshirley katherine olarte nievesAún no hay calificaciones
- UT5. Técnicas de PCRDocumento12 páginasUT5. Técnicas de PCRNerea IzquierdoAún no hay calificaciones
- Clase Fundamentos PCRDocumento48 páginasClase Fundamentos PCRCateAún no hay calificaciones
- Sergio Analitica Cuadro Avance 3Documento3 páginasSergio Analitica Cuadro Avance 3sergiopoemapeAún no hay calificaciones
- Guia 8Documento13 páginasGuia 8Mariana PiedrahitaAún no hay calificaciones
- PCR 2022Documento24 páginasPCR 2022Wilmer ParedesAún no hay calificaciones
- Pasos PCRDocumento5 páginasPasos PCRLaura SanabriaAún no hay calificaciones
- Bioquímica IIDocumento2 páginasBioquímica IIa190099Aún no hay calificaciones
- Biologia Molecular Aplicada Al DiagnosticoDocumento149 páginasBiologia Molecular Aplicada Al Diagnosticoprometeo2014Aún no hay calificaciones
- Mapas Bio 3Documento26 páginasMapas Bio 3Valentini MAún no hay calificaciones
- Guia #2 - Grupo 10 - Gracia - Farida y SadyDocumento4 páginasGuia #2 - Grupo 10 - Gracia - Farida y SadyDaniela ReyesAún no hay calificaciones
- 019-Catalogo Hydromagic 2021Documento30 páginas019-Catalogo Hydromagic 2021Raymundo VazquezAún no hay calificaciones
- Mallas de PoliuretanoDocumento31 páginasMallas de PoliuretanoWilson Quispe Lopez100% (1)
- ATLASDELPLASTICOESPANOL2020Documento52 páginasATLASDELPLASTICOESPANOL2020Albert AnticonaAún no hay calificaciones
- Base Datos Sector PlasticoDocumento648 páginasBase Datos Sector PlasticoJose D. Sierra100% (1)
- Catalogo de Suspension 2023Documento120 páginasCatalogo de Suspension 2023Josue Loarca100% (1)
- Actividad 1. Unidad 2Documento19 páginasActividad 1. Unidad 2EDNA GUADALUPE ESCALANTE MORAAún no hay calificaciones
- Productos RothcoDocumento27 páginasProductos RothcomanuelzorroAún no hay calificaciones
- Cierre Bandejas PET Chile - 10 08 2021Documento12 páginasCierre Bandejas PET Chile - 10 08 2021Valentina RodriguezAún no hay calificaciones
- Polimerasas Del ADNDocumento2 páginasPolimerasas Del ADNAbril ChaviraAún no hay calificaciones
- ISOMETRICO 2-ModeloDocumento1 páginaISOMETRICO 2-ModeloE Ivan JuárezAún no hay calificaciones
- MecanismosPolimerizacionDocumento20 páginasMecanismosPolimerizacionalexadame2301Aún no hay calificaciones
- Activdades-Recuperación - 3º ESO (1º Parte)Documento15 páginasActivdades-Recuperación - 3º ESO (1º Parte)Carmen SoberonAún no hay calificaciones
- PolimerosDocumento18 páginasPolimerosAl Espinoza FloresAún no hay calificaciones
- Correas de SincronismoDocumento3 páginasCorreas de Sincronismoicqbujanda9772Aún no hay calificaciones
- Complemento Primavera VeranoDocumento84 páginasComplemento Primavera VeranoXochitl VillanuevaAún no hay calificaciones
- Tageado y Señalizacion CablesDocumento4 páginasTageado y Señalizacion CablesAnonymous gacFnRPYsAún no hay calificaciones
- El Plástico Es Considerado Un Material Polimérico OrgánicoDocumento10 páginasEl Plástico Es Considerado Un Material Polimérico OrgánicoAbelVirgenAún no hay calificaciones
- Nte Inen 2034-1995 EsponjasDocumento8 páginasNte Inen 2034-1995 EsponjasPaola Andrea Contreras MartinezAún no hay calificaciones
- Clasificación de Los Polímeros para Envase y EmbalajeDocumento4 páginasClasificación de Los Polímeros para Envase y EmbalajeJuan GomezAún no hay calificaciones
- CandyDocumento21 páginasCandyEmnamagali Lopez CervantesAún no hay calificaciones
- Inventario en Informe de Almacén (Detallado)Documento3 páginasInventario en Informe de Almacén (Detallado)Yeinerth GonzalezAún no hay calificaciones
- Ryocco 5-2021Documento124 páginasRyocco 5-2021William Paez FlorezAún no hay calificaciones
- 1 Empaque Plasticos 16-04-2020Documento23 páginas1 Empaque Plasticos 16-04-2020Dayana GsAún no hay calificaciones
- Especialidad EVADocumento11 páginasEspecialidad EVAJosé DanielAún no hay calificaciones
- CERAS - Bonger Química5 PDFDocumento5 páginasCERAS - Bonger Química5 PDFjsk789Aún no hay calificaciones
- Diapositivas Gasa y Empaquetamiento QuirurgicoDocumento30 páginasDiapositivas Gasa y Empaquetamiento QuirurgicoMartin S. Peralta33% (3)
- Tarea 5Documento2 páginasTarea 5JOSE DAVID MARTINEZ MENCO ESTUDIANTEAún no hay calificaciones
- Polímeros: Características y aplicacionesDocumento15 páginasPolímeros: Características y aplicacionesJessica Celeste Yauri SantiagoAún no hay calificaciones
- Papelera rectangular 30L para oficinas y hogarDocumento1 páginaPapelera rectangular 30L para oficinas y hogarJuro_244303Aún no hay calificaciones