Documentos de Académico
Documentos de Profesional
Documentos de Cultura
Seminario 1 PDF
Cargado por
maria0 calificaciones0% encontró este documento útil (0 votos)
23 vistas9 páginasTítulo original
SEMINARIO 1.pdf
Derechos de autor
© © All Rights Reserved
Formatos disponibles
PDF, TXT o lea en línea desde Scribd
Compartir este documento
Compartir o incrustar documentos
¿Le pareció útil este documento?
¿Este contenido es inapropiado?
Denunciar este documentoCopyright:
© All Rights Reserved
Formatos disponibles
Descargue como PDF, TXT o lea en línea desde Scribd
0 calificaciones0% encontró este documento útil (0 votos)
23 vistas9 páginasSeminario 1 PDF
Cargado por
mariaCopyright:
© All Rights Reserved
Formatos disponibles
Descargue como PDF, TXT o lea en línea desde Scribd
Está en la página 1de 9
SEMINARIO 1
1. Una preparación de una muestra de DNA lineal se divide en tres alícuotas:
-Una se digiere con la enzima BamHI, generando tres fragmentos de 4 kb,6 kb y 15 kb.
-Otra alícuota se digiere con la enzima SmaI, generando dos fragmentos de 7 kb y 18
kb.
-La tercera, se digiere con ambas enzimas, generando fragmentos de 3 kb, 4 kb, 12 kb
y 6 kb.
a) ¿Cuántas dianas hay en la molécula de DNA para cada una de las enzimas de
restricción?
b) Dibuja un mapa de restricción de la molécula de DNA indicando la posición de las
dianas de las tres enzimas utilizadas. ¿Puede haber más de un mapa que encaje con
estos datos?
2. El plásmido pBR325 se digirió con distintas enzimas de restricción. Se realizó
una electroforesis de los digeridos. El DNA de lambda cortado con BstEII se utilizó
como patrón de tamaños. Deduce el mapa de restricción del plásmido.
Calle 1: Lambda/BstEII. Calle 2: EcoRI. Calle 3: EcoRI + SalI. Calle 4: SalI +
PstI. Calle 5: EcoRI + HindIII. Calle 6: PstI + HindIII. Calle 7: PvuII +
HindIII. Calle 10: BamHI + EcoRI. Calle 11: BamHI + PvuII.
Realizando la curva patrón e interpolando las distancias
recorridas por los diversos fragmentos de restricción
podemos calcular sus tamaños:
EcoRI EcoRI SalI EcoRI PstI PvuII EcoRI HindIII EcoRI PvuII
SalI PstI HindIII HindIII PvuII PvuII BamHI BamHI
6.0 4.1 3 1.3 3.6 3.4 3.3 2.6 4.4 2.6
1.9 4.7 2.4 2.6 2.6 2 1.6 1.7
1.4
3. Un DNA circular se digiere, en tres reacciones separadas, con dos enzimas de
restricción; el análisis por electroforesis de los fragmentos obtenidos da los siguientes
resultados:
a) Digestión con EcoRI: 2 fragmentos de 5030 y 2690 pb.
b) Digestión con BamHI: 1 fragmento de 7720 pb.
c) Digestión con EcoRI y BamHI : 3 fragmentos de 1100, 2690 y 3930 pb.
Dibuja el mapa de restricción.
4. En la tabla se indican los resultados de la caracterización de dos tipos de ácidos
nucleicos extraídos de los bacteriófagos X e Y. Se dan los contenidos en tres de las
bases, la sensibilidad a dos ribonucleasas y dos desoxirribonucleasas distintas y la
absorbancia a 260 nm (A260) de muestras de ácido nucleico antes y después de
calentarlas a 100 ⁰C durante 15 minutos. El signo + indica que el ácido nucleico es
degradado por la nucleasa correspondiente, y el signo -, que no es degradado. Indica la
naturaleza molecular de los ácidos nucleicos de los fagos X e Y, con el mayor detalle
que permitan los datos. ¿Qué posible/s explicación/es justificaría el hecho de que la
desorribonucleasa 2 no actúe sobre el ácido nucleico Y?
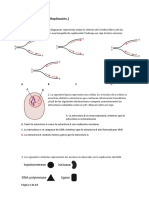
5. Las moléculas de DNA bicatenario que se representan en la figura tienen grupos
fosfato en sus extremos 5’ e hidroxilos en los 3’. Indica qué enzimas emplearías y en
qué orden y, en su caso, qué sustratos adicionales serían necesarios, para:
a-convertir en romos los extremos sobresalientes de la molécula A sin acortarla.
b-marcar radiactivamente los extremos 5’ de B.
c-para circularizar D.
e-para marcar radiactivamente la mayor parte de E.
f- para circularizar F de la manera más eficaz posible.
(Las circularizaciones se llevan a cabo in vitro y dan lugar a moléculas de DNA
covalentemente cerradas.)
6. Se desea unir dos fragmentos de DNA, uno con extremos PstI (CTGGA’G) y otro con
extremos BamHI (G’GATCC), recuperando ambas dianas en la molécula final. Se
desea, además, que entre los dos fragmentos exista una diana EcoRI (G’AATTC), una
SspI (AAT’ATT) y otra KpnI (G’GTACC). Diseña la molécula adaptadora para poder
realizar el objetivo.
7. Se desea obtener una molécula de DNA circular que incorpore la región B presente
en p1 y la región Z presente en p2. Para ellos se realizaron distintos tratamientos a p1 y
p2 antes de proceder a la unión de los fragmentos con DNA ligasa. La estabilidad de
una molécula circular requiere la presencia de la región A o de la Z, pero nunca de las
dos. Indica qué moléculas circulares se obtendrán formadas por uno o dos fragmentos
producto y qué tratamiento sería el óptimo para la obtención de la molécula deseada
con el menor número de subproductos. Las enzimas EcoRI y BamHI no rinden
extremos compatibles.
Tratamiento 1: digestión con EcoRI de p1 y p2.
T2: digestión con BamHI de p1 y p2.
T3: + EcoRI y fosfatasa de p1 y digestión con EcoRI p2.
T4: + EcoRI de p1 y doble digestión con EcoRI y BamHI de p2.
T5: doble digestión de p1 y p2 con EcoRI y BamHI.
T6: doble digestión de p1 con EcoRI y BamHI, electroforesis y
selección de la banda B; doble digestión de p2 con EcoRI y BamHI.
T7: doble digestión de p1 y p2 con EcoRI y BamHI, electroforesis y
selección de la banda B; doble digestión de p2 con EcoRI y BamHI y
posterior tratamiento con fosfatasa.
8. La fibrosis quística es una enfermedad autosómica recesiva. Los mapas de las dianas de
EcoRI (R) en los alelos silvestre y mutante del gen de la fibrosis quística se muestran en la
parte superior de la figura. Se obtuvieron muestras de DNA de un feto (F) y sus padres (M y
P), que fueron sometidas a una electroforesis en gel de agarosa seguida de una
transferencia a membrana por el método de Southern. La membrana fue hibridada con una
sonda FQ marcada radiactivamente, obteniéndose la autorradiografía mostrada en la parte
inferior de la figura.
¿Padecerá el feto la enfermedad? Justifica tu respuesta
y haz un esquema del procedimiento.
Los polimorfismos de longitud de fragmentos de restricción (RFLP)
representan las diferencias en las secuencias de DNA homólogo a nivel
de variación de fragmentos de restricción de tamaños diferentes tras la
digestión con enzimas de restricción específicas. Pueden utilizarse
como marcadores, ya que ambos alelos pueden ser detectados y son
altamente específicos de locus. El análisis de la variación genética del
DNA genómico genera patrones de bandas muy complejas cuya
interpretación se facilita mediante la utilización de la técnica de
southern blot con sondas marcadas específicas de los alelos que se
quieren detectar.
9. La huella molecular* tiene entre otros usos el de intentar identificar niños robados, con
el propósito de devolverlos a sus padres biológicos. Sarah Sting fue secuestrada cuanto
tenía 4 años, y 12 años después, se acusó del secuestro a Eric y Kristin Neman, que
vivían con su hija Betty de 16 años. Se tomaron muestras de DNA del padre (PS)y la
madre (MS) de Sarah, así como de Betty (BN), Eric (EN) y Kristin (KN) Neman, y se
obtuvieron sus huellas moleculares empleando una sonda multilocus para una familia de
VNTR (Variable Number of Tandem Repeats), cuyo resultado se muestra en la figura.
Explica los pasos necesarios para llevar a cabo este proceso.
¿Crees que los resultados de la autorradiografía
permiten saber si Betty es hija o no de Eric y Kristin?
¿Podríamos concluir con mucha probabilidad o total
certeza si Betty Neman es Sarah Sting?
*La “huella genética (DNA fingerprinting)” es una técnica mediante
la cual se detectan simultáneamente varios minisatélites del
genoma obteniéndose un patrón único para cada individuo. La
probabilidad de que dos personas que no sean gemelos idénticos
tengan la misma huella o patrón es muy pequeña.
También podría gustarte
- Introducción a la Biología: RESÚMENES UNIVERSITARIOSDe EverandIntroducción a la Biología: RESÚMENES UNIVERSITARIOSCalificación: 5 de 5 estrellas5/5 (1)
- Biología Celular - Núcleo-9-16Documento34 páginasBiología Celular - Núcleo-9-16lup M V.Aún no hay calificaciones
- PCR - Reaccion en Cadena de La Polimerasa y AluDocumento9 páginasPCR - Reaccion en Cadena de La Polimerasa y AluArantxa González IturraAún no hay calificaciones
- GUÍA 1 ELEC Modelo y Replicación de ADNDocumento8 páginasGUÍA 1 ELEC Modelo y Replicación de ADNCarla Javiera Abarca0% (1)
- Ejercicios de GeneticaDocumento4 páginasEjercicios de Geneticasol roque tueros100% (1)
- Ejercicios de GenéticaDocumento32 páginasEjercicios de GenéticaSheila GarcíaAún no hay calificaciones
- Biologia Molecular Enero 2004Documento2 páginasBiologia Molecular Enero 2004maferfranciaAún no hay calificaciones
- Guía de Problemas - 1ra Parte - BCM Nutricion 2023 - Clase 1Documento8 páginasGuía de Problemas - 1ra Parte - BCM Nutricion 2023 - Clase 1leonardo farioliAún no hay calificaciones
- Replicación Del DNA 2022Documento101 páginasReplicación Del DNA 2022jeniferAún no hay calificaciones
- Herramienta Moleculares y Su Aplicación ClínicaDocumento15 páginasHerramienta Moleculares y Su Aplicación ClínicaZUHEIRY PCAún no hay calificaciones
- Actividad Replicacion DnaDocumento6 páginasActividad Replicacion DnaJOAQUIN SEBASTIAN GONZALES CHIRINOSAún no hay calificaciones
- Taller Bases Quimicas de La HerenciaDocumento4 páginasTaller Bases Quimicas de La HerenciaEDISON CANO ESCOBARAún no hay calificaciones
- Biología Tema 4 LibretaDocumento3 páginasBiología Tema 4 Libretasara caro lópezAún no hay calificaciones
- Pauta Solemne 1 BIOL240 2nd SemestreDocumento9 páginasPauta Solemne 1 BIOL240 2nd SemestreLeonidas LarrondoAún no hay calificaciones
- TP 02 Electroforesis en Geles de Agarosa - 1c23Documento7 páginasTP 02 Electroforesis en Geles de Agarosa - 1c23saaciarciancioAún no hay calificaciones
- Genetica Seminario 2Documento5 páginasGenetica Seminario 2Lucia RamirezAún no hay calificaciones
- Replicación Del ADNDocumento13 páginasReplicación Del ADNRomina OrejuelaAún no hay calificaciones
- Guia de Seminarios Unidad 2 y 3 FinalDocumento13 páginasGuia de Seminarios Unidad 2 y 3 FinalFlorencia LoboAún no hay calificaciones
- ProblemarioDocumento10 páginasProblemariomagAún no hay calificaciones
- Ebau Genética Molecular y Mutaciones Corregidos PDF Rna AdnDocumento1 páginaEbau Genética Molecular y Mutaciones Corregidos PDF Rna Adndanielasev.06Aún no hay calificaciones
- ADN ForenseDocumento5 páginasADN ForenseGonzalo AguilarAún no hay calificaciones
- 02 A Estructura y Propiedades Físico-Químicas Del DNA23Documento52 páginas02 A Estructura y Propiedades Físico-Químicas Del DNA23gutierrezfk33Aún no hay calificaciones
- Solemnes Biología MolecularDocumento12 páginasSolemnes Biología MolecularLeonidas LarrondoAún no hay calificaciones
- Z.biol Mol Cues 1 2021Documento3 páginasZ.biol Mol Cues 1 2021Celia Gonzales PérezAún no hay calificaciones
- PreguntasDocumento7 páginasPreguntasDaniela IbarraAún no hay calificaciones
- Biología MolecularDocumento6 páginasBiología MolecularNatalia MorenoAún no hay calificaciones
- Replicacion ADNDocumento39 páginasReplicacion ADNVidal RTAún no hay calificaciones
- INFORME 1 Merged MergedDocumento22 páginasINFORME 1 Merged MergedJosecarlosSalasSalasAún no hay calificaciones
- Taller Sobre Replicaciã - N, Transcripciã - N, Traduccion, Mitosis, Meiosis, Nomenclatura de Cromosomas y CariotiposDocumento5 páginasTaller Sobre Replicaciã - N, Transcripciã - N, Traduccion, Mitosis, Meiosis, Nomenclatura de Cromosomas y CariotiposEva Maria SolanoAún no hay calificaciones
- Genética Molecular 6Documento16 páginasGenética Molecular 6lu sanAún no hay calificaciones
- Tarea 1. Abeso MangueDocumento10 páginasTarea 1. Abeso ManguePedro Ernesto Abeso MangueAún no hay calificaciones
- Problemario DNA y RNADocumento3 páginasProblemario DNA y RNAJoel GomezAún no hay calificaciones
- Guia Biologia 4medioDocumento9 páginasGuia Biologia 4mediocammygenAún no hay calificaciones
- Caso 2 BCMDocumento5 páginasCaso 2 BCMCecilia CaiafaAún no hay calificaciones
- GBQ BM MAYO 2017-Tipo01finalDocumento9 páginasGBQ BM MAYO 2017-Tipo01finalAlberto BalbinAún no hay calificaciones
- Biotecnologia PDV PDFDocumento22 páginasBiotecnologia PDV PDFkarenyrodrigoforeverAún no hay calificaciones
- Problemas IIDocumento4 páginasProblemas IITort's Vincent DamonAún no hay calificaciones
- Taller Biotecnología UnalDocumento4 páginasTaller Biotecnología UnalRodolfo AriasAún no hay calificaciones
- Duplicación de ADN PDFDocumento4 páginasDuplicación de ADN PDFRuben Diaz DiazAún no hay calificaciones
- Guía GenéticaDocumento6 páginasGuía Genéticawilson rodriguezAún no hay calificaciones
- EnzimasDocumento72 páginasEnzimasJose MarioAún no hay calificaciones
- Ejercicios de Enzimas y Mapas de Restriccion23Documento8 páginasEjercicios de Enzimas y Mapas de Restriccion23jessieAún no hay calificaciones
- Informe 6 RFLPDocumento5 páginasInforme 6 RFLPZully FerrerAún no hay calificaciones
- Fragmento de OkasakiDocumento8 páginasFragmento de OkasakiYaritza Anzaldo PérezAún no hay calificaciones
- Taller Dogma CentralDocumento9 páginasTaller Dogma CentralMaria CorreaAún no hay calificaciones
- Extraccion de DNADocumento10 páginasExtraccion de DNACoello trinidad bartolo0% (2)
- Actividades GenéticaDocumento6 páginasActividades Genéticainfojordi.06Aún no hay calificaciones
- Ejercicios UD 4 y 5 VoluntariosDocumento3 páginasEjercicios UD 4 y 5 Voluntariosjulia rivero andurellAún no hay calificaciones
- Replicacion AdnDocumento4 páginasReplicacion AdnDAMARIS MARISLEY VELASQUEZ POMAAún no hay calificaciones
- Tema 12 - Genética Molecular I - Replicación y Transcripción Del ADNDocumento72 páginasTema 12 - Genética Molecular I - Replicación y Transcripción Del ADNCHARINI.-Aún no hay calificaciones
- Informe Caso 2 - DiagnósticoDocumento5 páginasInforme Caso 2 - Diagnósticovictor arrietaAún no hay calificaciones
- Unidad 3:: Relación Entre Expresión y Regulación GénicaDocumento47 páginasUnidad 3:: Relación Entre Expresión y Regulación GénicaMORELIA MONJE MORAAún no hay calificaciones
- S2 - Ácidos NucleicosDocumento5 páginasS2 - Ácidos NucleicosValentinaAún no hay calificaciones
- TALLER 3 - Replicación - 2024Documento3 páginasTALLER 3 - Replicación - 2024Paula Sophía CastiblancoAún no hay calificaciones
- Tema 4 Fundamentos Del Mapeo Genético - 14pgs Col PDFDocumento14 páginasTema 4 Fundamentos Del Mapeo Genético - 14pgs Col PDFFrancisco Diaz FAún no hay calificaciones
- TALLER 3 - ReplicaciónIyIIDocumento5 páginasTALLER 3 - ReplicaciónIyIIShin Hyun YooAún no hay calificaciones
- Actividad 2 - Adn y Replicación (Hecho)Documento23 páginasActividad 2 - Adn y Replicación (Hecho)Saioa VegaAún no hay calificaciones
- Presentación1 MolDocumento37 páginasPresentación1 MolDany CéspedesAún no hay calificaciones
- Tecnicas Moleculares para Replicación DNADocumento7 páginasTecnicas Moleculares para Replicación DNAMelissa GalanAún no hay calificaciones
- Clase 12Documento40 páginasClase 12alfredoAún no hay calificaciones
- Práctica No.3 "Simulación de Extracción de ADN, PCR, Electroforesis y Microarreglos Nivel Laboratorio in Silico"Documento7 páginasPráctica No.3 "Simulación de Extracción de ADN, PCR, Electroforesis y Microarreglos Nivel Laboratorio in Silico"Stephanie ChaparroAún no hay calificaciones
- Práctica 2 BQ-1 21-22Documento4 páginasPráctica 2 BQ-1 21-22mariaAún no hay calificaciones
- PRACTICA 1 Curso 21-22 PDFDocumento4 páginasPRACTICA 1 Curso 21-22 PDFmariaAún no hay calificaciones
- Texto PresentaciónDocumento7 páginasTexto PresentaciónmariaAún no hay calificaciones
- Caracterización de LysPBC4Documento11 páginasCaracterización de LysPBC4mariaAún no hay calificaciones
- Continua Fisio B1Documento9 páginasContinua Fisio B1mariaAún no hay calificaciones
- Preguntas Fisio ContinuaDocumento97 páginasPreguntas Fisio ContinuamariaAún no hay calificaciones
- C2 Prueba - Revisión Do Intento PDFDocumento1 páginaC2 Prueba - Revisión Do Intento PDFmariaAún no hay calificaciones
- Seminario 3 PDFDocumento9 páginasSeminario 3 PDFmariaAún no hay calificaciones
- C1 Prueba - Revisión Do Intento PDFDocumento1 páginaC1 Prueba - Revisión Do Intento PDFmariaAún no hay calificaciones
- Manual Pract GIIDocumento148 páginasManual Pract GIIromerilloAún no hay calificaciones
- Tecnicas de GenéticaDocumento27 páginasTecnicas de GenéticaE PrenssaAún no hay calificaciones
- Guia 2Documento10 páginasGuia 2Kasa Store 01Aún no hay calificaciones
- CN - 6° - CelulaDocumento37 páginasCN - 6° - CelulacelestinaAún no hay calificaciones
- Tema IIDocumento17 páginasTema IINinoska HernándezAún no hay calificaciones
- Práctica 7 (Defectos en La Fase Germinativa) Parte 1 Grupo 1Documento7 páginasPráctica 7 (Defectos en La Fase Germinativa) Parte 1 Grupo 1Yankuam AntunAún no hay calificaciones
- Cuestionario Transferencia GenesDocumento28 páginasCuestionario Transferencia GenesZuri ManriqueAún no hay calificaciones
- Biotecnologia Verde y MarronDocumento26 páginasBiotecnologia Verde y MarronJONATHAN ALEJANDRO VARGAS CERINZAAún no hay calificaciones
- Resolucion de Problemas 1Documento6 páginasResolucion de Problemas 1Yamilee NikoleAún no hay calificaciones
- CitoesqueletoDocumento38 páginasCitoesqueletoniniAún no hay calificaciones
- Optica, Acustica, Mecanica, Termodinamica, EtcDocumento8 páginasOptica, Acustica, Mecanica, Termodinamica, EtcMike GuzmanAún no hay calificaciones
- Quimica CatDocumento9 páginasQuimica CatLucas Miranda CassaniAún no hay calificaciones
- PCR Darwin SanchezDocumento1 páginaPCR Darwin SanchezCarlosGutierrezAún no hay calificaciones
- Evolución y Revolución TecnológicasDocumento2 páginasEvolución y Revolución TecnológicasCarlos Ortega & Adiela JiménezAún no hay calificaciones
- Sexado de Aves Con Ausencia de DismorfismoDocumento12 páginasSexado de Aves Con Ausencia de DismorfismoLuis Francisco JaimesAún no hay calificaciones
- Mapa Conceptual MitosisDocumento2 páginasMapa Conceptual MitosisNani brockAún no hay calificaciones
- Resumen Tema 5Documento4 páginasResumen Tema 5miriamAún no hay calificaciones
- Relaciones Entre GenesDocumento2 páginasRelaciones Entre GenesFaku MorenoAún no hay calificaciones
- Clase 2 Odont0Documento3 páginasClase 2 Odont0Elizabeth RysAún no hay calificaciones
- Biología 2°medioDocumento8 páginasBiología 2°mediomargarita niloAún no hay calificaciones
- Proyecto IntegradorDocumento7 páginasProyecto IntegradorAriadna LNAún no hay calificaciones
- Respuestas NatuDocumento12 páginasRespuestas Natujamer moreloAún no hay calificaciones
- La Forma de Los VirusDocumento1 páginaLa Forma de Los Virusdiana robles (slamduk307)Aún no hay calificaciones
- EXAMEN DEL SEGUNDO TRIMESTRE 2do SOLODocumento2 páginasEXAMEN DEL SEGUNDO TRIMESTRE 2do SOLOJúnior Zambrano100% (1)
- 6ta Tarea de BiologiaDocumento4 páginas6ta Tarea de Biologiachocolatecafe02Aún no hay calificaciones
- Biología en Agricultura y GanaderíaDocumento19 páginasBiología en Agricultura y GanaderíaJunior Rafael Ubiera Solano50% (4)
- Bacterias ClaseDocumento10 páginasBacterias ClaseYdnif Fabiana Rodriguez MoraAún no hay calificaciones
- Comparación Del Desarrollo Embrionario Temprano en Distintas EspeciesDocumento10 páginasComparación Del Desarrollo Embrionario Temprano en Distintas EspeciesMaria Jose NavaAún no hay calificaciones
- Microorganismos de Uso IndustrialDocumento4 páginasMicroorganismos de Uso IndustrialDavid OspinaAún no hay calificaciones