Documentos de Académico
Documentos de Profesional
Documentos de Cultura
Biología Molecular
Cargado por
nwg2303Derechos de autor
Formatos disponibles
Compartir este documento
Compartir o incrustar documentos
¿Le pareció útil este documento?
¿Este contenido es inapropiado?
Denunciar este documentoCopyright:
Formatos disponibles
Biología Molecular
Cargado por
nwg2303Copyright:
Formatos disponibles
Biología Molecular
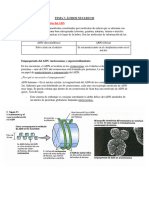
Tema 1 - Estructura de los ácidos nucleicos
Tema 2 - Genes y Genoma
Estructura de los ácidos nucleicos
1. Estructura Primaria
Cromosomas: Estructuras que contienen material genético, compuesto de ADN +
proteínas.
- ADN se encuentra en forma de doble hélice
- Cada una de las hebras que constituyen la
hélice está compuesta de nucleótidos
- Los nucleótidos están unidos mediante
enlace covalente fosfodiéster
- El enlace es fosfodiéster porque hay dos
enlaces fosfoesters en la molécula
- El enlace fosfodiéster une el grupo fosfato
de un nucleótido con el azúcar del
nucleótido adyacente (3´)
- Un nucleótido está constituido por un grupo fosfato
unido a una pentosa (en el caso del DNA es una
desoxirribosa) con una base nitrogenada (A, T, C y G)
unida a su vez a la pentosa.
- Nucleótidos de la misma hebra se unen siempre de la
misma forma, el fosfato 5' con el 3' hidroxilo del
siguiente nucleótido, estableciéndose siempre la misma
dirección cabeza-cola (5’->3’)
- -Nucleósido: base nitrogenada + azúcar
- -Nucleótido: base nitrogenada + azúcar + grupo fosfato
- -Enlace N-glucosídico: entre el C de la base nitrogenada y el azúcar.
- -Enlace fosfoéster: entre el carbono del azúcar y el oxígeno del grupo fosfato.
- -Enlace fosfodiéster: dos enlaces fosfoéster.
- Las dos hebras de DNA se mantienen juntas
gracias a la formación de puentes de H entre sus
bases nitrogenadas: A-T (dos enlaces) y C-G (tres
enlaces)
- Estas hebras son complementarias y antiparalelas.
- Bases púricas: A y G (dos anillos fusionados )
- Bases pirimidínicas: T, C y U (un anillo)
2. Reglas de Chargaff para ADN de doble hélice
•La proporción de Adenina (A) es igual a la de Timina (T). A = T. La relación entre
Adenina y Timina es igual a la unidad (A/T = 1).
• La proporción de Guanina (G) es igual a la de Citosina (C). G= C. La relación entre
Guanina y Citosina es igual a la unidad ( G/C=1).
• La proporción de bases púricas (A+G) es igual a la de las bases pirimidínicas (T+C).
(A+G) = (T + C). La relación entre (A+G) y (T+C) es igual a la unidad (A+G)/(T+C)=1.
• La proporción entre (A+T) y (G+C) es característica de cada organismo, pudiendo
tomar por tanto, diferentes valores según la especie estudiada. Este resultado indicaba
que los ácidos nucleicos no eran la repetición monótona de un tetranucleótido. Existía
variabilidad en la composición de bases nitrogenadas.
Si el genoma es monocatenario, esta regla no se cumple. (formado por una sola
cadena)
Normalmente la composición de bases de una molécula de ADN de un organismo se
expresa como su contenido de G+C. Se usan para calcular la Tm (temperatura de
fusión: la necesaria para desnaturalizar el ADN). Tiene aplicaciones para el diseño de
oligos para PCR.
2. Estructura secundaria del B-ADN
Rosalind Franklin:
Rosalind Franklin y Maurice Wilkins obtuvieron
mediante difracción de rayos X imágenes de alta
resolución de fibras de ADN, demostrando la forma B
del ADN que proporcionó evidencia de que el ADN tenía
una estructura helicoidal
(la forma B del ADN no es un tipo diferente de ADN,
sino simplemente una descripción de la estructura de
doble hélice)
- La forma B del DNA que se encuentra en disoluciones acuosas es una hélice regular
que hace un giro completo cada 3,4 nm (34 A).
- La distancia entre nucleótidos adyacentes es 0,34nm (3,4 A)
- Hay 10 nucleótidos por vuelta.
- Apertura (Distancia entre fosfato y azúcar de izquierda y derecha) es de 20
Armstrong
Surco mayor y menor:
- Carbono 1 de los azucares no se dispone simétricamente alineado a las bases por
lo cual aparecen surcos de tamaños desiguales en la hélice
- Como consecuencia de la asimetría de los pares de nucleotidos se forman surcos
mayor y menores
- T y A forman el surco mayor
- C y G forman el surco menor
- Surco mayor: Más ancho y profundo que el surco
menor, bases nitrogenadas menos protegidas facilitan el
acceso de proteínas (sitio de mayot interacción con
proteínas, Proteínas reconocen surco mayor para
silenciar cortaar etc.)
- Surco menor: Más estrecho y profundo, bases
nitrogenadas más protegidas y menos accesible para
proteínas (interacciones menos especificas y más
débiles con proteinas)
3. Variaciones en la estructura del ADN
B-ADN es la forma principal de ADN (cuando está en una solución acuosa)
La forma A-ADN (más comprimido) predomina en una disolución pobre en agua (ej
solución con mucha sal) (11 NUCLEOTIDOS POR VUELTA)
La forma Z (más estirada) presenta una rotación a izquierdas de la hélice (12
NUCLEOTIDOS POR VUELTA)
4. Variantes locales de la estructura secudaria del B-ADN
- Curvatura de la doble hélice: pequeñas diferencias locales en la estructura de los
DNAs que dependen de los nucleótidos integrantes. En otros casos, es forzada por
la unión de una proteína al DNA. Ej: El fago utiliza este motivo para unirse al DNA.
- Palíndromos: regiones del DNA donde la secuencia es la misma en ambas hebras y
con una simetría rotacional de 180o alrededor del palídromo. Dan lugar a
estructuras secundarias peculiares en el DNA y RNA. Las secuencias palindrómicas
son autocomplementarias.
- Suelen ser lugar de unión de las proteínas, (el ADN simboliza de esta forma dónde
tienen que suceder cosas):
- Sirven como lugares diana de enzimas de restricción que cortan al ADN (Enzimas de
restricción reconocen el bucle y hacen cortes).
- Lugares diana suelen ser secuencias específicas de bases en la molécula de ADN que
son reconocidas por proteínas específicas (Los bucles son sitios de reconocimiento
para las enzimas de restricción)
- Sirven como centros reguladores de la expresión génica (unión de factores de
transcripción)
- Las secuencias palindrómicas son autocomplementarias. Estos se forman de manera
espontánea durante la replicación. Un ejemplo lo vemos en el ARNt
(Cuando se dice que estas secuencias son "autocomplementarias", significa que la secuencia de bases en un lado de la molécula es complementaria a la
secuencia en el otro lado. En la estructura del ADN, esto significa que las bases adenina (A) se emparejan con timina (T) y las bases citosina (C) se emparejan con
guanina (G). Por lo tanto, si tienes una secuencia palindrómica en el ADN, su complemento será una réplica exacta de sí misma cuando se lee en sentido inverso)
Palíndromo en cadena sencilla se llama horquilla
Palindromo en doble hebra se llama cruciforme
H-DNA (triple hélice): estructura poco habitual
formada por 3 hebras. Se descubrió por primera
vez en RNA, pero es más frecuente en DNA y se
forma en regiones ricas en pirimidinas (CT) en
una hebra y en purinas (AG) en la
complementaria. La zona rica en CT se repliega
con la rica en AG mediante enlaces de Hoogsteen
(tipo especial de puentes de H). Son frecuentes
en los extremos de los cromosomas.
5. Motivos proteicos
Algunas estructuras tridimensionales se repiten en distintas proteínas y son
responsables de su interacción con secuencias específicas de DNA.
A estas estructuras se las denomina "motivos estructurales".
Son especialmente importantes en las proteínas llamadas factores de transcripción. Los
principales motivos responsables de la interacción con el DNA son:
Hélice-giro-hélice, Hélice-bucle-hélice, Homeodominio, Cremallera de leucina, Dedo de
zinc
- Los motivos proteicos forman parte de la estructura terciaria de la proteína e
interaccionan con la doble hélice del DNA, normalmente por el surco mayor.
- La interacción suele producir un cambio de conformación que provoca una
desnaturalización local, que favorece el acceso de otras proteínas
Motivo hélice-giro-hélice (helix-turn-helix): se encuentra en
proteínas reguladoras de expresión génica (en virus,
procariotas y eucariotas). Está formado por dos segmentos
peptídicos en α-hélice, de estructura rígida separados por un
giro (pocos aá). La hélice de reconocimiento encaja en el
surco mayor del DNA.
Motivo hélice-bucle-hélice (hélix-loop- hélix):
consta también de dos segmentos en α-hélice, pero en este caso el
péptido que lo une es más largo (más aá), con más posibilidades
de orientación de la hélice.
Motivo homeodominio (homeodomain):
Muchas repeticiones seguidas del motivo hélice-giro-hélice que
aparece repetidamente. Es importante en proteínas que regulan el
desarrollo embrionario, especialmente en Drosophila.
(La gran mayoría de los homeodominios reconocen un elemento
basal del DNA altamente conservado que sirve como promotor en
muchos genes (motivo TATA). Aminoácidos de interacción: Ser,
Arg, Asn. )
Motivo dedo de Zinc (zinc finger):
Frecuente en proteínas Eucariotas como el TFIIIA y el receptor de
estrógenos. Suelen observarse múltiples dedos de zinc
consecutivos. Está formado por 30 aas, de los cuales 2 Cys y 2 His
aparecen coordinadas tetraédricamente con un ion Zn 2+. Se
unen por enlaces de coordinación.
Motivo cremallera de leucina (leucine zipper):
Dos alfa hélices que tienen un residuo de Leu cada 7
aminoácidos (cada 2 vueltas de hélice). 2 cadenas de este tipo
se asocian hidrofóbicamente intercalando restos de Leu que
encajan en el surco mayor del DNA. Formación de una
estructura en forma de “Y”. Ej: proteínas reguladoras de la
transcripción bZIP.
6. Estructuras de orden superior. Superenrollamiento
El superenrollamiento explica como el DNA puede alojarse en el interior de la célula. La
torsión de la hélice B genera una Superhélice.
Superenrrollamiento positivo:
Se generan 2 vueltas más y en cada vuelta se pierde 0,5 pares de bases
22 vueltas de hélice
10 pares de bases por vuelta
Superenrrollamiento negativo:
Se generan 2 vueltas menos y en cada vuelta 0,5 pares de bases más
18 vueltas de hélice
11,1 pares de bases por vuelta
(DNA Relajado tiene 210 pares de bases, 10,5 por vuelta)
- Organización del cromosoma, explicando cómo el DNA puede
alojarse en el interior de la célula
- Se da en bacterias, en ADN de doble cadena circular. Mitocondrias
y cloroplastos
¿Qué hace que las moléculas de ADN en las células adopten conformaciones
superenrolladas?
Topoisomerasas: (Son distintas en procariotas y eucariotas)
- Topoisomerasa I (En células eucariotas y algunos procariotas)
Corta UNA hebra y pasar permitiendo que la hebra cortada gire alrededor de la
hebra intacta antes de volver a sellarla
Alivia el superenrollamiento negativo.
Resulta en la generación de una sola vuelta de superenrollamiento en el ADN
Se usa como diana terapéutica (antitumoral): la camptotecina impide el re-ligado
de la hebra de ADN cortada por la Top. 1
Aplicaciones de los inhibidores de las topoisomerasas: antitumorales
IRINOTECAN: se emplea en el cáncer colorrectal avanzado combinado con
5-Fluorouracilo en pacientes sin quimioterapia anterior o en monoterapia
para pacientes que han fracasado a un régimen anterior con 5-
Fluorouracilo
CAMPTOTECINA: interacciona con el complejo Topo I-DNA produciendo
citotoxicidad al impedir el re-ligando de la hebra de ADN cortada por la
TOP
(Tanto el Irinotecan, Topotecan, SN-38 son de síntesis artificial partiendo de la base
de la Camptotecina)
- Topoisomerasa II (En células eucariotas)
Cortan las DOS hebras
Resulta en la generación de multiples vueltas de superenrollamiento
Superenrolamiento positivo
Importante durante la replicación y transcripción del ADN
Se usa como diana terapéutica: los etopósidos (derivados sintéticos de podofilina)
se usan para impedir el religado en tratamiento de leucemias y cánceres.
- VEPESID: (Topoisomerasa II)
se usa en tumores testiculares, leucemias, cáncer microcítico de pulmón y
linfomas.
- TENIPOSIDO: Vumon (Topoisomerasa II)
Se emplea en algunos tipos de leucemias y linfomas asi como en los tumores
intracraneales malignos.
Inhibidoras de la topoisomerasa II como antimicrobianos:
También podría gustarte
- Introducción a la Biología: RESÚMENES UNIVERSITARIOSDe EverandIntroducción a la Biología: RESÚMENES UNIVERSITARIOSCalificación: 5 de 5 estrellas5/5 (1)
- ADN Estructura y FunciónDocumento11 páginasADN Estructura y FunciónPabloAún no hay calificaciones
- Acidos NucleicosDocumento6 páginasAcidos Nucleicospuchiti1234Aún no hay calificaciones
- Biologia CN 5to-2022Documento59 páginasBiologia CN 5to-2022joelcusmano2008Aún no hay calificaciones
- Ácidos NucléicosDocumento11 páginasÁcidos NucléicosJavier SosaAún no hay calificaciones
- Estructura DNADocumento8 páginasEstructura DNAsonia.alehdezAún no hay calificaciones
- Fármacos Que Interactúan Con Los Ácidos Nucleicos DainDocumento4 páginasFármacos Que Interactúan Con Los Ácidos Nucleicos DainYossellyIraholaMedinaAún no hay calificaciones
- Cuestionario AdnDocumento4 páginasCuestionario AdnLeonora PaelAún no hay calificaciones
- Acidos Nucleicos S1 2023 ModeloDocumento99 páginasAcidos Nucleicos S1 2023 Modelonahi pcAún no hay calificaciones
- Resumen Clases de Biología Molecular PDFDocumento50 páginasResumen Clases de Biología Molecular PDFGiselle ToscanoAún no hay calificaciones
- ADN y ARNDocumento18 páginasADN y ARNAnthonyAún no hay calificaciones
- Los Acidos NucleicosDocumento9 páginasLos Acidos NucleicoscalosraAún no hay calificaciones
- Ácido Desoxirribonucleico (Adn) : Propiedades Físicas y QuímicasDocumento4 páginasÁcido Desoxirribonucleico (Adn) : Propiedades Físicas y QuímicasMiguel Angel Rodas HerreraAún no hay calificaciones
- Semana 9 Acidos Nucleicos ReplicaciónDocumento16 páginasSemana 9 Acidos Nucleicos ReplicaciónShantal Ponce JáureguiAún no hay calificaciones
- Ácidos NucleicosDocumento7 páginasÁcidos NucleicosMARÍA CAMILA MORALES PERDOMOAún no hay calificaciones
- BCM - GenéticaDocumento52 páginasBCM - GenéticaCiber EspumitaAún no hay calificaciones
- Genomas estructura DNADocumento20 páginasGenomas estructura DNAMística Epul SotoAún no hay calificaciones
- Guia MedicinaDocumento13 páginasGuia MedicinaCarlos CalderwoodAún no hay calificaciones
- 17 PrimerasDocumento8 páginas17 PrimerasFernando RamiroAún no hay calificaciones
- Nucleótidos y AdnDocumento14 páginasNucleótidos y AdnRichivillAún no hay calificaciones
- Resumen Final Biol. Cel. y Molec.Documento211 páginasResumen Final Biol. Cel. y Molec.Hugo MedusAún no hay calificaciones
- Genetica Molecular 1. Replicacion y Sintesis de ProteinasDocumento6 páginasGenetica Molecular 1. Replicacion y Sintesis de ProteinasCarolina DávalosAún no hay calificaciones
- Guía Evaluada Paes 4 MedioDocumento4 páginasGuía Evaluada Paes 4 MedioBenjamín GutiérrezAún no hay calificaciones
- Ácidos NucleicosDocumento7 páginasÁcidos NucleicosmanonisghighiAún no hay calificaciones
- ADNDocumento12 páginasADNMisael Espinoza HuamánAún no hay calificaciones
- ADNDocumento5 páginasADNCAMILA GOGIN VASQUEZAún no hay calificaciones
- Taller 1 Acidos NucleicosDocumento3 páginasTaller 1 Acidos NucleicosMARY YOLANDA MARIACA PUYOAún no hay calificaciones
- 4 Acidos NucleicosDocumento8 páginas4 Acidos Nucleicoscyber pgAún no hay calificaciones
- Tema 20 y 21 IUDocumento12 páginasTema 20 y 21 IUWiam Kenzaoui EssalamaAún no hay calificaciones
- Estructura de Los Ácidos NucleicosDocumento5 páginasEstructura de Los Ácidos NucleicosYesid HernandezAún no hay calificaciones
- El ADN PDFDocumento3 páginasEl ADN PDFAndrés Cánovas LópezAún no hay calificaciones
- DNA recombinante y biología molecularDocumento22 páginasDNA recombinante y biología molecularMaria Cespedes Peres100% (1)
- Tema 5 - Los Nucleótidos y Los Ácidos NucleicosDocumento4 páginasTema 5 - Los Nucleótidos y Los Ácidos NucleicosMateoAún no hay calificaciones
- BioquimicaDocumento57 páginasBioquimicaDebra Lucia Cedeño TorresAún no hay calificaciones
- Tema 7. Acidos NucleicosDocumento37 páginasTema 7. Acidos NucleicosLaorden LucíaAún no hay calificaciones
- Estructura del DNADocumento22 páginasEstructura del DNAJonatan DuranAún no hay calificaciones
- Ácidos NucleicosDocumento15 páginasÁcidos NucleicosSir. ChyzioAún no hay calificaciones
- APUNTES OFICIALES TEMA 7 pdfDocumento18 páginasAPUNTES OFICIALES TEMA 7 pdfClara RoldánAún no hay calificaciones
- Biologia MolecularDocumento9 páginasBiologia MolecularMirna Aranda BarbozaAún no hay calificaciones
- Ácidos Nucleicos y Genoma HumanoDocumento5 páginasÁcidos Nucleicos y Genoma HumanoAngie CeronAún no hay calificaciones
- Tema 5 - Ácidos NucléicosDocumento10 páginasTema 5 - Ácidos NucléicosSiham ZebdaouiAún no hay calificaciones
- 05-Nucleótidos y Ácidos nucleicos-SEDocumento8 páginas05-Nucleótidos y Ácidos nucleicos-SETelmo Olivas SanchesAún no hay calificaciones
- Quím de Ácidos NucleicosDocumento10 páginasQuím de Ácidos NucleicosLizbeth Oyarce OrtizAún no hay calificaciones
- Biolomolo PDFDocumento66 páginasBiolomolo PDFCRISTINA DIAZ LOPEZAún no hay calificaciones
- Tema 25Documento8 páginasTema 25Elena ManchonAún no hay calificaciones
- Ac. Nucleicos 2018-19Documento34 páginasAc. Nucleicos 2018-19lukibrispalaciosAún no hay calificaciones
- El ADN y ARNDocumento3 páginasEl ADN y ARNjuana gregoria lara lazoAún no hay calificaciones
- Practica 5 Extraccion de ADNDocumento8 páginasPractica 5 Extraccion de ADNCésar AstudilloAún no hay calificaciones
- Estructura y conformaciones del ADNDocumento11 páginasEstructura y conformaciones del ADNAbi HernandezAún no hay calificaciones
- ENZIMOLOGIADocumento44 páginasENZIMOLOGIAPaola OlivosAún no hay calificaciones
- Acidos NucleicosDocumento2 páginasAcidos NucleicosGarmadilloAún no hay calificaciones
- Adn y ArnDocumento5 páginasAdn y ArnSandra AguirreAún no hay calificaciones
- Los NucleosomasDocumento9 páginasLos NucleosomasStevenSilvaGodoyAún no hay calificaciones
- Sistema CirculatorioDocumento47 páginasSistema CirculatorioRosario Huatuco MoronAún no hay calificaciones
- Ácidos NucleicosDocumento12 páginasÁcidos NucleicosIris LissiAún no hay calificaciones
- Biologia #1Documento4 páginasBiologia #1Beatriz De La Cruz100% (2)
- Apuntes de Biologia MolecularDocumento11 páginasApuntes de Biologia MolecularAngelica Carolina Del rosario MartinezAún no hay calificaciones
- Cuestionario Previo 5Documento7 páginasCuestionario Previo 5juanAún no hay calificaciones
- Acidos Nucleicos IIDocumento50 páginasAcidos Nucleicos IIAntonella SantaellaAún no hay calificaciones
- Acidosnucl 1679366456Documento8 páginasAcidosnucl 1679366456eduwin alejandroAún no hay calificaciones
- Discusión Elodea...Documento3 páginasDiscusión Elodea...Kevin HenaoAún no hay calificaciones
- Determinación de organofosforados en alimentos por cromatografía en capa finaDocumento12 páginasDeterminación de organofosforados en alimentos por cromatografía en capa finaOsbel Campos SandovalAún no hay calificaciones
- Exploración ExtrabucalDocumento4 páginasExploración ExtrabucalAna BelénAún no hay calificaciones
- Taller Gnética MendelianaDocumento8 páginasTaller Gnética MendelianavalentinaAún no hay calificaciones
- Guia Grado 6 Divison CelularDocumento20 páginasGuia Grado 6 Divison CelularMaria ParodiAún no hay calificaciones
- Genero y DelincuenciaDocumento46 páginasGenero y DelincuenciaRomeo RostinhuerAún no hay calificaciones
- El Hombre Como Visión Organicista o Como Visión MecanicistaDocumento3 páginasEl Hombre Como Visión Organicista o Como Visión MecanicistaCamila Orozco CamargoAún no hay calificaciones
- Plan Anual 2017 - Pueblo NuevoDocumento17 páginasPlan Anual 2017 - Pueblo NuevoZoila Calderon TrujilloAún no hay calificaciones
- Psicología de La EnvidiaDocumento8 páginasPsicología de La EnvidiaCarlos Ivan100% (2)
- Proceso de PartoDocumento5 páginasProceso de PartoPIERINA GERALDINE BENITES FARFANAún no hay calificaciones
- Cien 8 b3 p3 Est PDFDocumento10 páginasCien 8 b3 p3 Est PDFLuz Arley Hincapie MesaAún no hay calificaciones
- Vaso Dilatados Med TesisDocumento38 páginasVaso Dilatados Med TesisMaria Pacheco AlejosAún no hay calificaciones
- Análisis Bibliométrico - Aurum ApesDocumento3044 páginasAnálisis Bibliométrico - Aurum ApesJohan Esteban Romero De La CruzAún no hay calificaciones
- 2043-Texto Del Artículo-6005-1-10-20180913Documento13 páginas2043-Texto Del Artículo-6005-1-10-20180913Mirel LobAún no hay calificaciones
- Seminario de Edema 16195Documento129 páginasSeminario de Edema 16195Francesca VargasAún no hay calificaciones
- Manual Manipulador de Productos Fitosanitarios. Nivel Cualificado. Manual para El Alumno PDFDocumento410 páginasManual Manipulador de Productos Fitosanitarios. Nivel Cualificado. Manual para El Alumno PDFCarlos Delgado100% (1)
- Historia de La MicrobiologíaDocumento6 páginasHistoria de La MicrobiologíaDiegoAún no hay calificaciones
- El Orador y Las Cualidades para Una Buena VozDocumento2 páginasEl Orador y Las Cualidades para Una Buena VozEdgar LemAún no hay calificaciones
- Percepcion y Tipos de EspacioDocumento6 páginasPercepcion y Tipos de EspacioRoberto Carlos Limaymanta NuñezAún no hay calificaciones
- Seg - Espec - Rodolfo Amado Arevalo Marcos PDFDocumento54 páginasSeg - Espec - Rodolfo Amado Arevalo Marcos PDFORFITAAún no hay calificaciones
- Teoría lingüística latinaDocumento23 páginasTeoría lingüística latinaDaniel GargueAún no hay calificaciones
- Bitacora 2Documento4 páginasBitacora 2Isabel MosqueraAún no hay calificaciones
- Sueño Adulto MayorDocumento4 páginasSueño Adulto MayorNicolas CastroAún no hay calificaciones
- Informe Educacion SanitariaDocumento68 páginasInforme Educacion SanitariaEvelynJhovannaAún no hay calificaciones
- Importancia de la célula vegetal en la agronomíaDocumento4 páginasImportancia de la célula vegetal en la agronomíaTita JuradoAún no hay calificaciones
- Adaptación Física Al EjercicioDocumento14 páginasAdaptación Física Al EjercicioMarian Nieto100% (1)
- La importancia de los manglares y el sistema excretor humanoDocumento7 páginasLa importancia de los manglares y el sistema excretor humanodaniela salgadoAún no hay calificaciones
- Cuestionario sobre vías metabólicas claveDocumento8 páginasCuestionario sobre vías metabólicas claveGrinch FixAún no hay calificaciones
- Informe Biologia SinteticaDocumento14 páginasInforme Biologia SinteticaAnita OrellanaAún no hay calificaciones
- Examen ReproducciónDocumento3 páginasExamen ReproducciónLuis mario GonzalesAún no hay calificaciones
- Batidos Verdes Depurativos y Antioxidantes: Aumenta tu Vitalidad con Smoothie Detox Durante 10 Días Para Adelgazar y Bajar de Peso: Aumenta tu vitalidad con smoothie detox durante 10 días para adelgazar y bajar de pesoDe EverandBatidos Verdes Depurativos y Antioxidantes: Aumenta tu Vitalidad con Smoothie Detox Durante 10 Días Para Adelgazar y Bajar de Peso: Aumenta tu vitalidad con smoothie detox durante 10 días para adelgazar y bajar de pesoCalificación: 5 de 5 estrellas5/5 (2)
- En busca de la mente: El largo camino para comprender la vida mental (y lo que aún queda por descubrir)De EverandEn busca de la mente: El largo camino para comprender la vida mental (y lo que aún queda por descubrir)Calificación: 4.5 de 5 estrellas4.5/5 (3)
- Genética general: Libro de textoDe EverandGenética general: Libro de textoCalificación: 4.5 de 5 estrellas4.5/5 (11)
- Resumen de Pensar rápido pensar despacio de Daniel KahnemanDe EverandResumen de Pensar rápido pensar despacio de Daniel KahnemanCalificación: 4.5 de 5 estrellas4.5/5 (11)
- 50 técnicas de mindfulness para la ansiedad, la depresión, el estrés y el dolor: Mindfulness como terapiaDe Everand50 técnicas de mindfulness para la ansiedad, la depresión, el estrés y el dolor: Mindfulness como terapiaCalificación: 4 de 5 estrellas4/5 (37)
- Sesgos Cognitivos: Una Fascinante Mirada dentro de la Psicología Humana y los Métodos para Evitar la Disonancia Cognitiva, Mejorar sus Habilidades para Resolver Problemas y Tomar Mejores DecisionesDe EverandSesgos Cognitivos: Una Fascinante Mirada dentro de la Psicología Humana y los Métodos para Evitar la Disonancia Cognitiva, Mejorar sus Habilidades para Resolver Problemas y Tomar Mejores DecisionesCalificación: 4.5 de 5 estrellas4.5/5 (13)
- El autismo: Reflexiones y pautas para comprenderlo y abordarloDe EverandEl autismo: Reflexiones y pautas para comprenderlo y abordarloCalificación: 4 de 5 estrellas4/5 (7)
- Terapia de vidas pasadas: Un camino hacia la luz del alma. Técnica y prácticaDe EverandTerapia de vidas pasadas: Un camino hacia la luz del alma. Técnica y prácticaCalificación: 4.5 de 5 estrellas4.5/5 (11)
- Diccionario de neurociencias: Aplicadas a organizaciones y personasDe EverandDiccionario de neurociencias: Aplicadas a organizaciones y personasCalificación: 5 de 5 estrellas5/5 (4)
- GuíaBurros Análisis clínicos: Todo lo que necesitas saber para entender tus análisisDe EverandGuíaBurros Análisis clínicos: Todo lo que necesitas saber para entender tus análisisCalificación: 4 de 5 estrellas4/5 (9)
- Abrázame fuerte: Siete conversaciones para un amor duraderoDe EverandAbrázame fuerte: Siete conversaciones para un amor duraderoManu BerásteguiCalificación: 4.5 de 5 estrellas4.5/5 (13)
- Plan De Dieta Para La Resistencia A La Insulina & Sistema Inmunológico En EspañolDe EverandPlan De Dieta Para La Resistencia A La Insulina & Sistema Inmunológico En EspañolCalificación: 3.5 de 5 estrellas3.5/5 (2)
- Neuroanatomía: Fundamentos de neuroanatomía estructural, funcional y clínicaDe EverandNeuroanatomía: Fundamentos de neuroanatomía estructural, funcional y clínicaCalificación: 4 de 5 estrellas4/5 (16)
- El cerebro del niño explicado a los padresDe EverandEl cerebro del niño explicado a los padresCalificación: 4.5 de 5 estrellas4.5/5 (147)
- 200 tareas en terapia breve: 2ª ediciónDe Everand200 tareas en terapia breve: 2ª ediciónCalificación: 4.5 de 5 estrellas4.5/5 (33)
- Bases biológicas del comportamiento animal y humanoDe EverandBases biológicas del comportamiento animal y humanoCalificación: 4 de 5 estrellas4/5 (4)
- Mujeres Con Trastornos Por Déficit De Atención: Cómo aceptar sus diferencias y transformar su vidaDe EverandMujeres Con Trastornos Por Déficit De Atención: Cómo aceptar sus diferencias y transformar su vidaCalificación: 3.5 de 5 estrellas3.5/5 (3)
- Manual técnico de refrigerantesDe EverandManual técnico de refrigerantesCalificación: 4 de 5 estrellas4/5 (4)
- Anatomía del caballo: Guía práctica ilustradaDe EverandAnatomía del caballo: Guía práctica ilustradaCalificación: 4 de 5 estrellas4/5 (9)
- Despeja Tu Mente: Como Dejar De Pensar Demasiado, Vencer A Tu Crítico Interno, Y Replantear Tus Pensamientos Negativos Con Hábitos SaludablesDe EverandDespeja Tu Mente: Como Dejar De Pensar Demasiado, Vencer A Tu Crítico Interno, Y Replantear Tus Pensamientos Negativos Con Hábitos SaludablesAún no hay calificaciones
- Fisiología renal y metabolismo hidrosalino: Segunda ediciónDe EverandFisiología renal y metabolismo hidrosalino: Segunda ediciónAún no hay calificaciones
- Medicina con plantas sagradas: La sabiduría del herbalismo de los aborígenes norteamericanosDe EverandMedicina con plantas sagradas: La sabiduría del herbalismo de los aborígenes norteamericanosCalificación: 4 de 5 estrellas4/5 (10)