Documentos de Académico
Documentos de Profesional
Documentos de Cultura
Diagrama de La Detección de Colonias Bacterianas Con X-Gal e Iptg
Diagrama de La Detección de Colonias Bacterianas Con X-Gal e Iptg
Cargado por
Kenia PantojaDescripción original:
Título original
Derechos de autor
Formatos disponibles
Compartir este documento
Compartir o incrustar documentos
¿Le pareció útil este documento?
¿Este contenido es inapropiado?
Denunciar este documentoCopyright:
Formatos disponibles
Diagrama de La Detección de Colonias Bacterianas Con X-Gal e Iptg
Diagrama de La Detección de Colonias Bacterianas Con X-Gal e Iptg
Cargado por
Kenia PantojaCopyright:
Formatos disponibles
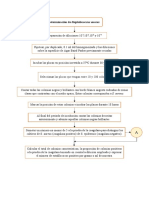
Procedimiento que se utiliza para identificar plásmidos recombinantes
DETECCIÓN DE COLONIAS mediante complementación alfa. El sustrato cromogénico X-gal se mezcla con
BACTERIANAS CON X-GAL
LISISE el cultivo bacteriano, se combina con agar superior fundido y luego se esparce
IPTG: COMPLEMENTACIÓN
ALCALINA en placas de selección. Con recursos limitados, el volumen requerido de X-gal
con SDS puede ser esparcido sobre una placa de agar (protocolo alterno).
ALFA
9.-Seleccionar y cultivar colonias que porten
plásmidos recombinantes.
1.-Disperse alícuotas (de 3 ml para placas de 90
mm y de 7 ml para placas de 150 ml) de agar
superior fundido en tubos de 17 x 100 mm.
8.-Identificar las colonias que portan
1.5.-Coloque los tubos en un bloque plásmidos recombinantes.
calefactor a 45°C hasta que sean necesarios.
7.-Saque las placas de la incubadora y
2.-Retire el primer tubo y agregue 0.1 ml de guárdelas durante varias horas a 4°C, para
suspensión bacteriana con <3000 bacterias permitir que se desarrolle el color azul.
viables para una placa de 90 mm y <10,000 para
una de 150 mm.
6.-Endurecer agar sofi a temperatura ambiente.
2.5.-Cierre la parte superior del tubo e Limpiar la tapa de las placas e incubar las placas
inviértalo varias veces para dispersar las invertidas (12-16 horas a 37°C).
bacterias a través del agar fundido.
5.-Repetir los pasos 2 a 4 hasta que todas las
3.-Abra el tubo y agregue cantidades apropiadas de muestras se hayan plaqueado.
X-gal e IPTG (si es necesario).
3.5.-Cierre el tubo e inviértalo suavemente
4.- Verter rápido el agar superior fundido en el
varias veces para mezclar el contenido.
centro de una placa de agar endurecido con el
antibiótico apropiado y distribuir girando.
PROTOCOLO ALTERNATIVO: Preparación alternativa de agar esparciendo una solución
LISIS concentrada de X-gal en la superficie de una placa de agar
APLICACIÓN DIRECTA DE X-
ALCALINA
GAL E IPTG A PLACAScon
AGAR prefabricada, en lugar de incorporar el galactósido
SDS
halogenado en todo el volumen del medio de agar. Tener cuidado al esparcir la
solución X-gal. Las colonias en
el centro de la placa pueden
ser de un azul más profundo
1.-Pipetear 40 µl de X-gal al 2% y, si es necesario, 7
debido a variaciones en la
µl de IPTG al 20% en el centro de una placa de agar concentración de X-gal a través
de 90 mm prefabricada (LB o YT) con el antibiótico. de la placa.
1.5.-Para una placa de agar de 15 cm de
diámetro, transfiera 100 µl de X-gal y, si es
necesario, 20 µl de IPTG al centro de la placa.
6.-Identificar las colonias que portan plásmidos
recombinantes.
2.-Con un esparcidor estéril (o pipeta Pasteur
doblada con punta sellada en una llama) extender las
soluciones por toda la superficie de la placa e
incubar a 37°C hasta que se absorba todo el líquido.
2.5.-Debido a la baja volatilidad de la 5.-Saque la placa de la incubadora y guárdela a
dimetilformamida, este proceso puede tardar 4°C durante varias horas, durante las cuales el
hasta 3-4 horas si la placa está recién hecha. color azul se desarrolla en toda su extensión.
3.-Inocular placa con las bacterias a analizar;
sembrando con un asa bacteriana, colocando clones 4.- Una vez absorbido el inóculo, incubar la placa
con palillos de dientes o esparciendo hasta 100 µl de en posición invertida durante 12-19 horas a 37°C.
una suspensión bacteriana (50.000 celis/ml) sobre un
plato de agar de 90 mm o 200 µl en plato de 15 cm.
Reseña de Método
Detección de colonias bacterianas con X-gal e IPTG: complementación alfa
Este protocolo presenta métodos para identificar plásmidos recombinantes mediante
complementación alfa. El sustrato cromogénico X-gal se mezcla con el cultivo bacteriano, se
combina con agar superior fundido y luego se esparce en placas de selección. Si los recursos son
limitados, el volumen requerido de X-gal puede ser Esparcir sobre una placa de agar (protocolo
alterno con aplicación directa de X-gal e IPTG a las placas de agar).
IPTG (isopropil-β-D-tiogalactósido) es un análogo no fermentable de lactosa que inactiva el
represor lacZ y, por tanto, induce la transcripción del operón lac. La mayoría de las cepas de
bacteria comúnmente utilizadas (complementación alfa), sin embargo, no sintetiza cantidades
significativas de represor lac. En consecuencia, normalmente no hay necesidad de inducir la
síntesis de los fragmentos de β-galactosidasa codificados por el huésped y el plásmido para el
análisis histoquímico de colonias bacterianas. Si la cepa bacteriana lleva el alelo del represor lac
y/o si el plásmido lleva un gen lacl, debería usarse IPTG para inducir la síntesis de ambos
fragmentos de la enzima.
La eficiencia de transformación es ligeramente mayor cuando las bacterias se colocan en placas
de agar superior en lugar de en la superficie de las placas de agar. Quizás las bacterias
transformadas prefieren el estado ligeramente anaeróbico con el agar blando o la isosmolaridad
proporcionada por el medio de agar.
NOMBRE: PANTOJA GAMEZ KENIA IVETH
NÚMERO DE CUENTA: 15219828
GRADO-GRUPO:3-1
MATERIA: INGENIERIA GENETICA
FECHA: 20/12/2020
BIBLIOGRAFÍA:
Sambrook J. & Green M. R., (2001). Molecular Cloning: A Laboratory Manual (Vol. 1).
Cold Spring Harbor Laboratory Press. Protocol 27: Screening Bacterial Colonies Using X-
gal and IPTG: alfa-Complementation
También podría gustarte
- Ejercicios UT 7Documento2 páginasEjercicios UT 7Ignacio Blagodyr100% (1)
- Tema 7 InteractomicaDocumento184 páginasTema 7 InteractomicaSoniaGarrichuAún no hay calificaciones
- BBM Resultados 20-21Documento18 páginasBBM Resultados 20-21Dina ISMAEL BARDÁNAún no hay calificaciones
- Metodo de Doble SeleccionDocumento3 páginasMetodo de Doble SeleccionCarolinaAún no hay calificaciones
- Practica Transformacion Bacteriana 2022Documento7 páginasPractica Transformacion Bacteriana 2022PIERO JOSE MIRANDA ALVINOAún no hay calificaciones
- Bitacora AgaresDocumento7 páginasBitacora AgaresLUISA FERNANDA PINILLA HERNANDEZAún no hay calificaciones
- Electroforesis Gel AgarosaDocumento4 páginasElectroforesis Gel AgarosaPablo RodriguezAún no hay calificaciones
- Protocolo DiatomeasDocumento9 páginasProtocolo DiatomeassaulblancoAún no hay calificaciones
- Aislamiento de MicroorganismosDocumento9 páginasAislamiento de MicroorganismosJoselyn KarinaAún no hay calificaciones
- Busqueda FallasDocumento19 páginasBusqueda FallasAnalli AcoltAún no hay calificaciones
- Practico IIIDocumento8 páginasPractico IIIRosalía PattyAún no hay calificaciones
- Practica 1Documento6 páginasPractica 1johana itzel Villeda RuizAún no hay calificaciones
- Esquema Practica N°6 Ashly ToroDocumento7 páginasEsquema Practica N°6 Ashly ToroASHLY TOROAún no hay calificaciones
- Manual de Biologia Molecular UCSMDocumento61 páginasManual de Biologia Molecular UCSMSoany M. Alanoca CoaquiraAún no hay calificaciones
- Unidad 3. Citogenética y Análisis CromosómicoDocumento7 páginasUnidad 3. Citogenética y Análisis CromosómicoLauraAún no hay calificaciones
- Southern BlotDocumento5 páginasSouthern BlotMireyaAún no hay calificaciones
- Crema y Azul Minimalista Monótono Negocio Informe de Avances de Los ODS - 20231217 - 192424 - 0000Documento6 páginasCrema y Azul Minimalista Monótono Negocio Informe de Avances de Los ODS - 20231217 - 192424 - 0000Emmanuel BernalAún no hay calificaciones
- Transformación E. Coli Con GFPDocumento11 páginasTransformación E. Coli Con GFPViktoryia RybakovaAún no hay calificaciones
- Prácticas de InmunologíaDocumento16 páginasPrácticas de InmunologíaCecilia Núñez de AndrésAún no hay calificaciones
- Vicente Torres, Pilar. Recuento de Enterobacterias Totales.Documento9 páginasVicente Torres, Pilar. Recuento de Enterobacterias Totales.toniberruezoAún no hay calificaciones
- Conservacion de Cepas Bacterianas ReferencialesDocumento5 páginasConservacion de Cepas Bacterianas ReferencialesEdu ReyesAún no hay calificaciones
- Género Streptococcus, EnterococcusDocumento12 páginasGénero Streptococcus, EnterococcusBárbara MansillaAún no hay calificaciones
- HemolisisDocumento7 páginasHemolisisKatherine Lemus SepúlvedaAún no hay calificaciones
- Manual de Prácticas de Bioquímica IIDocumento22 páginasManual de Prácticas de Bioquímica IIJonathan Islas0% (1)
- Materiales y MétodosDocumento4 páginasMateriales y MétodosIsäbellä CuellarAún no hay calificaciones
- Practico Semana 4 F.B IIDocumento32 páginasPractico Semana 4 F.B IIJhôn Töô WëēdAún no hay calificaciones
- Gonzalez Iglesias Veronica BMC04 TareaDocumento10 páginasGonzalez Iglesias Veronica BMC04 TareaVeronica Gonzalez IglesiasAún no hay calificaciones
- TEMA 13 Univ Wara Aguilar LuisDocumento6 páginasTEMA 13 Univ Wara Aguilar LuisPablo David EspinozaAún no hay calificaciones
- INEN 1529-14-2014 Staphylococcus Aureus, Recuento en PlacaDocumento12 páginasINEN 1529-14-2014 Staphylococcus Aureus, Recuento en PlacaEsther Cortez PazmiñoAún no hay calificaciones
- Practica de Laboratorio 4Documento11 páginasPractica de Laboratorio 4Ivonne RiveraAún no hay calificaciones
- Esporas y ColoraciónDocumento3 páginasEsporas y ColoraciónJorge MartinezAún no hay calificaciones
- Procedimiento para Enterobacter y Pseudomona en AguasDocumento4 páginasProcedimiento para Enterobacter y Pseudomona en AguasDeivis HernandezAún no hay calificaciones
- Procedimiento para Enterobacter y Pseudomona en AguasDocumento4 páginasProcedimiento para Enterobacter y Pseudomona en AguasDeivis HernandezAún no hay calificaciones
- 48 Transformación e Coli Con Plásmido RecombinanteDocumento5 páginas48 Transformación e Coli Con Plásmido RecombinantePatricia Gaete Ceballos100% (1)
- Protocolo Dario Extracción ADN de Fagos Feb 2023Documento2 páginasProtocolo Dario Extracción ADN de Fagos Feb 2023bryan guachiAún no hay calificaciones
- CoproparasitoscopicoDocumento43 páginasCoproparasitoscopicoGwen SM86% (14)
- Informe de MicroDocumento11 páginasInforme de MicroOmar Antonio Saenz CentenoAún no hay calificaciones
- Pruebas Fenotípicas CNADocumento4 páginasPruebas Fenotípicas CNAMikeAún no hay calificaciones
- StaphylococcusDocumento7 páginasStaphylococcusPaulette MendozaAún no hay calificaciones
- DF S Aureus 1Documento2 páginasDF S Aureus 1luzAún no hay calificaciones
- Electroforesis Guía de LaboratorioDocumento2 páginasElectroforesis Guía de LaboratorioFrancisco Mallea CatalánAún no hay calificaciones
- Deteccion Pseudomonas Aureginosa AguaDocumento6 páginasDeteccion Pseudomonas Aureginosa AguaMaria Josefa Henriquez Sanhueza100% (1)
- Preparación de Muestra de PCRDocumento7 páginasPreparación de Muestra de PCRKathi ÁlvarezAún no hay calificaciones
- Practica de PCR. Biologia Molecular AplicadaDocumento10 páginasPractica de PCR. Biologia Molecular AplicadaPedro AnguianoAún no hay calificaciones
- APHA Traducción Alicyclobacillus Concentrado de FrutasDocumento4 páginasAPHA Traducción Alicyclobacillus Concentrado de FrutasSORY SANCHEZ CARDOZAAún no hay calificaciones
- Pruebas de Identificacion MicrobiologicaDocumento65 páginasPruebas de Identificacion MicrobiologicaCarla Hernandez AracenaAún no hay calificaciones
- Prácticas de Laboratorio 3Documento10 páginasPrácticas de Laboratorio 3BRYAN ALEXANDER MOPOSITA TOAPANTAAún no hay calificaciones
- Biot BM 20161102 PPDocumento9 páginasBiot BM 20161102 PPJocelyne Calderon PadillaAún no hay calificaciones
- Screenshot 2022-06-07 at 12.30.27Documento16 páginasScreenshot 2022-06-07 at 12.30.27Jhoe Jauregui RudasAún no hay calificaciones
- NEISSERIADocumento14 páginasNEISSERIAmarina marinAún no hay calificaciones
- Aglutinación y PrecipitaciónDocumento49 páginasAglutinación y Precipitaciónlisy bolañosAún no hay calificaciones
- Informe Practica de Laboratorio Virtual MicrobiologíaDocumento3 páginasInforme Practica de Laboratorio Virtual MicrobiologíaAngelica VargasAún no hay calificaciones
- Extracción de RNAmDocumento5 páginasExtracción de RNAmLaura Daiana AlcarazAún no hay calificaciones
- APNB 32017 Recuento Aeróbios MesófilosDocumento5 páginasAPNB 32017 Recuento Aeróbios MesófilosRafael RiosAún no hay calificaciones
- AOAC 966.23. EspañolDocumento2 páginasAOAC 966.23. EspañolMaria Eugenia GuayacundoAún no hay calificaciones
- Practica de Staphylococcus.1Documento6 páginasPractica de Staphylococcus.1GINA PAOLA PALACIOS JAUREGUIAún no hay calificaciones
- Protocolo de Extraccion de ADN Bacteriano Kit Promega EspañolDocumento4 páginasProtocolo de Extraccion de ADN Bacteriano Kit Promega EspañolTatiana Paola Jiménez OchoaAún no hay calificaciones
- ProtocoloDocumento4 páginasProtocoloJeniffer ChaAún no hay calificaciones
- El Método Hanahan para La Preparación y Transformación Baterias EcoliDocumento4 páginasEl Método Hanahan para La Preparación y Transformación Baterias EcoliDavidAún no hay calificaciones
- Practica - ElectroforesisDocumento8 páginasPractica - ElectroforesisClaudia Romina Castañeda BulnesAún no hay calificaciones
- StaphylococcusDocumento5 páginasStaphylococcusHilder Baldeon NuñezAún no hay calificaciones
- Alimentos Del FuturoDocumento3 páginasAlimentos Del FuturoKenia PantojaAún no hay calificaciones
- Aljanabi-Diagrama de FlujoDocumento3 páginasAljanabi-Diagrama de FlujoKenia PantojaAún no hay calificaciones
- Biorreactor Tanque AgitadoDocumento2 páginasBiorreactor Tanque AgitadoKenia PantojaAún no hay calificaciones
- BiorremediacionDocumento3 páginasBiorremediacionKenia PantojaAún no hay calificaciones
- Esquema de Proceso Biotecnologico.Documento1 páginaEsquema de Proceso Biotecnologico.Kenia PantojaAún no hay calificaciones
- Sindrome de LaronDocumento7 páginasSindrome de LaronKenia PantojaAún no hay calificaciones
- 1.1 Hist de La Nut y GenéticaDocumento21 páginas1.1 Hist de La Nut y GenéticaKenia PantojaAún no hay calificaciones
- Articulo Subrayado y Traducido-BioseparacionDocumento9 páginasArticulo Subrayado y Traducido-BioseparacionKenia PantojaAún no hay calificaciones
- Resumen BTECDocumento32 páginasResumen BTECLautaro SanchezAún no hay calificaciones
- Clonaje Del ADNDocumento4 páginasClonaje Del ADNSeries HugoAún no hay calificaciones
- Genetica Bacteriana. Tema 16 VERSIÓN LARGADocumento68 páginasGenetica Bacteriana. Tema 16 VERSIÓN LARGAMagdalena Oñate SerranoAún no hay calificaciones
- Apunts Enginyeria GenèticaDocumento47 páginasApunts Enginyeria GenèticaXavier100% (1)
- Practica 7Documento8 páginasPractica 7KARLA LOPEZ BALDERASAún no hay calificaciones
- Ingeniería Genética en Procariotas y Análisis EstructuralDocumento36 páginasIngeniería Genética en Procariotas y Análisis EstructuralRaquel Pérez FernándezAún no hay calificaciones
- 22 05 16 Ingeniería GenéticaDocumento19 páginas22 05 16 Ingeniería Genéticaangeles Hernandez EspinozaAún no hay calificaciones
- Recombinación ADNDocumento3 páginasRecombinación ADNAna.Aún no hay calificaciones
- Reporte de Práctica 1. PCR. Ing GenéticaDocumento30 páginasReporte de Práctica 1. PCR. Ing GenéticaEunice Hernández GaleanaAún no hay calificaciones
- DBC FolletoDocumento2 páginasDBC Folletoapi-3697331Aún no hay calificaciones
- Práctica N - 6. Caracterización Fenotípica y Física de ADN RecombinanteDocumento10 páginasPráctica N - 6. Caracterización Fenotípica y Física de ADN RecombinanteMiguelYGracielaAún no hay calificaciones
- Genetica Primer ParcialDocumento104 páginasGenetica Primer ParcialCarolina Perez SanchezAún no hay calificaciones
- 48 Transformación e Coli Con Plásmido RecombinanteDocumento5 páginas48 Transformación e Coli Con Plásmido RecombinantePatricia Gaete Ceballos100% (1)
- Apuntes Ingenieria Genetica MolecularDocumento49 páginasApuntes Ingenieria Genetica MolecularAdriiana Bareas BouAún no hay calificaciones
- X GalDocumento3 páginasX GalManu CanoAún no hay calificaciones
- Clase Fago Lambda y Otros2Documento56 páginasClase Fago Lambda y Otros2Andrea Cardozo100% (6)
- Ingeniería Genética en Procariotas y Análisis EstructuralDocumento37 páginasIngeniería Genética en Procariotas y Análisis EstructuralRaquel Pérez FernándezAún no hay calificaciones
- Topicos de MicrobiologiaDocumento3 páginasTopicos de MicrobiologiaFABIANACHVAún no hay calificaciones
- Guia Problemas BM LicBiotecnologia 2018-Parte IDocumento17 páginasGuia Problemas BM LicBiotecnologia 2018-Parte IGuapomonAún no hay calificaciones
- Clonación de Un Fragmento de DnaDocumento19 páginasClonación de Un Fragmento de DnaXhavii BlAún no hay calificaciones
- Herramientas de La Ingeniería GenéticaDocumento19 páginasHerramientas de La Ingeniería GenéticaEDILMAHERNANDEZAún no hay calificaciones
- TallerDocumento3 páginasTallerChristian Paredes100% (1)
- Biología de Plásmidos. Dic 2020Documento30 páginasBiología de Plásmidos. Dic 2020Bryan Antonio Maza CordovaAún no hay calificaciones
- Determinar Contaminantes Quimicos Presentes en La Miel Producida en La Comunidad Autónoma de AragónDocumento132 páginasDeterminar Contaminantes Quimicos Presentes en La Miel Producida en La Comunidad Autónoma de AragónMelivea Paez HerediaAún no hay calificaciones
- PlásmidosDocumento13 páginasPlásmidosRodrigo WolfgonAún no hay calificaciones
- Metodo de Doble SeleccionDocumento3 páginasMetodo de Doble SeleccionCarolinaAún no hay calificaciones
- ADN RecombinanteDocumento28 páginasADN RecombinanteCARLOS ANDRÉS DODINOAún no hay calificaciones