Documentos de Académico
Documentos de Profesional
Documentos de Cultura
6 Mantenimiento Del Centrómero en La Replicación Del ADN
Cargado por
AsasTítulo original
Derechos de autor
Formatos disponibles
Compartir este documento
Compartir o incrustar documentos
¿Le pareció útil este documento?
¿Este contenido es inapropiado?
Denunciar este documentoCopyright:
Formatos disponibles
6 Mantenimiento Del Centrómero en La Replicación Del ADN
Cargado por
AsasCopyright:
Formatos disponibles
Traducido del inglés al español - www.onlinedoctranslator.
com
noticias y vistas
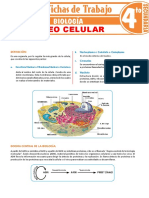
CICLO CELULAR
Mantenimiento del centrómero durante la replicación del ADN.
La identidad del centrómero debe mantenerse a través de múltiples generaciones. Un nuevo estudio revela una retención dependiente de la red
asociada a centrómeros constitutivos (CCAN) de CENP-A, una marca epigenética clave para los centrómeros, en los centrómeros durante la
replicación del ADN y una corrección de errores dependiente de la replicación para eliminar la CENP-A ectópica en los brazos cromosómicos.
Masatoshi Hara y Tatsuo Fukagawa
C
los cromosomas duplicados durante la fase S Centrómero brazo cromosómico
deben dividirse por igual en células hijas CENP-A CCAN (CENP-C)
durante la fase M. Una región importante de
H3 preexistente GRAMO1
la cromatina para la segregación cromosómica
H3 recién depositado
precisa en la división celular es el centrómero, que
proporciona una plataforma para establecer el horquilla de replicación
cinetocoro, un gran CCAN (CENP-C)
complejo proteico para unir los cromosomas y los
microtúbulos del huso1. En numerosas especies
eucariotas, incluidos los humanos, el centrómero está Dependiente de CENP-C
S reciclaje
presente en un lugar específico del cromosoma.2. La
pérdida o ganancia de centrómero conduce a la mala
segregación y rotura de los cromosomas, lo que da
como resultado aneuploidía y trastornos genéticos
posteriores, incluido el cáncer. Los centrómeros
humanos abarcan regiones de megapares de bases
en un locus particular en cada cromosoma2. Las
regiones centroméricas humanas contienen matrices GRAMO2
complejas de repetición de orden superior (HOR) de
satélites α2. Sin embargo, la secuencia repetida del
satélite α no es esencial ni suficiente para la identidad Retención CENP-A centromérica Eliminación ectópica de CENP-A
del centrómero en células humanas.1,2. En cambio, una
Figura 1 |Retención centromérica de CeNP-a y eliminación ectópica de CeNP-a durante la replicación del ADN. Durante la fase
variante de histona H3 específica del centrómero,
S, los nucleosomas preexistentes en la cromatina duplicada son desalojados por la horquilla de replicación. Los CENP-A desalojados
CENP-A, juega un papel fundamental en la
en centrómeros que contienen CCAN (CENP-C) se vuelven a depositar en hebras nacientes de una manera dependiente de CENP-C
especificación del centrómero, sirviendo como una
(reciclado dependiente de CENP-C); por lo tanto, CENP-A se retiene en la cromatina centromérica después de la replicación del ADN.
marca epigenética importante.1,2. CENP-A forma un
Los sitios depositados por CENP-A durante la fase S son los mismos que en G1fase. Por el contrario, después del desalojo por la
nucleosoma octámero con histonas canónicas (H4,
horquilla de replicación, las CENP-A incorporadas ectópicamente en el brazo cromosómico no se vuelven a depositar en la cromatina
H2A y H2B), que recluta un subcomplejo de cinetocoro
naciente durante la replicación del ADN. La retención de CENP-A en los centrómeros y la eliminación de CENP-A en los brazos
llamado CCAN1. La cromatina CENP-A debe
cromosómicos durante la replicación del ADN aseguran el mantenimiento de la identidad del centrómero a través de múltiples
conservarse a través de múltiples divisiones celulares
generaciones.
para mantener la identidad del centrómero.
Deposición de CENP-A de novo en los centrómeros a
principios de G1fase está mediada por su chaperona
específica HJURP en células humanas1. Sin embargo,
no está claro cómo se mantienen los nucleosomas eliminar CENP-A mal incorporado en brazos modelos de referencia, Nechemia-Arbely et al.4
preexistentes que contienen CENP-A en los cromosómicos no centroméricos (Fig.1). identificó secuencias precisas de unión a CENP-A en
centrómeros durante la replicación del ADN, ya que la Los nucleosomas que contienen CENP-A centrómeros humanos. Aislaron mononucleosomas
maquinaria de replicación del ADN requiere la representan ~4% de la región del centrómero CENP-A de células HeLa que expresan CENP-A
eliminación de los nucleosomas existentes para la humano5. Sin embargo, la posición precisa de marcado con localización y purificación por afinidad
síntesis del ADN.3. En la edición actual deBiología unión a CENP en los centrómeros aún no está (LAP) a niveles endógenos o niveles elevados de CENP-
celular de la naturaleza, Nechemia-Arbely et al.4 clara, ya que los centrómeros humanos A marcado con purificación por afinidad en tándem
demostrar que CCAN juega un papel fundamental en comprenden largas matrices de ADN de satélite α (TAP) sincronizado en G1o g2Fase y lectura de
la recarga precisa del CENP-A centromérico, que se casi idénticos, lo que impide los análisis de todo el secuencias de ADN unidas a nucleosomas CENP-A.
desaloja durante la progresión de la horquilla de genoma en los centrómeros. El progreso reciente Debido a que los modelos de referencia de
replicación, y que la replicación del ADN proporciona en tecnología de secuenciación y algoritmos ha centrómero incluyen variaciones de secuencia en
un sistema de corrección de errores para ayudado a generar modelos de referencia de matrices HOR, las lecturas de secuencia se asignaron
centrómeros para 23 cromosomas humanos: 22 correctamente a cada matriz HOR. Primero, el satélite
autosomas (incorporados en GRCh38) y el α más activo
cromosoma X6,7. Utilizando el
naturaleza biología celular|VOL 21 | JUNIO 2019 | 665–673 |www.nature.com/naturecellbiology 669
noticias y vistas
las matrices se enriquecen para la ocupación Eliminación de CENP-A y el momento de la Usando un análisis de secuencia completo de los
CENP-A (niveles de expresión endógena) de 20 a replicación del ADN, Nechemia-Arbely et al.4 ADN de unión a CENP-A con modelos de referencia de
40 veces4. Diecisiete centrómeros comprenden combinó la secuenciación de ADN de unión a centrómeros humanos recientes, Nechemia-Arbely et
múltiples conjuntos de satélites α, entre los cuales CENP-A con Repli-seq, que mapea secuencias de al.4demuestran la retención de CENP-A dependiente
seis centrómeros contienen solo un conjunto que hebras de replicación de ADN naciente en células de CENP-C dentro de los centrómeros durante la
se une activamente a CENP-A4. Los 11 centrómeros sincronizadas para determinar regiones de replicación del ADN y la eliminación dependiente de la
restantes tienen dos o más arreglos que están replicación temprana, media y tardía. replicación de ADN de CENP-A ectópico en los brazos
ocupados por CENP-A4, consistente con el hallazgo Demostraron que más del 90% de los sitios de cromosómicos (Fig.1). Estos mecanismos reguladores
de centrómeros en el cromosoma 17, donde CENP- unión ectópica de CENP-A en el junto con la deposición de CENP-A de novo por HJURP
A se une a diferentes matrices en cada homólogo8. GRAMO1Las células de la fase se replicaron en la fase a principios de G1fase permite la herencia estable de la
Además, los niveles elevados de expresión de S temprana o media, y los sitios de unión a CENP-A identidad del centrómero a través de múltiples
CENP-A no cambiaron el número de sitios de unión mapeados ectópicamente se eliminaron poco generaciones, en consonancia con nuestro estudio
de CENP-A, pero mejoraron su acumulación en después de la replicación del ADN, lo que respalda anterior que informa que CCAN retiene las posiciones
cada sitio en los centrómeros.4, apoyando así el firmemente la participación de la horquilla de del centrómero en las células DT40 de pollo, que
modelo de acción de masas CENP-A propuesto replicación en la eliminación de CENP-A ectópico en contienen centrómeros no repetitivos en algunos
anteriormente5. los brazos cromosómicos.4(Higo.1). A diferencia de la cromosomas12. Estos estudios sugieren que el
La deposición de CENP-A de novo por HJURP región del brazo, la secuencia de ADN centromérico mantenimiento de la identidad del centrómero en la
ocurre a principios de G1fase1,2, y los sitios de unión que contiene satélites α se replica en la fase S tardía y posición precisa es una función crucial de CCAN en la
a CENP-A en los centrómeros son estables a lo largo se detectó ocupación de CENP-A en los mismos sitios interfase, mientras que CCAN juega un papel clave en
de múltiples ciclos celulares. El número de en los centrómeros antes y después de la replicación el reclutamiento de complejos de unión a
nucleosomas CENP-A y el tamaño de la cromatina del ADN.4. Aunque el 10% de las secuencias de unión a microtúbulos para establecer una estructura de
centromérica permanecen sin cambios durante el CENP-A no centroméricas se replicaron en la fase S cinetocoro funcional en
ciclo celular. Debido a que las histonas en la tardía, el CENP-A depositado en estas regiones
cromatina duplicada son desalojadas y recargadas también se eliminó después de la replicación del ADN.
durante la replicación del ADN.3, Nechemia-Arbely et Esto sugiere que la retención de CENP-A en el fase M13. Curiosamente, otro estudio reciente
al.4 centrómero durante la replicación del ADN está muestra que CENP-A expulsado por la horquilla de
examinó cómo la replicación del ADN afecta la mediada por un mecanismo específico en lugar de un replicación se retiene en la cromatina
distribución de CENP-A en los centrómeros usando simple evento de fase S tardía.4(Higo.1). centromérica por MCM2 y HJURP.14. Dado que la
células sincronizadas en G1o g2fase. Su análisis de retención de CENP-A en los centrómeros depende
satélite α de copia única utilizando células con niveles de CENP-C durante la replicación del ADN4, una
elevados de CENP-A reveló que casi todos los sitios Para comprender cómo se retiene CENP-A en pregunta clave posterior que debe abordarse es
centroméricos únicos unidos a CENP-A se mapearon la cromatina centromérica durante la replicación cómo se vincula el sistema MCM2-HJURP con
en G1fase se retuvieron en G2fase, y la abundancia de del ADN, Nechemia-Arbely et al.4 CENP-C en este proceso. El dímero canónico de la
CENP-A en cada sitio también se mantuvo antes y nucleosomas CENP-A aislados de células en fase S histona H3-H4 forma un complejo con MCM2 y
después de la replicación del ADN4. Las células que tardía e identificaron sus proteínas interactuantes. ASF1, una chaperona H3-H4, generando así un
expresan CENP-A a niveles endógenos también Estos incluyen las subunidades p150 y p60 de CAF-1, modelo en el que MCM2 retiene H3-H4 expulsado
mostraron perfiles de unión de CENP-A sin cambios un complejo de chaperonas de histonas que media la por la horquilla de replicación del ADN y funciona
dentro de los centrómeros.4. Estos datos sugieren un deposición de histonas acoplada a la replicación.3, y con ASF1 para recargar los antiguos complejos H3-
mecanismo para la retención de CENP-A en los componentes del complejo de helicasa replicativa H4 en cromatina replicada10. Los datos
centrómeros. MCM2-7, que desempeña un papel clave en el bioquímicos sugieren que los dímeros CENP-A–H4
reciclaje de histonas durante la progresión de la se unen simultáneamente a MCM2 y HJURP4,14. Sin
Además, Nechemia-Arbely et al.4 horquilla de replicación3,10. Por lo tanto, Nechemia- embargo, HJURP se une no solo a la hélice α2 de
identificó los sitios de unión a CENP-A asignados a Arbely et al.4proponen que el reciclaje de CENP-A CENP-A sino también a la hélice α1, que también
brazos cromosómicos no centroméricos, en específico del centrómero junto con la replicación del se prevé que se una a MCM2 (refs.10,15),
consonancia con estudios previos5,9: 11.390 y 40.279 ADN está mediado por los complejos CAF-1 y MCM2-7. aumentando la posibilidad de colisión entre MCM2
sitios en G1-Células en fase que expresan CENP-A a Porque los nucleosomas que contienen CENP-A y HJURP en CENP-A. Por otro lado, MCM2 puede
niveles endógenos y elevados, respectivamente. Estos también están asociados con CCAN que se localiza en unirse a HJURP de manera independiente de las
sitios de unión a CENP-A ectópicos en los brazos los centrómeros a lo largo del ciclo celular.1, los histonas.10. Por lo tanto, una comprensión precisa
cromosómicos se superponen con marcas de autores plantearon la hipótesis de que CCAN involucra de cómo interactúan CENP-A–H4, MCM2 y HJURP
transcripción activas de modificaciones de histonas y a CENP-A para el reciclaje de CENP-A específico de es un desafío importante. Además, a veces se
sitios hipersensibles a la ADNasa I, lo que sugiere que centrómero. Entre los componentes de CCAN, forma un nuevo centrómero en un sitio ectópico
el CENP-A ectópico se carga preferentemente para Nechemia-Arbely et al.4 en el brazo del cromosoma como un
abrir la cromatina activa en los brazos cromosómicos.4, neocentrómero.2. Sería interesante investigar
como se observó anteriormente para la mala se centró particularmente en CENP-C, dada su cómo la región del neocentrómero evade el
localización de CENP-A dependiente de DAXX (proteína interacción con el nucleosoma que contiene CENP-A mecanismo de corrección de errores dependiente
asociada al dominio de la muerte) en líneas celulares y la proteína CENP-B de unión a satélite α11. De de la replicación del ADN en el brazo
de cáncer4,9. Sin embargo, los picos CENP-A no hecho, el rápido agotamiento de CENP-C en la fase S cromosómico.
centroméricos identificados en G1-Las células de fase temprana comprometió la retención de CENP-A en
no se detectaron en G2-células en fase, lo que indica la las regiones centroméricas durante la replicación del
eliminación de la ocupación ectópica no centromérica ADN. Esto sugiere que CENP-C juega un papel crítico -
CENP-A después de la replicación del ADN4(Higo.1). en la retención centromérica de CENP-A,
Para investigar más a fondo la asociación entre contribuyendo así a la herencia centrómero en masatoshi hara *y
hebras hermanas de ADN naciente.4(Higo.1). Tatsuo Fukagawa *
Escuela de Graduados en Biociencias Fronterizas, Osaka
670 naturaleza biología celular|VOL 21 | JUNIO 2019 | 665–673 |www.nature.com/naturecellbiology
noticias y vistas
Universidad, Suita, Osaka, Japón. 2. Fukagawa, T. y Earnshaw, WCdesarrollo Celúla30, 496–508 10. Huang, H. et al.Nat. Estructura. mol. Biol.22, 618–626
(2014). (2015).
* correo electrónico:mahara@fbs.osaka-u.ac.jp;
3. Anuncio, ATbioquimica Biografía. acta1819, 196– 11. Hoffmann, S. et al.Informes de celda17, 2394–2404 (2016).
tfukagawa@fbs. osaka-u.ac.jp 210 (2013). 12. Hori, T. et al.J. Cell Biol.216, 101–113 (2017).
4. Nechemia-Arbely, Y. et al.Nat. Biol celular.https://doi.org/10.1038/ 13. Hara, M., Ariyoshi, M., Okumura, E.-I., Hori, T. y Fukagawa, T. Nat.
Publicado en línea: 3 de junio de 2019 https:// s41556-019-0331-4(2019). Biol celular.20, 1378–1388 (2018).
5. Bodor, DL et al.eLife3, e02137 (2014). 14. Zasadzińska, E. et al.desarrollo Celúla47, 348–362.e7 (2018).
doi.org/10.1038/s41556-019-0335-0
6. Miga, KH et al.Genoma Res.24, 697–707 (2014). 15. Hu, H. et al.Genes Dev.25, 901–906 (2011).
7. Schneider, VA et al.Genoma Res.27, 849–864 (2017).
Referencias 8. Maloney, KA et al.proc. Academia Nacional. ciencia EE.UU
1. Quesero, MIHarb de primavera fría. Perspectiva. Biol.6,
Conflicto de intereses
109, 13704–13709 (2012).
a015826 (2014). Los autores declaran no tener conflictos de intereses.
9. Lacoste, N. et al.mol. Celúla53, 631–644 (2014).
CÉLULAS MADRE
Un impulso hacia la totipotencia de las células madre
Las células madre embrionarias derivadas de la masa celular interna pueden diferenciarse en todos los linajes embrionarios de cualquier tipo de
célula adulta y, hasta cierto punto, en tejidos extraembrionarios. Ahora, un estudio permite la generación de células madre porcinas y humanas
con un mayor potencial de diferenciación hacia todos los destinos embrionarios y extraembrionarios, un paso más cerca del estado totipotente del
óvulo fertilizado.
Fred Etoc y Ali Brivanlou
W
Cuando se disocian las células de un Células denominadas 'pluripotencialidad expandida' células madre de potencial expandido (mEPSC),
embrión temprano, se pueden mantener fueron descubiertas recientemente7. En este número de que se pueden colocar entre el estado similar a 2C
en cultivo bajo un ambiente estable y Biología celular de la naturaleza, Gao et al. ampliar este y el estado ingenuo. De acuerdo con este
estado pluripotente proliferativo. El descubrimiento hallazgo para generar células madre de potencial posicionamiento único dentro del espectro de
inicial de las células madre embrionarias (ESC) en el expandido (EPSC) porcino y humano, proporcionando un estados pluripotentes, las mEPSC podrían dar
sistema murino1seguido por la derivación de sus enfoque general para derivar estas células madre de lugar a linajes tanto embrionarios como
contrapartes humanas2condujo a una explosión en el otras especies de mamíferos8. extraembrionarios por diferenciación in vitro o
campo de la biología ESC. Sin embargo, el embrión es Durante las etapas previas a la implantación del cuando se formaron quimeras por reintroducción
muy dinámico, y el hecho de que sus células puedan desarrollo embrionario, todas las células en blastocistos.7.
aislarse y mantenerse en una etapa de desarrollo experimentan una serie de transiciones y finalmente Tras estos descubrimientos, la siguiente
estable y congelada sigue siendo un hallazgo se segregan dentro de la población embrionaria, el pregunta era si el estado pluripotente mejorado
sorprendente. Aunque las ESC humanas y de ratón se epiblasto o uno de los dos destinos es universal y si pueden alcanzarlo células madre
derivan de embriones en etapas similares, se extraembrionarios: el trofoblasto y el hipoblasto. de otras especies. En este sentido, las células
observan en cultivo dentro de dos estados Dependiendo del posicionamiento temporal de sus madre porcinas son un desafío interesante dado
pluripotentes diferentes asociados con diferentes estados pluripotentes con respecto a esas que no se han derivado ESC de buena fe. Sin
requisitos de señalización.3. Las ESC de ratón se transiciones en la línea de tiempo del desarrollo, embargo, debido a las diferencias entre especies,
califican como "ingenuas" y coinciden con la masa algunos ESC pierden la capacidad de dar lugar de es poco probable que el medio desarrollado
celular interna (ICM) del blastocisto antes de la manera eficiente a destinos extraembrionarios y anteriormente para crear mEPSCs7se traduciría
implantación, mientras que las ESC humanas se pueden diferenciarse preferentemente hacia destinos directamente al modelo porcino. Por lo tanto,
asemejan a una etapa posterior del desarrollo del embrionarios cuando se reimplantan en el entorno para optimizar las condiciones de cultivo, Gao et
epiblasto, después de la implantación pero justo embrionario.9(Higo.1). En comparación con las células al. no partió de embriones de ocho células, sino
antes de la gastrulación, en un estado "preparado".4. preparadas, las células vírgenes pueden contribuir a que desarrolló una ingeniosa plataforma de
Otros estudios han identificado condiciones de los animales quiméricos de manera mucho más detección a partir de células madre pluripotentes
señalización para que los ESC cambien de un estado eficiente cuando se implantan en blastocistos.10, pero inducidas porcinas (iPSC)8. La expresión de los
a otro5e intentó devolverlos a la etapa de desarrollo ni los ESC ingenuos ni preparados pueden contribuir factores de Yamanaka bajo el control de la
totipotente más temprana del embrión en etapa de a los linajes derivados del trofoblasto (p. ej., la doxiciclina (dox) en fibroblastos porcinos condujo
dos células6(similar a 2C). placenta)11. Para resolver esta limitación, un estudio a su reprogramación y a la creación de iPSC
anterior aisló blastómeros de embriones de ratón en dependientes de dox. La eliminación de dox
Dado el potencial de la investigación de ESC en etapa de ocho células para establecer células condujo a la inhibición de la expresión de los
biotecnologías y el uso de organismos modelo de pluripotentes que podrían diferenciarse hacia linajes factores de Yamanaka y una rápida pérdida de
mamíferos para estudiar enfermedades, se requiere embrionarios y extraembrionarios.7. Luego, dados los pluripotencia. Usando esta herramienta básica y
de manera crítica el desarrollo de métodos para componentes conocidos de la ruta de señalización de una receta de cultivo inicial con su medio mEPSC
derivar de manera confiable ESC de diferentes la transición ICM-trofoblasto, desarrollaron un previamente optimizado, los autores buscaron
especies de mamíferos bajo un conjunto común de conjunto de condiciones para bloquear condiciones de cultivo que mantuvieran las iPSC
condiciones que dan lugar a estados similares y sistemáticamente estas señales y generar murino porcinas pluripotentes tras la eliminación de dox.
potentes de pluripotencia. . En este contexto, un estable. En última instancia, Gao et al.8
estado pluripotente para ratón
naturaleza biología celular|VOL 21 | JUNIO 2019 | 665–673 |www.nature.com/naturecellbiology 671
También podría gustarte
- Acceso a Universidad para Mayores de 25 años. Biología 2013-2017.: Solucionario Pruebas 2013-2017De EverandAcceso a Universidad para Mayores de 25 años. Biología 2013-2017.: Solucionario Pruebas 2013-2017Aún no hay calificaciones
- Clase 4 Unidad 3 - ReplicaciónDocumento41 páginasClase 4 Unidad 3 - ReplicaciónLeilaAún no hay calificaciones
- Replicacion Del ADN 26.03.2023Documento12 páginasReplicacion Del ADN 26.03.2023GabrielaAún no hay calificaciones
- Proyectos 5tos 2023: Proyecto TODocumento22 páginasProyectos 5tos 2023: Proyecto TOSlandyAún no hay calificaciones
- Nucleo Celular - Replicacion 2021-IIDocumento28 páginasNucleo Celular - Replicacion 2021-IIKaren FelixAún no hay calificaciones
- BIOLOGIA-ADN (01-06-2021) TareaDocumento5 páginasBIOLOGIA-ADN (01-06-2021) TareaEmanuel HFAún no hay calificaciones
- Práctica 4Documento8 páginasPráctica 4Antonella Ortiz100% (1)
- Adn, ArnDocumento2 páginasAdn, ArnIsabella AladinoAún no hay calificaciones
- Evaluación TranscripciónDocumento2 páginasEvaluación TranscripciónDIANA CAROLINA POSADA LINARESAún no hay calificaciones
- Preguntas y EjerciciosDocumento3 páginasPreguntas y EjerciciosMilena CruzAún no hay calificaciones
- Ciclo intensivo Biología UNALMDocumento5 páginasCiclo intensivo Biología UNALMdayroAún no hay calificaciones
- 5 SeminarioDocumento2 páginas5 Seminariocrashito247Aún no hay calificaciones
- Dogma central de la biología molecularDocumento4 páginasDogma central de la biología molecularAndres FelipeAún no hay calificaciones
- Replicación: Síntesis de ADN: Biología Molecular y GenéticaDocumento34 páginasReplicación: Síntesis de ADN: Biología Molecular y GenéticaJosé CisternasAún no hay calificaciones
- Taller de Trascripcion y TraduccionDocumento7 páginasTaller de Trascripcion y TraduccionmcAún no hay calificaciones
- Actividad Complementaria Síntesis de ADNDocumento3 páginasActividad Complementaria Síntesis de ADNAndrea Paola Chaparro OrtizAún no hay calificaciones
- Cuestionario Unidad 4Documento15 páginasCuestionario Unidad 4muriel aravena makucAún no hay calificaciones
- Genética BacterianaDocumento55 páginasGenética BacterianaRoque JuncalAún no hay calificaciones
- Actividad - Genetica Molecular Noveno12Documento6 páginasActividad - Genetica Molecular Noveno12Alexandra Pedroza100% (1)
- C Sem10 BiologiaDocumento7 páginasC Sem10 BiologiaEG YasadiAún no hay calificaciones
- Segundo Bloque ThaliaDocumento6 páginasSegundo Bloque ThaliaMaria Fernanda SilvaAún no hay calificaciones
- Nucleo Celular para Cuarto Grado de SecundariaDocumento4 páginasNucleo Celular para Cuarto Grado de SecundariaGloria LorenzoAún no hay calificaciones
- Teoria Genetica MolecularDocumento153 páginasTeoria Genetica MolecularLucre CarnuccioAún no hay calificaciones
- Adn y ArnDocumento12 páginasAdn y Arnefrain juliaoAún no hay calificaciones
- Genética molecular: transcripción, replicación y expresión génicaDocumento9 páginasGenética molecular: transcripción, replicación y expresión génicaCardonaCristianAún no hay calificaciones
- Preicfes Nariño Con RespuestasDocumento9 páginasPreicfes Nariño Con RespuestasLuis Fernando Sanchez PucheAún no hay calificaciones
- Mendelclass Anatomia Celula 3 Nucleo y Ciclo Celular-17121020399Documento11 páginasMendelclass Anatomia Celula 3 Nucleo y Ciclo Celular-17121020399Claudio Galarzza FernándezAún no hay calificaciones
- GenéticaDocumento60 páginasGenéticaAguirre Gómez100% (1)
- Bmi TDocumento176 páginasBmi TJuan Arcos HuasasquicheAún no hay calificaciones
- Grupo 1 - Final Biología 15 - 02 - 2024Documento9 páginasGrupo 1 - Final Biología 15 - 02 - 2024themodmedbrAún no hay calificaciones
- PREICFES 9 Ciencias NaturalesDocumento3 páginasPREICFES 9 Ciencias NaturalesEstefania SalazarAún no hay calificaciones
- Genes, genoma y organización cromosómicaDocumento82 páginasGenes, genoma y organización cromosómicaDaniela TejadaAún no hay calificaciones
- Biología General - Semana Ix Expresión Génica - SemilleroDocumento5 páginasBiología General - Semana Ix Expresión Génica - SemilleroWilman IshpilcoAún no hay calificaciones
- Resumen Biologia CelularDocumento59 páginasResumen Biologia CelularCaru LitkeAún no hay calificaciones
- s3 Trascribiendo Genes en Plantas JDMMDocumento54 páginass3 Trascribiendo Genes en Plantas JDMMMax PlasenciaAún no hay calificaciones
- Clase 4. Replicacion Del ADNDocumento37 páginasClase 4. Replicacion Del ADNLuis Fabian Rosado PicazaAún no hay calificaciones
- Seminario BC 7-2020Documento68 páginasSeminario BC 7-2020Andrea Soledad PerezAún no hay calificaciones
- Cuestionario Sobre ADNDocumento1 páginaCuestionario Sobre ADNLibrofilo100% (2)
- Solemnes Biología MolecularDocumento12 páginasSolemnes Biología MolecularLeonidas LarrondoAún no hay calificaciones
- TALLER 04 Genetica Molecular 2017Documento2 páginasTALLER 04 Genetica Molecular 2017Taydith Teresa Sierra Tibamoza0% (1)
- Semana 10. Estructura Del ADN y ARN - GRADO 9Documento13 páginasSemana 10. Estructura Del ADN y ARN - GRADO 9Castro Abogados & AsesoresAún no hay calificaciones
- CROMATINADocumento3 páginasCROMATINASalomé EstévezAún no hay calificaciones
- Tema 2.1 Técnicas Citogenéticas y Moleculares.Documento6 páginasTema 2.1 Técnicas Citogenéticas y Moleculares.Cony CastañedaAún no hay calificaciones
- Acidos Nucleicos ADN y ARNDocumento3 páginasAcidos Nucleicos ADN y ARNLuis A. Romero0% (1)
- Evaluacion Sintesis de ProteinasDocumento1 páginaEvaluacion Sintesis de ProteinasRosiDanisAún no hay calificaciones
- SEMANA 10-Dogma CentralDocumento2 páginasSEMANA 10-Dogma CentralEduardo33% (3)
- Práctica 4 - Expresión Génica y Código GenéticoDocumento3 páginasPráctica 4 - Expresión Génica y Código GenéticoRuth ParedesAún no hay calificaciones
- Dogma Central 2018 EpigeneticaDocumento151 páginasDogma Central 2018 EpigeneticaOsmar Leonardo Gavilan SotoAún no hay calificaciones
- Expresión génica ANUAL UNIDocumento3 páginasExpresión génica ANUAL UNIDaniel TorresAún no hay calificaciones
- ACIDOS NUCLEICOS y TRANSMISION de La INFORMACION GENETICADocumento57 páginasACIDOS NUCLEICOS y TRANSMISION de La INFORMACION GENETICAJudith SheilaAún no hay calificaciones
- 4.1 Bases Físicas de La Herencia 1Documento25 páginas4.1 Bases Físicas de La Herencia 1Dario VelaAún no hay calificaciones
- Preguntas BiologiaDocumento6 páginasPreguntas BiologiaAndrea Pérez AriasAún no hay calificaciones
- Núcleo InterfásicoDocumento4 páginasNúcleo InterfásicoPatricia EncinaAún no hay calificaciones
- 6 - Mini Apunte Transcripción Y TraducciónDocumento12 páginas6 - Mini Apunte Transcripción Y TraducciónLeonardo BatistaAún no hay calificaciones
- Semana 07 - BiologíaDocumento4 páginasSemana 07 - BiologíaLuis Daniel Llontop TenorioAún no hay calificaciones
- Reconocimiento Unidad 2Documento6 páginasReconocimiento Unidad 2Manuel Ricardo Bayona PerezAún no hay calificaciones
- Semana 3.1-Dogma de La BiologiaaDocumento9 páginasSemana 3.1-Dogma de La Biologiaaaimipatogallina1234Aún no hay calificaciones
- MGB 1 - TeoríaDocumento32 páginasMGB 1 - TeoríaAgustin TuroneAún no hay calificaciones
- Genética MicrobianaDocumento31 páginasGenética MicrobianaPaula DanielaAún no hay calificaciones
- REPLICACIONDocumento21 páginasREPLICACIONElias CortezAún no hay calificaciones
- Reaccion Quimica Diferente de MezclaDocumento3 páginasReaccion Quimica Diferente de MezclaAsasAún no hay calificaciones
- Lab Microbiología Bajo CostoDocumento2 páginasLab Microbiología Bajo CostoAsasAún no hay calificaciones
- 02 Stem Tecno WebDocumento24 páginas02 Stem Tecno WebAsasAún no hay calificaciones
- 5-La Vida Al Interior Del Genoma-EnviadoDocumento8 páginas5-La Vida Al Interior Del Genoma-EnviadoBRYAN CAMILO MUÑOZ AVILESAún no hay calificaciones
- CONTABILIDAD BÁSICA - Practico 06 Ejercicios Sobre Registración en Libro DiarioDocumento4 páginasCONTABILIDAD BÁSICA - Practico 06 Ejercicios Sobre Registración en Libro DiarioDiego Soto MoyaAún no hay calificaciones
- Plantilla - Presentación - Diagrama V de GowinDocumento4 páginasPlantilla - Presentación - Diagrama V de GowinyurleyAún no hay calificaciones
- Catálogo Final Remate Luis GuzmánDocumento68 páginasCatálogo Final Remate Luis GuzmánLopecito LpdtAún no hay calificaciones
- Devocional BíblicoDocumento16 páginasDevocional BíblicoMartin Antonio Santa Cruz QuevedoAún no hay calificaciones
- Tañido de Una FlautaDocumento2 páginasTañido de Una FlautaSergio Alberto Cortés RonquilloAún no hay calificaciones
- Test PsicológicosDocumento5 páginasTest PsicológicosFilomena Andrea GonzálezAún no hay calificaciones
- Del Sexo A La Super Conciencia - OshoDocumento27 páginasDel Sexo A La Super Conciencia - Oshomiryamrugo100% (1)
- Exp1 Eba IntermedioDocumento12 páginasExp1 Eba IntermedioSANDRA ArticaAún no hay calificaciones
- Bujia Arte Contemporaneo 1Documento42 páginasBujia Arte Contemporaneo 1Osvaldo Hernández MuroAún no hay calificaciones
- Iacc Control Segundo SemestreDocumento5 páginasIacc Control Segundo SemestreHANS KURTAún no hay calificaciones
- 18 Concreto Sin FinosDocumento5 páginas18 Concreto Sin FinosEloy A. Cabrera ZuñigaAún no hay calificaciones
- Bernaza, Cammotto y LongoDocumento20 páginasBernaza, Cammotto y LongoIsabel PérezAún no hay calificaciones
- 12 Piedras CementadasDocumento15 páginas12 Piedras CementadasCesar O. Retamar RomeroAún no hay calificaciones
- Practica 1Documento7 páginasPractica 1Edith R ChAún no hay calificaciones
- Manual de organización y funciones DEMEMDocumento66 páginasManual de organización y funciones DEMEMjomer stevenAún no hay calificaciones
- VRMMP Auditorías MortalidadDocumento4 páginasVRMMP Auditorías MortalidadJordany Placencia0% (1)
- Trabajo Final DinamicaDocumento8 páginasTrabajo Final DinamicaNorbil Correa gilAún no hay calificaciones
- CLASIFICACIÓN DE PARÁSITOS SEGÚN NIVEL DE BIOSEGURIDADDocumento1 páginaCLASIFICACIÓN DE PARÁSITOS SEGÚN NIVEL DE BIOSEGURIDADDiego Erazo GirónAún no hay calificaciones
- Poster PDFDocumento1 páginaPoster PDFEstaban Ossa100% (1)
- Puntos y Planos Cefalometricos (Word)Documento4 páginasPuntos y Planos Cefalometricos (Word)cnvelasquezAún no hay calificaciones
- Componentes Del Servicio Al ClienteDocumento1 páginaComponentes Del Servicio Al ClienteSonia Pascual ChavezAún no hay calificaciones
- La Obra de Juan Ferrando Badía y Su Significación en El Desarrollo Del Derecho Constitucional y de La Ciencia Política en EspañaDocumento38 páginasLa Obra de Juan Ferrando Badía y Su Significación en El Desarrollo Del Derecho Constitucional y de La Ciencia Política en EspañaRenzzo Paolo Retes PaucarAún no hay calificaciones
- Los Booms Petroleros ¿Qué Cambió en Los Últimos 40 Años Foro Economía EcuadorDocumento1 páginaLos Booms Petroleros ¿Qué Cambió en Los Últimos 40 Años Foro Economía EcuadorGame DylánAún no hay calificaciones
- Corrientes SociopolíticasDocumento9 páginasCorrientes SociopolíticasJair Exzacarias Hernández100% (2)
- Agenda Lunes 25 Al 29 de OctubreDocumento5 páginasAgenda Lunes 25 Al 29 de OctubreGisela PeñaAún no hay calificaciones
- Reg - Aux.consol 1T 2 A2 AritDocumento6 páginasReg - Aux.consol 1T 2 A2 AritElder Junior Roman LeonAún no hay calificaciones
- Desplazamiento MiscibleDocumento34 páginasDesplazamiento MisciblePedro Reynaldo Marin Dominguez100% (1)
- Portafolio. Nuevos Modelos de NegociosDocumento27 páginasPortafolio. Nuevos Modelos de NegociosVictor CavazosAún no hay calificaciones
- Anexo 1 - Colaborativa-238Documento10 páginasAnexo 1 - Colaborativa-238vanesa telloAún no hay calificaciones
- Responsabilidad Por La Función Pública BoliviaDocumento31 páginasResponsabilidad Por La Función Pública BoliviaSoriaVargas Edwin95% (21)
- Neuroanatomía: Fundamentos de neuroanatomía estructural, funcional y clínicaDe EverandNeuroanatomía: Fundamentos de neuroanatomía estructural, funcional y clínicaCalificación: 4 de 5 estrellas4/5 (16)
- Notas de clase. Manual de farmacognosia: Análisis microscópico y fitoquímico, y usos de plantas medicinalesDe EverandNotas de clase. Manual de farmacognosia: Análisis microscópico y fitoquímico, y usos de plantas medicinalesAún no hay calificaciones
- Genética general: Libro de textoDe EverandGenética general: Libro de textoCalificación: 4.5 de 5 estrellas4.5/5 (11)
- Batidos Verdes Depurativos y Antioxidantes: Aumenta tu Vitalidad con Smoothie Detox Durante 10 Días Para Adelgazar y Bajar de Peso: Aumenta tu vitalidad con smoothie detox durante 10 días para adelgazar y bajar de pesoDe EverandBatidos Verdes Depurativos y Antioxidantes: Aumenta tu Vitalidad con Smoothie Detox Durante 10 Días Para Adelgazar y Bajar de Peso: Aumenta tu vitalidad con smoothie detox durante 10 días para adelgazar y bajar de pesoCalificación: 5 de 5 estrellas5/5 (2)
- Resumen de Pensar rápido pensar despacio de Daniel KahnemanDe EverandResumen de Pensar rápido pensar despacio de Daniel KahnemanCalificación: 4.5 de 5 estrellas4.5/5 (11)
- 50 técnicas de mindfulness para la ansiedad, la depresión, el estrés y el dolor: Mindfulness como terapiaDe Everand50 técnicas de mindfulness para la ansiedad, la depresión, el estrés y el dolor: Mindfulness como terapiaCalificación: 4 de 5 estrellas4/5 (37)
- Neuropsicología: Los fundamentos de la materiaDe EverandNeuropsicología: Los fundamentos de la materiaCalificación: 5 de 5 estrellas5/5 (1)
- Los cinco ritos tibetanos de la eterna juventud: El ojo de la revelaciónDe EverandLos cinco ritos tibetanos de la eterna juventud: El ojo de la revelaciónCalificación: 4.5 de 5 estrellas4.5/5 (12)
- Cerebros rotos: Pacientes asombrosos que me enseñaron a vivirDe EverandCerebros rotos: Pacientes asombrosos que me enseñaron a vivirCalificación: 5 de 5 estrellas5/5 (3)
- El cerebro del niño explicado a los padresDe EverandEl cerebro del niño explicado a los padresCalificación: 4.5 de 5 estrellas4.5/5 (147)
- Hormonas: Mensajeros químicos y comunicación celularDe EverandHormonas: Mensajeros químicos y comunicación celularCalificación: 2.5 de 5 estrellas2.5/5 (2)
- 200 tareas en terapia breve: 2ª ediciónDe Everand200 tareas en terapia breve: 2ª ediciónCalificación: 4.5 de 5 estrellas4.5/5 (33)
- Minerales y vitaminas: Micronutrientes esenciales en la alimentación, nutrición y salud.De EverandMinerales y vitaminas: Micronutrientes esenciales en la alimentación, nutrición y salud.Calificación: 3 de 5 estrellas3/5 (2)
- Abrázame fuerte: Siete conversaciones para un amor duraderoDe EverandAbrázame fuerte: Siete conversaciones para un amor duraderoManu BerásteguiCalificación: 4.5 de 5 estrellas4.5/5 (13)
- GuíaBurros Análisis clínicos: Todo lo que necesitas saber para entender tus análisisDe EverandGuíaBurros Análisis clínicos: Todo lo que necesitas saber para entender tus análisisCalificación: 4 de 5 estrellas4/5 (9)
- Sesgos Cognitivos: Una Fascinante Mirada dentro de la Psicología Humana y los Métodos para Evitar la Disonancia Cognitiva, Mejorar sus Habilidades para Resolver Problemas y Tomar Mejores DecisionesDe EverandSesgos Cognitivos: Una Fascinante Mirada dentro de la Psicología Humana y los Métodos para Evitar la Disonancia Cognitiva, Mejorar sus Habilidades para Resolver Problemas y Tomar Mejores DecisionesCalificación: 4.5 de 5 estrellas4.5/5 (13)
- El autismo: Reflexiones y pautas para comprenderlo y abordarloDe EverandEl autismo: Reflexiones y pautas para comprenderlo y abordarloCalificación: 4 de 5 estrellas4/5 (7)
- El perfil psicológico de Jesús: Aprendamos del Maestro a manejar efectivamente nuestras emocionesDe EverandEl perfil psicológico de Jesús: Aprendamos del Maestro a manejar efectivamente nuestras emocionesCalificación: 4 de 5 estrellas4/5 (4)
- Terapia de vidas pasadas: Un camino hacia la luz del alma. Técnica y prácticaDe EverandTerapia de vidas pasadas: Un camino hacia la luz del alma. Técnica y prácticaCalificación: 4.5 de 5 estrellas4.5/5 (11)
- Bases biológicas del comportamiento animal y humanoDe EverandBases biológicas del comportamiento animal y humanoCalificación: 4 de 5 estrellas4/5 (4)
- La familia: Comprensión dinámica e intervenciones terapéuticasDe EverandLa familia: Comprensión dinámica e intervenciones terapéuticasCalificación: 5 de 5 estrellas5/5 (1)
- Nuestra mente nos engaña: Sesgos y errores cognitivos que todos cometemosDe EverandNuestra mente nos engaña: Sesgos y errores cognitivos que todos cometemosCalificación: 3.5 de 5 estrellas3.5/5 (6)