Documentos de Académico
Documentos de Profesional
Documentos de Cultura
Análisis Conglomerados Mixturas
Análisis Conglomerados Mixturas
Cargado por
María Vargas MagánDescripción original:
Título original
Derechos de autor
Formatos disponibles
Compartir este documento
Compartir o incrustar documentos
¿Le pareció útil este documento?
¿Este contenido es inapropiado?
Denunciar este documentoCopyright:
Formatos disponibles
Análisis Conglomerados Mixturas
Análisis Conglomerados Mixturas
Cargado por
María Vargas MagánCopyright:
Formatos disponibles
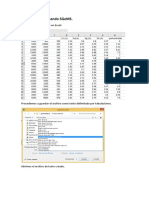
Análisis conglomerados mixturas.
Ver introducción en el HTML.
La selección del modelo adecuado se puede realizar según algún criterio que conjugue
verosimilitud y complejidad, como AIC o BIC. Cuanto mayor sea el BIC, mejor es la calidad del
conglomerado. BIC es el coeficiente que determina el mejor modelo. Usamos la librería
library(mclust)
La matriz “Sigma”, en modelos con varianzas iguales (EII,EEI, EEE) es la estimación de la matriz
de dispersión común.
Las matrices “sigma” son las matrices de covarianzas en cada grupo.
Ej.9. Ajustar un modelo de mixturas de normales bivariantes sobre los datos faithful de la
librería mclust, correspondiente a datos de tiempo de espera entre las erupciones y la
duración de la erupción del géiser Old Faithful en el Parque Nacional Yellowstone, Wyoming,
EE.UU.
library(mclust)
data(faithful)
Primero dibujamos el plot de los datos para saber si tiene sentido plantearse la
mixtura.
plot(faithful)# tiene sdo porque parece que tendremos dos
conglomerados
Búsqueda del mejor modelo de mixtura. Con la orden Mclust()
Mclusfaith=Mclust(faithful) #BUSCAR EL MEJOR MODELO
Mclusfaith
# Obtenemos model object: (EEE,3)
El segundo (3) nos dice que la muestra se puede ajustar por 3
normales bivariantes
Además, (EEE) nos dice que tenemos 3 matrices de dispersión y que
son iguales. La matriz es la misma para las tres componentes.
Describamos el objeto creado. (ayuda a entender lo anterior)
names(Mclusfaith)
Mclusfaith$modelName # mejor modelo seleccionado
Mclusfaith$n # Tamaño muestral
Mclusfaith$d # Dimensión del vector aleatorio
Sale que es 2, es decir, tenemos normales BIVARIANTES
Mclusfaith$G # Número óptimo de grupos
Mclusfaith$BIC #BIC para los modelos analizados, resaltando los tres
mejores modelos (notar que queremos MAXIMIZAR EL BIC)
Mclusfaith$bic # Valor BIC óptimo
Mclusfaith$loglik # Valor de la log-verosimilitud óptimo
Mclusfaith$df # Número de parámetros estimados
Parámetros estimados
Mclusfaith$parameters$pro # Vector con las probabilidades de la
mixtura estimadas
##[1] 0.1656784 0.3563696 0.4779520. Para la primera normal
bivariante, para la segunda, para la tercera.
Mclusfaith$parameters$mean # Estimación de la media de cada componente
Mclusfaith$parameters$variance #Estimación de la matriz de dispersión
de cada componente
Para cada caso, prob. de pertenencia a cada grupo. (AQUÍ
OBTENEMOS LOS Z)
matz<-Mclusfaith$z # Matriz que contiene para cada caso, la
probabilidad de pertenencia
round(matz[1:10,],4) #SON LOS Z. Son las probabilidades de
pertenencia a cada una de las k componentes de mixtura.
## [,1] [,2] [,3]
## 1 0.9718 0.0000 0.0282
## 2 0.0000 1.0000 0.0000
## 3 0.9967 0.0000 0.0032
## 4 0.0000 1.0000 0.0000
## 5 0.0117 0.0000 0.9883
## 6 0.0031 0.9969 0.0000
## 7 0.0030 0.0000 0.9970
## 8 0.9761 0.0000 0.0239
## 9 0.0000 1.0000 0.0000
## 10 0.0552 0.0000 0.9448
Por ejemplo, tenemos 10 casos. La prob de que el primer caso sea
del conglomerado 1 es 0.97, de que sea del 2 es 0, y de que sea
del 3 es 0.0282.
El plot del objeto nos dice qué cantidad de componentes hace que el bic sea mayor.
plot(Mclusfaith)
-en el primer plot vemos el número de conglomerados, distribuciones
-en el segundo, eruptions, vemos además elipses, que son RC para los
vectores de media de cada una de las normales.
-En el tercer dibujo observamos la incertidumbre. La incertidumbre es
1-Za, con la matriz de Z que vimos anteriormente. Si Z es alto, poca
incertidumbre.
-en el ultimo, tenemos regiones de densidad cte. Puede indicar si
existe un grupo separado o no.
Ej. 10. Ajustar un modelo de mixturas sobre los datos wreath de la librería mclust. Los
datos contienen 1000 observaciones realizadas de una mixtura normal bidimensional
de 14 componentes en las que las covarianzas de las componentes tienen el mismo
volumen o tamaño y forma, pero diferente orientación.
data(wreath)
plot(wreath) #14 componentes. Con el plot vemos si tiene sdo.
grid()
En este ejercicio usamos, la instrucción mclustBIC que guarda sólo los valores del
criterio BIC, y no las medias.
wreathBIC<- mclustBIC(wreath)
plot(wreathBIC,legendArgs=list(x="topleft"))
No somos capaces de obtener el numero de componentes que haga máximo el BIC.
Dado que el BIC se va optimizando conforme aumentamos el número de
componentes, ampliamos este número, es decir, conviene extender la búsqueda del
mejor K (en la orden, “G=1:20” )
Tenemos que aumentar. Lo hacemos:
wreathBIC<- mclustBIC(wreath,G=1:20,x=wreathBIC)
plot(wreathBIC,G=10:20,legendArgs=list(x="bottomright"))
vemos que a partir de 14 se estabiliza. No obstante, lo vemos con el summary para
saber que modelo es el mejor.
summary(wreathBIC,data=wreath)
EEV,14. Es decir, tenemos un total de 14 normales bivariantes y lass
matrices de dispersión ya no son totalmente iguales.
wreathBIC
El mejor modelo es EEV con 14 componentes, es decir, matriz de covarianzas:
λDkADTk
Igual volumen (λ), igual forma (A), distinta orientación (Dk)
Para que quede mas bonito:
plot(wreath, col=summary(wreathBIC,data=wreath)$classification )
También podría gustarte
- FN Sem11Documento12 páginasFN Sem11Melidy Eided Hinostroza MitaccAún no hay calificaciones
- Trabajo de Metodos NumericosDocumento38 páginasTrabajo de Metodos NumericosJulio Fernando Siguencia67% (6)
- Taller Algoritmos EnrutamientoDocumento3 páginasTaller Algoritmos EnrutamientoSANTIAGO SABOGAL CORREAAún no hay calificaciones
- Métodos Matriciales para ingenieros con MATLABDe EverandMétodos Matriciales para ingenieros con MATLABCalificación: 5 de 5 estrellas5/5 (1)
- ViabilidadDocumento26 páginasViabilidadjoseAún no hay calificaciones
- Ejercicio 1Documento10 páginasEjercicio 1Ismael BPAún no hay calificaciones
- Trabajo Definitivo Ramboarnoldshuarnegerrobocopjordy 1Documento20 páginasTrabajo Definitivo Ramboarnoldshuarnegerrobocopjordy 1Jordy Alexander Rodríguez RosalesAún no hay calificaciones
- Tarea 1 20231Documento3 páginasTarea 1 20231Ismael BPAún no hay calificaciones
- (P2-1) Estadistica DescriptivaDocumento24 páginas(P2-1) Estadistica DescriptivaDavid AyalaAún no hay calificaciones
- Arreglos y VectoresDocumento20 páginasArreglos y VectoresNew NuciAún no hay calificaciones
- Eval1 2022Documento4 páginasEval1 2022Andrés EspinozaAún no hay calificaciones
- PRUEBA SABER Enero-2018-1Documento11 páginasPRUEBA SABER Enero-2018-1Emiliano CordobaAún no hay calificaciones
- Estadística AplicadaDocumento7 páginasEstadística AplicadaLogAún no hay calificaciones
- Serie 1 Proba WendolinnneDocumento28 páginasSerie 1 Proba WendolinnneJohnJasenAún no hay calificaciones
- Matrices y Vectores-ProgramacionDocumento21 páginasMatrices y Vectores-ProgramacionCarito171210Aún no hay calificaciones
- Probabilidad 2016 Tema 3 FinalDocumento30 páginasProbabilidad 2016 Tema 3 FinalDaniel Montilla MorenoAún no hay calificaciones
- CD Tarea3 Antonio MarinoDocumento6 páginasCD Tarea3 Antonio MarinoGerman Galdamez OvandoAún no hay calificaciones
- Ejercicios Resueltos Estimación Inferencia EstadísticaDocumento9 páginasEjercicios Resueltos Estimación Inferencia EstadísticaMarcelohenAún no hay calificaciones
- Intervalos de ConfianzaDocumento63 páginasIntervalos de ConfianzaErikaAún no hay calificaciones
- Radicales Multiplicacion y Division PDFDocumento22 páginasRadicales Multiplicacion y Division PDFHector LimonAún no hay calificaciones
- KmeansDocumento9 páginasKmeansLuis Alfred LópezAún no hay calificaciones
- Ajuste de DistribucionesDocumento5 páginasAjuste de DistribucionesByron Alexis PalazziAún no hay calificaciones
- Cubo de RubikDocumento5 páginasCubo de Rubikmgl94100% (1)
- Muestreo e Intervalos de ConfianzaDocumento64 páginasMuestreo e Intervalos de ConfianzaRoberto JulcaAún no hay calificaciones
- Aplicacion Del Lenguaje R1Documento3 páginasAplicacion Del Lenguaje R1David ÁlvarezAún no hay calificaciones
- Ejercicio de TreeDecision - KMeansDocumento4 páginasEjercicio de TreeDecision - KMeansALEJANDROAún no hay calificaciones
- CluDocumento8 páginasCluEdwin Johny Asnate SalazarAún no hay calificaciones
- Ejercicios R PDFDocumento4 páginasEjercicios R PDFVicky Mota López0% (1)
- Variables AleatoriasDocumento51 páginasVariables AleatoriasJordan Kelvin CastilloAún no hay calificaciones
- DB3 - 2B Bryan MatailoDocumento6 páginasDB3 - 2B Bryan MatailoBryan MatailoAún no hay calificaciones
- NotasDocumento37 páginasNotasjulio hernandezAún no hay calificaciones
- ConjuntosDocumento29 páginasConjuntosNoelia OrozcoAún no hay calificaciones
- Latex Parte 2Documento45 páginasLatex Parte 2Gabriel Jose CarvajalAún no hay calificaciones
- Laboratorio 6 y 7 Modelos ProbabilisticosDocumento40 páginasLaboratorio 6 y 7 Modelos ProbabilisticosCristian CRAún no hay calificaciones
- 1 Tipos Numéricos en MatlabDocumento57 páginas1 Tipos Numéricos en MatlabEddy SanchezAún no hay calificaciones
- Practica IiiDocumento7 páginasPractica IiiInoel FernandezAún no hay calificaciones
- 4ta Práctica Calificada Parte 01Documento14 páginas4ta Práctica Calificada Parte 01Cristian OrbegosoAún no hay calificaciones
- Produccion Final MAT 329Documento11 páginasProduccion Final MAT 329Werlin AGAún no hay calificaciones
- Mi Acordeon 1Documento5 páginasMi Acordeon 1Mar YgAún no hay calificaciones
- Recursividad PDFDocumento34 páginasRecursividad PDFAlfredo ChoqueAún no hay calificaciones
- Xdoc - MX Practicas RecursividadDocumento10 páginasXdoc - MX Practicas RecursividadLioney Ortiz TorrezAún no hay calificaciones
- Teordivi PDFDocumento49 páginasTeordivi PDFAlejo Ortiz AlvarezAún no hay calificaciones
- Tema 4 - Teoria de Numeros y Aritmetica Modular - FVCDocumento19 páginasTema 4 - Teoria de Numeros y Aritmetica Modular - FVCJosé Moruno ArenaAún no hay calificaciones
- Calcule La Prueba de KruskalDocumento35 páginasCalcule La Prueba de KruskalJuan Carlos Arellan ManriqueAún no hay calificaciones
- 1.09 The Kernel TrickDocumento9 páginas1.09 The Kernel TrickMartín CoronelAún no hay calificaciones
- Consolidado Algebra LinealDocumento8 páginasConsolidado Algebra Linealjuliana bedoyaAún no hay calificaciones
- Análisis de Cluster, Un Ejemplo SencilloDocumento9 páginasAnálisis de Cluster, Un Ejemplo SencillojustorfcAún no hay calificaciones
- Algoritmo de La DivisiónDocumento42 páginasAlgoritmo de La DivisiónJesus C. Martinez100% (1)
- Artic 1 2Documento6 páginasArtic 1 2Evelyn de CristoAún no hay calificaciones
- Mínimo Comun MultiploDocumento2 páginasMínimo Comun MultiplolaaventuradelpoetaAún no hay calificaciones
- Transformaciones HomogéneasDocumento12 páginasTransformaciones HomogéneasRigoberto XVIIIAún no hay calificaciones
- Banco Ejercicios de RecursividadDocumento37 páginasBanco Ejercicios de RecursividadRoberto ValenzuelaAún no hay calificaciones
- Programa de Mejoramiento Guía 01 Conjuntos Numéricos MathtypeDocumento31 páginasPrograma de Mejoramiento Guía 01 Conjuntos Numéricos Mathtypecvcarlosvilla1Aún no hay calificaciones
- Explicación Paso A PasoDocumento16 páginasExplicación Paso A Pasokarlafilian_Aún no hay calificaciones
- MATLAB E J e R C I C I o MANDARDocumento13 páginasMATLAB E J e R C I C I o MANDARadrianaAún no hay calificaciones
- Maximo Comun Divisor TerminadoDocumento16 páginasMaximo Comun Divisor TerminadoVianney Clemente HoyosAún no hay calificaciones
- Mínimo Común Múltiplo: Múltiplo (Abreviado M.C.M) de Dos o MásDocumento22 páginasMínimo Común Múltiplo: Múltiplo (Abreviado M.C.M) de Dos o MásErvin Geovany Estuardo Macz cuAún no hay calificaciones
- AM 22 1 Pec02soDocumento6 páginasAM 22 1 Pec02sobadly83Aún no hay calificaciones
- GUÍA 4 (Cuarta Parte) Resolución de Problemas MatemáticosDocumento21 páginasGUÍA 4 (Cuarta Parte) Resolución de Problemas MatemáticosJuan Pablo Fonseca SanchezAún no hay calificaciones
- Introduction To Data Mining For Business Intelligence: Principal Component Analysis - PCA Cluster AnalysisDocumento45 páginasIntroduction To Data Mining For Business Intelligence: Principal Component Analysis - PCA Cluster AnalysisNacho PuertasAún no hay calificaciones
- Problem Set 3 Inferencia EstadisticaDocumento8 páginasProblem Set 3 Inferencia EstadisticafelipeAún no hay calificaciones
- Clustering en RDocumento13 páginasClustering en RrolyAún no hay calificaciones
- FleuryDocumento1 páginaFleuryChristian ZambranoAún no hay calificaciones
- Resumen Capitulo 2 David Poole 2edDocumento13 páginasResumen Capitulo 2 David Poole 2edKeops Ocampo OcampoAún no hay calificaciones
- 9º Taller 4 - Método de SustituciónDocumento2 páginas9º Taller 4 - Método de SustituciónNelly OrtegonAún no hay calificaciones
- 6 - Aplicacciones Con MathematicaDocumento13 páginas6 - Aplicacciones Con MathematicaChristianAún no hay calificaciones
- Introduccion A MatlabDocumento31 páginasIntroduccion A Matlabkenidy jeysonAún no hay calificaciones
- Algoritmica y ProgramacionDocumento3 páginasAlgoritmica y ProgramacionWanaadi Sotoi Tamatamañanocä Dhe'cwanacäAún no hay calificaciones
- Derivación Haca AtrasDocumento10 páginasDerivación Haca AtrasANDRES ESTEBAN NOBOA VILLACISAún no hay calificaciones
- ExactitudDocumento2 páginasExactitudalexmc000123Aún no hay calificaciones
- LagrangeDocumento5 páginasLagrangeSarc AsserAún no hay calificaciones
- Metodo de Iteracion Vectorial InversoDocumento8 páginasMetodo de Iteracion Vectorial InversoGus Jesús MartínezAún no hay calificaciones
- Trabajo Colaborativo Algebra Lineal PoliDocumento11 páginasTrabajo Colaborativo Algebra Lineal PoliDaniel Silva Ballen100% (4)
- Algoritmos de Funciones HashDocumento7 páginasAlgoritmos de Funciones HashvarceAún no hay calificaciones
- Busqueda BinariaDocumento19 páginasBusqueda BinariaJose Darwin Chacan TamameAún no hay calificaciones
- Aproximacion Newton RegresionesDocumento49 páginasAproximacion Newton RegresionesJHAMPIER ANCCASI HUAMANAún no hay calificaciones
- EntropiaDocumento5 páginasEntropiaHJayMbAún no hay calificaciones
- BNFDocumento7 páginasBNFRaul Baturoni ChavezAún no hay calificaciones
- Métodos Cuantitativos (Clase)Documento38 páginasMétodos Cuantitativos (Clase)Equipo 2Aún no hay calificaciones
- Informe - Taipe Rojas Luis EmilioDocumento3 páginasInforme - Taipe Rojas Luis EmiliojhancarlosAún no hay calificaciones
- Actividad 2 T4: Estadística Inferencial IDocumento7 páginasActividad 2 T4: Estadística Inferencial IEvelyn Diaz100% (1)
- Taller Empresa Estadistica Ii #3Documento4 páginasTaller Empresa Estadistica Ii #3Jose RomeroAún no hay calificaciones
- Distribución de BernoulliDocumento4 páginasDistribución de BernoulliYajaira ZambadaAún no hay calificaciones
- Cifrado de HillDocumento18 páginasCifrado de HillLeonardo Guzman OlayaAún no hay calificaciones
- SOADocumento8 páginasSOADavid GarcíaAún no hay calificaciones
- Investigación Operativa TiDocumento19 páginasInvestigación Operativa Tidennis.arturo.1212Aún no hay calificaciones
- Tema 3 - 2 - 12Documento21 páginasTema 3 - 2 - 121111Aún no hay calificaciones