Documentos de Académico
Documentos de Profesional
Documentos de Cultura
Tema8 Max Verosimilitud Selecc Modelos UANL Monterrey Jun08
Cargado por
Rufino ValladaresDerechos de autor
Formatos disponibles
Compartir este documento
Compartir o incrustar documentos
¿Le pareció útil este documento?
¿Este contenido es inapropiado?
Denunciar este documentoCopyright:
Formatos disponibles
Tema8 Max Verosimilitud Selecc Modelos UANL Monterrey Jun08
Cargado por
Rufino ValladaresCopyright:
Formatos disponibles
Tema 8: mxima verosimilitud, estima de parmetros y seleccin de modelos
Introduccin a la Inferencia Filogentica y Evolucin Molecular
23-26 Junio 2008, Fac. C. Biolgicas - UANL Pablo Vinuesa (vinuesa@ccg.unam.mx) Centro de Ciencias Genmicas-UNAM, Mxico http://www.ccg.unam.mx/~vinuesa/
Todo el material del curso lo puedes descargar desde: http://www.ccg.unam.mx/~vinuesa/UANL08
Intro. Biol. Filogentica - Lic. Biotecnologa Genmica, Univ. Autnoma de Nuevo Len, Monterrey
Mtodos de reconstruccin filogentica Mxima Verosimilitud
Mxima verosimilitud: dadas dos topologas, la que hace los datos observados ms probables (menos sorprendentes) es la preferida
El mtodo de mxima verosimilitud (ML) considera cada sitio variable del alineamiento (includos singletones). Bajo el criterio de ML se busca la topologa que hace ms verosmil el patrn de sustituciones de un alineamiento dado un modelo evolutivo explcito! As, para un set de datos D y una hiptesis evolutiva (topologa) H, la verosimilitud de dichos datos viene dado por la expresin:
LD=Pr(D|H)
que es la probabilidad de obtener D dada H (una probabilidad condicional) !
Por tanto la topologa que hace nuestros datos el resultado evolutivo ms probable corresponde a la estima de mxima verosimilitud de la filogenia (likelihood score valor de verosimilitud).
Tema 8: Criterios de optimizacin II: mxima verosimilitud
1. 2. 3. 4. 5. El criterio de mxima verosimilitud y su aplicacin en filogentica Estima de mxima verosimilitud de parmetros del modelo de sustitucin Seleccin de modelos de sustitucin usando pruebas de razones de verosimilitud (LRTs) Modelos paramtricos de evolucin de secuencias de DNA la familia GTR Prcticas con PAUP*, ModelTest y proml
la probabilidad est relacionada con la sorpresividad de los datos Estaramos sorprendidos de obtener este resultado, dada su bajsima probabilidad (1/6)20 1 en 3,656,158, 440,062,976! Pero la probabilidad depende del modelo probabilstico asumido En filogentica, las distintas topologas representan a los distintos modelos, y se selecciona aquel modelo que nos hace sorprendernos menos de los datos que hemos coleccionado
Mxima verosimilitud y estima de parmetros de modelos de sustitucin
La inferencia filogentica bajo el criterio de mxima verosimilitud se basa en el uso de una cantidad llamada log-likelihood para evaluar topologas alternativas con el fin de encontrar aquella que maximiza este valor. El log-likelihood es el ln de la verosimilitud, que es igual a la probabilidad de los datos observados dadas una topologa particular ( ), set de longitudes de rama ( ) y modelo de sustitucin ( ). Ntese que la verosimilitud no representa la probabilidad de que un rbol sea correcto; sta viene determinada por la probabilidad posterior de la estadstica bayesiana. Hablar de la verosimilitud de un conjunto de datos no es correcto ya que la verosimilitud es un funcin de los parmetros de un modelo estadstico, y no de los datos (D). Los datos son constantes siendo el modelo lo que es variable al calcular verosimilitudes. Se puede por lo tanto hablar de verosimilitudes como funciones de modelos o hiptesis (H ). La verosimilitud de una hiptesis dado un set de datos es igual a la probabilidad condicional de los datos dada una hiptesis. Formalmente: L (H |D) = Pr(D |H ) = Pr(D | )
Mxima verosimilitud y estima de parmetros de modelos de sustitucin
L (H |D) = Pr(D |H ) = Pr(D| )
Lo mejor es pensar en los rboles como modelos. La verosimilitud de una topologa particular ( ) ser la probabilidad de los datos dada esa topologa. Cada topologa tiene como parmetros las longitudes de rama ( ), y la verosimilitud de un modelo ( ) cambia segn varen los valores de los parmetros de longitud de rama Por lo tanto se puede concebir la filogentica bajo el criterio de mxima verosimilitud como un problema de seleccin de modelos. Se trata de encontar las estimas de los valores de cada parmetro del modelo y luego comparar las verosimilitudes de los distintos modelos, escogiendo el mejor (topologa) en base a su verosimilitud La topologa que hace de nuestros datos el resultado evolutivo ms probable (dado un modelo de sust.) es la estima de mxima verosimilitud de nuestra filogenia. Por tanto, al contrario que bajo los criterios de optimizacin de MP, LS o ME, bajo ML se trata de seleccionar modelos y parmetros que maximicen la funcin de optimizacin.
Pablo Vinuesa 2008, vinuesa@ccg.unam.mx http://www.ccg.unam.mx/~vinuesa
Tema 8: mxima verosimilitud, estima de parmetros y seleccin de modelos
Mxima verosimilitud y estima de parmetros de modelos de sustitucin Clculo del valor de mxima verosimilitud para una sola secuencia o rbol trivial con un solo nodo
primeros 25 nt del gen ropB de Bradyrhizobium japonicum USDA110
Intro. Biol. Filogentica - Lic. Biotecnologa Genmica, Univ. Autnoma de Nuevo Len, Monterrey
Mxima verosimilitud y estima de parmetros de modelos de sustitucin Clculo del valor de mxima verosimilitud para una sola secuencia o rbol trivial con un solo nodo
primeros 25 nt del gen ropB de Bradyrhizobium japonicum USDA110
ATGGCGCAGCAGACATTCACCGGTC
L = A T G G C G C A G C A G A C A T T C A C C G G T C
= AnA CnC GnG TnT = A6 C8 G7 T4 ln L = 6 ln (A) + 8 ln (C) + 7 ln (G) + 4 ln (T)
A = 0.24 C = 0.32 G = 0.28 T = 0.16
ATGGCGCAGCAGACATTCACCGGTC
- Clculo de lnL bajo el modelo de JC69
ln L = 6 ln (A) + 8 ln (C) + 7 ln (G) + 4 ln (T) = 6 ln (0.25) + 8 ln (0.25) + 7 ln (0.25) + 4 ln (0.25) = -29.1
- Clculo de lnL bajo el modelo de F81
ln L = 6 ln (A) + 8 ln (C) + 7 ln (G) + 4 ln (T) = 6 ln (0.24) + 8 ln (0.32) + 7 ln (0.28) + 4 ln (0.16) = -26.6
A primera vista podemos sospechar que el modelo de F81 se va a ajustar mejor a los datos que el de JC69, ya que las frecuencias de nucletidos difieren claramente de 0.25, con exceso de Cs y defecto de Ts
A = 0.24 C = 0.32 G = 0.28 T = 0.16
-Por lo tanto el modelo de F81 se ajusta mejor a los datos (-26.6 > -29.1). Esta diferencia ser tanto ms notoria cuanto ms larga sea la secuencia.
Mxima verosimilitud y estima de parmetros de modelos de sustitucin
Verosimilitud del rbol ms sencillo (dos nodos y una rama) bajo el modelo de JC69 1 2 La long. de la rama equivale a la dist. evolutiva entre las hojas o nodos terminales Probabilidades de transicin JC: 1.- prob. de no cambio, de i a i 2.- prob. de cambio, de j <-> i
Mxima verosimilitud y estima de parmetros de modelos de sustitucin
Estima del parmetro compuesto t del modelo JC69 para los primeros 30 nts de la globina de gorila y orangutan
Pii (t ) = Pr (i en sec. 1|i en sec. 2) = (1 + 3e-4t ) Pij (t ) = Pr (j en sec. 1|i en sec. 2) = (1 - e-4t )
GA GG
L =
L1
L2
Clculo de la verosimilitud por sitio (site likelihood) : para cada sitio Lk hay que calcular: prob. incondic. x prob. condicional
= [Pr (G ) Pr(G->G)] [Pr (A ) Pr(A->G)] = [][ (1 + 3e-4t )] [] [( -e-4t ) ] = [1/16 (1 + 3e-4t )] [1/16 (1 - e-4t )] * * * GAATCCGA
Cmo estimamos el valor de t ? La estima de mxima verosimilitud se obtiene del anlisis de la funcin de verosimilitud, esencialmente probando diversos valores para el parmetro y determinando cual maximiza la funcin
* * * GGATGCGT
L = L1 L2 ... L8 = [1/16 (1 + 3e-4t )]5 [1/16 (1 - e-4t )]3
ln L = 5 ln [1/16 (1 + 3e-4t )] + 3 ln [1/16 (1 - e-4t )]
Clculo de la verosimilitud global para un rbol con 2 nodos terminales y n nucletidos alineados:
dJC69 = 3t
= 3 (0.0237) = 0.0474
L = Lk
Pablo Vinuesa 2008, vinuesa@ccg.unam.mx http://www.ccg.unam.mx/~vinuesa
Tema 8: mxima verosimilitud, estima de parmetros y seleccin de modelos
Mxima verosimilitud y estima de parmetros de modelos de sustitucin
Esquema del procedimiento del clulo del valor de verosimilitud de un rbol con 4 OTUs * * * GAATCCGA GAATCCGA GGATGCGT * * * GGATGCGT En un rbol con slo 2 OTUs no tenemos ningn nodo interior o ancestral. El cmputo lo realizamos directamente sobre los datos observados
Intro. Biol. Filogentica - Lic. Biotecnologa Genmica, Univ. Autnoma de Nuevo Len, Monterrey
Mxima verosimilitud y estima de parmetros de modelos de sustitucin
Esquema del procedimiento del clculo del valor de verosimilitud de un rbol con 4 OTUs
L = L1 L2 ... L8 =
Para 4 OTUs existen 3 topologas posibles. Por ello hemos de repetir este clculo para cada una de ellas con el fin de encontrar la topol. ms verosmil La verosimilitud para cada sito representa la suma sobre todas las posibles asignaciones de estados de caracter en todas las ramas interiores de un rbol. La verosimilitud total es el producto de las veros. por sitio.
= [1/16 (1 + 3e-4t )]5 [1/16 (1 - e-4t )]3
La complicacin adicional que encontramos para el clculo de verosimilitudes de rboles con > 3 OTUs radica esencialmente en que tenemos ahora nodos interiores para los que carecemos de observaciones. Se trata de unidades taxonmicas hipotticas HTUs. En este caso, para calcular la verosimilitud del rbol tenemos que considerar cada posible estado de caracter para cada nodo interior y para cada topologa !!!.
Mxima verosimilitud y estima de parmetros de modelos de sustitucin
La inferencia filogentica bajo el criterio de mxima verosimilitud implica MUCHISIMO TRABAJO COMPUTACIONAL (=> mucho tiempo de trabajo de procesador) Las verosimilitudes globales han de ser maximizadas para cada topol. Para ello necesitamos: - encontrar EMV para cada long. de rama y cada parmetro del modelo de sust. - ello implica calcular la verosimilitud global muchas, pero que muchas veces En la prctica los rboles de ML se estiman en mltiples ciclos, en los que se van optimizando secuencialmente los diversos parmetros del modelo de sustitucin y longitudes de rama. La estima conjunta de todos los parmetros se hace computacionalmente prohibitiva Por lo general se comienzan estos ciclos partiendo de una topologa obtenida por un mtodo rpido, tal como NJ o MP. Sobre esta topologa se ajustan los valores de los parmetros del modelo. A continuacin se emplea algn mtodo de reajuste de topologa (branch swapping) y se ajustan las longitudes de rama, cerrando un ciclo. En mltiples ciclos consecutivos se va optimizando la topologa y long. de rama, hasta que convergen en la estima de mxima verosimilitud global
Mxima verosimilitud y estima de parmetros de modelos de sustitucin
1. La relevancia e impacto de los modelos de evolucin en filogentica y evol. molecular - Los modelos no son slo importantes por sus consecuencias en la estima filogentica, sino que adems lo son porque la caracterizacin del proceso evolutivo a nivel molecular es objeto de estudio en s mismo, en el mbito de la evolucin molecular. - Los modelos de evolucin son herramientas poderosas siempre y cuando, a pesar de las simplificaciones que hacen, describan adecuadamente las caractersticas ms salientes de los datos y permitan hacer predicciones precisas sobre el problema bajo estudio. - El rendimiento de un mtodo se maximiza cuando se satisfacen los supuestos subyacentes. - Es conveniente por tanto seleccionar el modelo ms adecuado para cada set particular de datos y cuantificar el ajuste de los datos al modelo seleccionado.
Pablo Vinuesa 2008, vinuesa@ccg.unam.mx http://www.ccg.unam.mx/~vinuesa
Tema 8: mxima verosimilitud, estima de parmetros y seleccin de modelos
Mxima verosimilitud y estima de parmetros de modelos de sustitucin
2. Seleccin de modelos de sustitucin de secuencias de DNA - En trminos generales modelos complejos se ajustan a los datos mejor que los simples. Idealmente se ha de seleccionar un modelo los suficientemente complejo (rico en parmetros) como para describir adecuadamente las caractersticas ms notables del patrn de sust. del set de datos, pero no sobreparametrizado para evitar colineariedad de parmetros (redundancia), tiempos excesivamente largos de cmputo y estimas poco precisas de los parmetros por excesiva varianza.
Intro. Biol. Filogentica - Lic. Biotecnologa Genmica, Univ. Autnoma de Nuevo Len, Monterrey
Mxima verosimilitud y estima de parmetros de modelos de sustitucin
2. Seleccin de modelos de sustitucin de secuencias de DNA y Protena - Se deben de usar pruebas estadsticas para seleccionar el modelo que mejor se ajusta a los datos de entre los disponibles. Este ajuste de los modelos a los datos puede ser evaluado usando pruebas de razones de verosimilitud (likelihood ratio tests, LRTs) o usando criterios de informacin de Akaike o bayesianao (AIC y BIC, respectivamente). Se puede usar una prueba de LRT para evaluar la capacidad que tiene un modelo particular en ajustar los datos.
ajuste a los datos observados producidos por una funcin polinomial vs. una func. lineal
aadir parmetros a un modelo generalmente mejora su ajuste a los datos observados modelos infra-parametrizados conducen a un pobre ajuste a los datos observados modelos supra-parametrizados conducen a una pobre prediccin de eventos futuros existen mtodos estadsticos para seleccionar modelos ajustados a cada set de datos
- Idealmente debemos de seleccionar el mejor modelo para cada gen o regin genmica que queramos analizar. No conviene hacerlo para una supermatriz de alineamientos concatenados. El uso de modelos particionados en los que se ajusta el modelo para cada posicin de los codones, por cada gen a analizar, resultan generalmente en ajustes globales significativamente mejores que modelos promediados para cada gen.
Mxima verosimilitud y estima de parmetros de modelos de sustitucin
3. Prueba de razn de verosimilitudes (LRT) Una manera natural y muy usada de comparar el ajuste relativo de dos modelos alternativos a una matriz de datos es contrastar las verosimilitudes resultantes mediante la prueba de razones de verosimilitud (RV) likelihood ratio test (LRT):
Mxima verosimilitud y estima de parmetros de modelos de sustitucin
3. Prueba de razn de verosimilitudes (LRT) - El LRT es por tanto una prueba estadstica para cuantificar la bondad relativa de ajuste entre dos modelos anidados. Veamos un ejemplo. Vamos seleccionar entre los modelos JC69, F81, HKY85 y TrN93 para el set de datos de mtDNA-primates.nex, considerando
= 2(loge L1 - loge L0)
donde L1 es el valor de ML global para la hiptesis alternativa (modelo ms rico en parmetros) y L0 es el valor de ML global para la hiptesis nula (el modelo ms simple). >= 0 siempre, ya que los parmetros adicionales van a dar una mejor explicacin de la variacin estocstica en los datos que el modelo ms sencillo. Cuando los modelos a comparar estn anidados (L0 es un caso especial de L1) el estadstico sigue aproximadamente una distribucin
2
slo las regiones codificadoras y eliminando Lemur_catta, Tarsius_syrichta y Saimiri_scireus y usando un rbol NJ sobre el cual estimar parmetros
Modelo JC69 F81 HKY85 TrN93
-lnL 3585.54820 3508.04085 3233.34395 3232.29439
Qu podemos conclur de estos valores de lnL en cuanto a la importancia relativa de los parmetros considerados por estos modelos en cuanto al nivel de ajuste a los datos que alcanzan ?
con q grados de libertad, donde
q = diferencia entre el no. de parmetros libres entre L1 y L0 .
Pablo Vinuesa 2008, vinuesa@ccg.unam.mx http://www.ccg.unam.mx/~vinuesa
Tema 8: mxima verosimilitud, estima de parmetros y seleccin de modelos
Mxima verosimilitud y estima de parmetros de modelos de sustitucin
3. Prueba de razn de verosimilitudes (LRT)
Intro. Biol. Filogentica - Lic. Biotecnologa Genmica, Univ. Autnoma de Nuevo Len, Monterrey
Mxima verosimilitud y estima de parmetros de modelos de sustitucin
3. Prueba de razn de verosimilitudes (LRT)
Modelo JC69 F81 HKY85 TrN93 modelos JC-F81 JC-HKY85 JC-TrN F81-HKY85 F81-TrN KHY-TrN
-lnL 3585.54820 3508.04085 3233.34395 3232.29439 diff. GL = q 3-0=3 4-0=4 50=5 43=1 53=2 54 =1
H0 a rechazar (o hiptesis anidadas a evaluar)
1. igual frec. de bases 2. Ti = Tv 3. tasas de Ti iguales Modelo HKY85 HKY85 +G HKY85 +I+G -lnL 3233.34395 3145.29031 3142.36439
H0 a rechazar (o hiptesis anidadas a evaluar)
1. tasa homognea de sust entre sitios 2. no existe proporcin de sitios invariantes
...
X2
P
0 0 0 0 0 0.15 modelos HKY85-vs. +G HKY85+G vs. I+G diff. GL = q 1 1
155 704.4 706.4 549.4 551.4 2.1
X2
176 5.85
P
0 0.015
Por lo tanto el modelo seleccionado es el HKY
Por lo tanto el modelo seleccionado es el HKY+G si tomamos 0.01 como punto de corte, o HKY+I+G si usamos alfa = 0.05.
http://www.fourmilab.ch/rpkp/experiments/analysis/chiCalc.html
Mxima verosimilitud y estima de parmetros de modelos de sustitucin
3. Esquema jerrquico de efectuar LRTs partiendo desde el modelo ms sencillo (JC69)
Mxima verosimilitud y estima de parmetros de modelos de sustitucin
3. Resumen de algunos modelos y sus parmetros libres - Dado que en los modelos de sust. de DNA la tasa promedio de sustitucin se considera = 1
H0
A = acepto R = rechazo
y los parmetros de tasa relativa se escalan de tal manera que la tasa promedio de sust. en equilibrio = 1, el modelo ms sencillo (JC69) no tiene ningn parmetro libre, dado que el nico parmetro () a estimar valdr en este contexto. Modelo JC F81 K2P HKY85 TrN93 GTR caractersticas nst= 1 basefreq= equal nst=1 basefreq=uneq nst=2 basefreq=eq nst=2 basefreq=uneq nst=3 basefreq=uneq nst=6 basefreq=uneq no. de parmetros libres 0 3 para las frec. de bases 1 para el tratio (ti/tv) 4 (1 para tratio y 3 para frec. de bases) 5 (2 tasas de ti y 3 para freq de bases) 8 (5 para tasas de subst y 3 para freq. de bases) 1 parmetro libre adicional para pinv 1 parmetro libre adicional para G 2 parmetros libres adicionales
HKY+I+G
proporcin de sitios invariantes (I) distribucin gamma (G) ambos combinados (I+G)
Pablo Vinuesa 2008, vinuesa@ccg.unam.mx http://www.ccg.unam.mx/~vinuesa
Tema 8: mxima verosimilitud, estima de parmetros y seleccin de modelos
Intro. Biol. Filogentica - Lic. Biotecnologa Genmica, Univ. Autnoma de Nuevo Len, Monterrey
Mxima verosimilitud y estima de parmetros de modelos de sustitucin
3. Seleccin de modelos usando criterios de informacin
LRT compara pares de modelos anidados. Los criterios de informacin como el Akaike information criterion (AIC) y Bayesian information criterion (BIC) comparan simultneamente todos los modelos en competicin y permiten seleccionar modelos aunque no sean anidados.
Se trata nuevamente de incorporar tanta complejidad (parmetros) al modelo como requieran los datos. La verosimilitud para cada modelo es penalizada en funcin del nmero de parmetros: a mayor cantidad de parmetros mayor penalizacin.
Mxima verosimilitud y estima de parmetros de modelos de sustitucin
3. Seleccin de modelos usando criterios de informacin
Mxima verosimilitud y estima de parmetros de modelos de sustitucin
3. Seleccin de modelos usando criterios de informacin: AIC Se pueden usar los estadsticos de diferencias en AIC (i) y ponderaciones de Akaike
AIC. Es un estimador no sesgado del parmetro de contenido de informacin de Kullback-Leibler, el cual es una medida de la informacin perdida al usar un modelo para aproximar la realidad. Por tanto, a menor valor de AIC mejor ajuste del modelo a los datos. Al penalizar por cada parmetro adicional, considera tanto la bondad de ajuste como la varianza asociada a la estima de los parmetros.
para cuantificar el nivel de incertidumbre en la seleccin del modelo. Las i son AICs re-escalados con respecto modelo con el AIC ms bajo (minAIC), de modo que i = AICi minAIC. Las i son fciles de interpretar y permiten ordenar los los modelos cadidatos. As, modelos con i en un rango de 1-2 con respecto al modelo ganador tienen un soporte sustancial y
AICi = -2ln Li + 2 Ni
Ni = no. de parmetros libres en el modelo i Li = verosimilitud bajo el modelo i
deben de ser considerados como modelos alternativos. Modelos con i en un rango de 3-7 con respecto al modelo ganador tienen un soporte significativamente inferior, y modelos con i > 10 carecen de soporte.
Pablo Vinuesa 2008, vinuesa@ccg.unam.mx http://www.ccg.unam.mx/~vinuesa
Tema 8: mxima verosimilitud, estima de parmetros y seleccin de modelos
Mxima verosimilitud y estima de parmetros de modelos de sustitucin
3. Seleccin de modelos usando criterios de informacin: AIC Las ponderaciones o pesos de Akaike (wi) son los AIC relativos normalizados para cada modelo en competicin y pueden ser interpretados como la probabilidad de que un modelo es la mejor abstraccin de la realidad dados los datos. Para R modelos candidatos a evaluar:
Intro. Biol. Filogentica - Lic. Biotecnologa Genmica, Univ. Autnoma de Nuevo Len, Monterrey
Criterios de optimizacin: la alteranativa Bayesiana
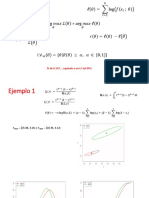
Aproximaciones tradicionales (matrices de distancia, ME, ML, MP) - la bsqueda tiene por objetivo encontrar la topologa ptima (estima puntual) - no pueden establecer el soporte relativo de las biparticiones a partir de una nica bsqueda MLE
LD
LH= Pr(D|H) = Pr(D|)
tree space (i )
Aproximacin Bayesiana
Una aplicacin muy til de los wi es que la inferencia se puede promediar a partir de los modelos que muestran valores de no wi triviales. As, una estima del valor del parmetro a de la distribucin gamma promediada a partir de varios modelos se calculara as: Tambin podemos reconstruir filogenias bajo los distintos modelos con peso significativo y combinar los rboles resultantes acorde a sus pesos de Akaike. Esta estrategia es particularmente til en un contexto bayesiano.
- no busca una solo topologa ptima sino una poblacin de rboles muestreados en funcin de su probabilidad posterior (algoritmos MCMC) - la muestra de rboles obtenidos en una sola sesin de bsqueda es usada para valorar el soporte de cada split en trminos de propabilidad posterior
pP
95% credibility interval
tree space (i )
Pablo Vinuesa 2008, vinuesa@ccg.unam.mx http://www.ccg.unam.mx/~vinuesa
También podría gustarte
- Tratamiento y Propagación de ErroresDocumento3 páginasTratamiento y Propagación de ErroresRufino ValladaresAún no hay calificaciones
- Nociones de TopografiaDocumento83 páginasNociones de TopografiaJorge Manuel Reyes SerranoAún no hay calificaciones
- Topografia Elemental Pub408Documento75 páginasTopografia Elemental Pub408habp610% (1)
- Manual OriginDocumento19 páginasManual OriginRufino Valladares0% (1)
- Solucionario AlgebraDocumento413 páginasSolucionario AlgebraRufino Valladares0% (1)
- Artículo Docente. Determinación de La Densidad de Un Líquido Con El Método Del PicnómetroDocumento5 páginasArtículo Docente. Determinación de La Densidad de Un Líquido Con El Método Del PicnómetroBenigno CruzAún no hay calificaciones
- Extracción de La Caseina y Determinación Del Punto IsoelectricoDocumento8 páginasExtracción de La Caseina y Determinación Del Punto IsoelectricoRufino Valladares100% (1)
- PUNTO DE CONGELACIÓN DE MEZCLAS DE NAFTALENO Y p-DICLOROBENCENODocumento3 páginasPUNTO DE CONGELACIÓN DE MEZCLAS DE NAFTALENO Y p-DICLOROBENCENORufino ValladaresAún no hay calificaciones
- Solucionario AlgebraDocumento413 páginasSolucionario AlgebraRufino Valladares0% (1)
- Viscosidad de GasesDocumento3 páginasViscosidad de GasesRufino ValladaresAún no hay calificaciones
- Medidas de La Viscosidad y La Densidad de Dos LiquidosDocumento10 páginasMedidas de La Viscosidad y La Densidad de Dos LiquidosRufino ValladaresAún no hay calificaciones
- Punto de FusionDocumento1 páginaPunto de FusionRufino ValladaresAún no hay calificaciones
- Manual-Estilo ECMFIL20110516 0001Documento87 páginasManual-Estilo ECMFIL20110516 0001mauriciovillotaAún no hay calificaciones
- Manual AwkDocumento119 páginasManual AwkFrancisco Javier Ramirez JaramilloAún no hay calificaciones
- Manual OriginDocumento19 páginasManual OriginRufino Valladares0% (1)
- Colombia, Cuentos Cortos - Eduardo Varela CidDocumento51 páginasColombia, Cuentos Cortos - Eduardo Varela Cidapi-3741297Aún no hay calificaciones
- Gerencia Decisiones Examen FinalDocumento8 páginasGerencia Decisiones Examen FinalClaudia MuñozAún no hay calificaciones
- Ejercicios Simulacion Gerencial ParcialDocumento135 páginasEjercicios Simulacion Gerencial ParcialJohanna Torres OrtizAún no hay calificaciones
- Ejercicio Distribucion Muestrales de ProporcionesDocumento2 páginasEjercicio Distribucion Muestrales de ProporcionesYesica VargasAún no hay calificaciones
- PDF Semana 8 Estadisticas Iacc DDDocumento4 páginasPDF Semana 8 Estadisticas Iacc DDSergio Vera IribarrenAún no hay calificaciones
- Practica 2 - Estimacion de ParametrosDocumento4 páginasPractica 2 - Estimacion de ParametrosJeiel Alarcon GerlAún no hay calificaciones
- Actividad 4 Distribuciones Binomial Poisson y NormalDocumento16 páginasActividad 4 Distribuciones Binomial Poisson y Normalelizabeth100% (1)
- 4.3.MEDIDAS ESTADISTICAS Parte 4Documento20 páginas4.3.MEDIDAS ESTADISTICAS Parte 4Syu TlvAún no hay calificaciones
- Clase 17 Proceso PoissonDocumento24 páginasClase 17 Proceso PoissonWilmer SuarezAún no hay calificaciones
- Informe WeibullDocumento12 páginasInforme WeibullJonatan Y. Grijalva100% (3)
- 21 27Documento2 páginas21 27Jonatan SanchezAún no hay calificaciones
- Taller Tercer Corte Teoria de ColasDocumento9 páginasTaller Tercer Corte Teoria de ColasJose Rafael Ramos ChiquilloAún no hay calificaciones
- Examen Unidad II - Estadistica Inferencial 2022Documento7 páginasExamen Unidad II - Estadistica Inferencial 2022Leidy Paola Peralta AvilaAún no hay calificaciones
- Introduccion A Los GLMDocumento46 páginasIntroduccion A Los GLMjosemuiangaAún no hay calificaciones
- Verosimilitud RelativaDocumento7 páginasVerosimilitud RelativaDavid GuamánAún no hay calificaciones
- Uni 4 EstadisticaDocumento34 páginasUni 4 EstadisticaDiianaa Paalaaciios100% (1)
- Medidas de dispersión en bioestadísticaDocumento3 páginasMedidas de dispersión en bioestadísticaMarco SossaAún no hay calificaciones
- Guía de Trabajo Nº1 Distribucion NormalDocumento2 páginasGuía de Trabajo Nº1 Distribucion NormalCatalina Gangas Moreno0% (1)
- Ejercicios AnovaDocumento3 páginasEjercicios AnovaDiego RodriguezAún no hay calificaciones
- Examen Parcial - Semana 4 - CB - Segundo Bloque-Estadistica II - (Grupo6) YulyDocumento8 páginasExamen Parcial - Semana 4 - CB - Segundo Bloque-Estadistica II - (Grupo6) YulyyulymaryAún no hay calificaciones
- Analisis Factorial Con Teoria y SpssDocumento56 páginasAnalisis Factorial Con Teoria y SpssAbraham RodriguezAún no hay calificaciones
- Pruebas de hipótesis e intervalos de confianzaDocumento5 páginasPruebas de hipótesis e intervalos de confianzaFelipe KempAún no hay calificaciones
- Ejercicio de Probabilidad y Variables Discretas para DistribucionesDocumento2 páginasEjercicio de Probabilidad y Variables Discretas para DistribucionesMichael sepulvedaAún no hay calificaciones
- Tarea Semana 5 Estadistica para La Gestion IACCDocumento5 páginasTarea Semana 5 Estadistica para La Gestion IACCDannae CamposAún no hay calificaciones
- Proyecto Poisson PDFDocumento14 páginasProyecto Poisson PDFtanialcivarAún no hay calificaciones
- Mpes U3 A2 LapbDocumento13 páginasMpes U3 A2 LapbLaura Pontón Becerril100% (1)
- Distribución de CauchyDocumento4 páginasDistribución de CauchyHTB videos youtubeAún no hay calificaciones
- Prueba de Hipótesis de Una Muestra Única - YFDocumento3 páginasPrueba de Hipótesis de Una Muestra Única - YFYobana FIERRO ALVAREZAún no hay calificaciones
- 1er Parcial 1ra Parte Multiple ChoiceDocumento3 páginas1er Parcial 1ra Parte Multiple ChoicemainCTUAún no hay calificaciones
- Funcion Generadora de MomentosDocumento16 páginasFuncion Generadora de MomentosEvelin PinargoteAún no hay calificaciones
- Estadistica Presentación, Grupo 1Documento7 páginasEstadistica Presentación, Grupo 1romanAún no hay calificaciones